추천글 : 【분자생물학】 변리사 2차 분자생물학 기출문제 풀이
a. 중심학설
b. DNA 테크놀로지
1 님 지적사항 수정 (23.04.23)
문제 1. 포유동물 A의 수컷 체세포는 18,000,000 염기쌍(base pair, bp) 크기의 X 염색체를 갖고 있다. 이 동물 A의 체세포는 11시간의 G1기, 10시간의 S기, 2시간의 G2기, 1시간의 M기의 세포주기(cell cycle)를 거쳐 증식하며, DNA 중합효소의 복제속도는 분당 3,000 bp이다. 다음 물음에 답하시오. (단, 이 X 염색체의 18,000,000 bp에는 TTAGGG 말단소체(telomere) 서열을 포함하지 않으며, 모든 복제개시점(origin of replication)으로부터 양방향(bidirectional)으로, 모든 오카자키 절편(Okazaki fragment)은 동일하게 150 bp 크기로, 복제가 일어난다고 가정한다.)
⑴ 1-1. 이론적으로 이 X 염색체의 복제에 필요한 복제개시점의 최소 개수를 구하시오. (단, 답을 구하는 과정을 설명하시오.)

Figure. 1. 1-1 문제상황
○ 복제원점이 n개 있으면 DNA는 n+1개로 나뉘어진다. 지연가닥과 선도가닥을 중합하는 속도가 같다고 가정하자. Figure. 1에서 위쪽 주형의 5' 말단 또는 3' 말단에 대응되는 딸가닥(A; 선도가닥)의 중합속도는 3,000 bp / min이고 가운데 지연가닥과 선도가닥이 동시에 중합되는 부분(B; 위쪽 주형에 대응되는 딸가닥에서 A를 제외한 나머지)은 6,000 bp / min이라고 할 수 있다. 이는 아래쪽 주형도 동일하다. 따라서 A의 뉴클레오타이드 수를 nt(A), B의 뉴클레오타이드 수를 nt(B)라고 하면 주어진 복제속도를 달성하기 위한 복제개시점의 개수를 최소로 하려면 nt(A) / 3000 = nt(B) / 6000임을 안다. 즉,
1. max { nt(A) / 3000, nt(B) / 6000 } ≤ 600
2. 2 × nt(A) + (n-1) × nt(B) ≒ 18,000,000
3. 2 × nt(A) = nt(B)
∴ nt(B) = 18,000,000 / n
∴ 600 × 6,000 = 18,000,000 / n → n = 5 → n ≥ 5
⑵ 1-2. 이 동물 체세포에서 X 염색체가 복제되는 동안 생성되는 오카자키 절편의 개수를 구하고, 이 X 염색체의 복제에 필요한 RNA 프라이머의 최소 개수를 구하시오. (단, 답을 구하는 과정을 설명하시오.)
○ 기존 풀이 : 오카자키 절편의 크기는 동일하게 150 bp이다. 두 딸가닥의 오카자키 절편들 모두를 합치면 대략 원래 주형가닥 하나 정도가 되므로 X 염색체가 복제되는 동안 생성되는 오카자키 절편의 개수는 18,000,000 / 150 = 120,000개이다. 따라서 RNA 프라이머 역시 120,000개가 필요하다.
○ 개선된 풀이 : 다만, 위 풀이는 복제 버블 당 RNA 프라이머가 2개씩 더 발생한다는 사실을 간과한 것이다. Figure. 1에서 RNA 프라이머가 18개가 아니라 18+6 = 24개가 필요한 것과 같은 이유이다. 따라서 RNA 프라이머는 120,000 + 2 × 6 = 120,012개가 필요하다.
문제 2. 포유류에서 유전체 각인(genomic imprinting)은 부모에게서 받은 두 개 대립유전자 중 부계 또는 모계 유래 한 개 대립유전자만을 발현하여 나타나는 현상이다. 다음 물음에 답하시오.
⑴ 2-1. 다음 그림은 유전체 각인으로 잘 알려져 있는 Igf2와 H19 유전자가 위치하는 염색체 일부이다. 조절서열 Ⅱ는 모계 유래 염색체에서는 H19 유전자에만 작용하고 부계 유래 염색체에서는 Igf2 유전자에만 작용하여 이들의 발현을 활성화시킨다. 조절서열 Ⅰ이 결손된 모계 염색체와 부계 염색체에서는 두 유전자 모두 발현이 활성화된다. Igf2와 H19 유전자들의 발현 조절에서의 조절서열 Ⅰ과 Ⅱ의 역할을 설명하고, 조절서열 Ⅰ이 조절서열 Ⅱ를 어떻게 조절하는지 설명하시오.
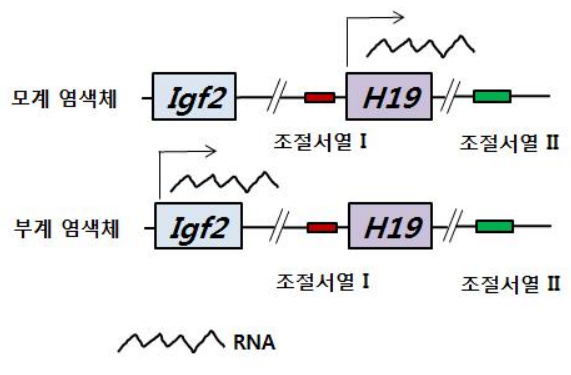
Figure. 2. 2-1 문제상황
○ 조절서열 Ⅱ는 유전자의 발현을 활성화시키므로 특정 유전자에 대한 인핸서(enhancer)로 작용한다. 또한, 조절서열 Ⅰ이 결손된 모계 염색체와 부계 염색체에서 두 유전자 모두 발현이 활성화되므로 조절서열 Ⅰ은 두 유전자 모두에 대한 사일렌서(silencer)로 작용한다고 보는 게 타당하다. 또한, 어떤 유전자에 대하여 조절서열 Ⅰ과 조절서열 Ⅱ가 경합하는 경우 조절서열 Ⅱ에 의해 유전자의 발현이 활성화되므로 조절서열 Ⅱ는 조절서열 Ⅰ을 억제한다.
⑵ 2-2. H19 유전자의 산물은 비암호화(non-coding) RNA로 antisense RNA와 유사한 작용을 하는 것으로 사료된다. antisense RNA에 의한 일반적인 유전자 발현 조절에 대하여 기술하시오.
○ RNA는 폴리펩타이드를 암호화하는 mRNA만 있는 것은 아니다. 아미노산을 전달하는 tRNA, 리보솜을 구성하는 rRNA, 번역 후 단백질 배달을 도와주는 SRP RNA, 스플라이싱을 조절하는 snRNA, RNA 간섭을 일으키는 miRNA 및 siRNA, 마지막으로 아직까지 기능이 밝혀지지 않은 ncRNA가 있다.
○ 최근 RNA 간섭이 대두되고 있다. miRNA 및 siRNA가 관여하는데 mRNA와 완전 상보적인 경우 mRNA가 분해되고 부분 상보적인 경우 mRNA의 번역이 중지된다. miRNA는 시작이 단일가닥인 경우로 헤어핀 구조가 있는 반면 siRNA는 시작이 이중가닥인 경우로 헤어핀 구조가 없다는 차이가 있다. 이 중 siRNA는 실험 목적으로 곧잘 도입되는 하는데 최근 모더나 신약이 이를 이용한 것으로 잘 알려져 있다.
문제 3. CRISPR-Cas9 시스템을 이용하여 생쥐에서 유전자 편집(gene editing)을 하고자 한다. 다음 물음에 답하시오.
⑴ 3-1. 박테리아에서 CRISPR-Cas9이 작동하는 과정을 기술하고, 실제 유전자 편집기술에서 사용되는 guide RNA와의 구조적 차이를 비교·설명하시오.
○ 1st. Cas9이 이중가닥 DNA에 버블을 만들고 그 사이로 특정 염기서열과 상보적인 sgRNA (guide RNA)가 결합
○ 2nd. Cas9이 동일한 위치의 주형 DNA, 비주형 DNA, sgRNA를 모두 절단
○ 3rd. Cas9과 sgRNA가 분리
○ 4th. 버블이 형성됐던 DNA는 수소결합에 의해 다시 결합
○ 5th. DNA 수선기작을 통해 절단된 주형, 비주형 DNA는 다시 연결
⑵ 3-2. 편집과정 중에 비상동 말단연결(non-homologous end joining) 또는 상동재조합(homologous recombination) 복구가 작동하는 이유와 각 경우에서 일어날 수 있는 타켓 유전자의 편집 결과를 기술하시오. (단, 상동재조합을 통한 유전자 편집에 필요한 요소를 포함하여 기술하시오.)
○ 5th - 1st. DNA 수선기작이 없는 바이러스는 CRISPR/Cas9 메커니즘에 의해 제거됨
○ 5th - 2nd. 비상동말단 연결(nonhomologous end joining; NHEJ) : 전혀 다른 염색체가 결합할 수 있음
○ 홀리데이 모델을 따름
○ 5th - 3rd. 상동재조합 수선(homology directed repair; HDR) : 상동염색체끼리 결합하여 특정 염기의 치환 효과가 나타남
○ 상동재조합을 통한 유전자 편집에 필요한 요소는 추후 보완할 예정
⑶ 3-3. 이 시스템을 유전자 치료에 적용할 때 타켓 유전자 이외의 부위가 편집되는 문제(off-target)를 해결하는 방법을 제시하시오.
○ 최근 Streptococcus pyogenes Cas9 (SpCas9) nuclease가 효율성을 이유로 가장 많이 사용되고 있지만 off-target 비율이 높다는 이슈가 제기돼 왔다. 이에 nuclease mutation, PAM(protospacer adjacent motif) sequence modification, gRNA(guide RNA) truncation 등의 방법이 제시되었다. (ref)
문제 4. Francis Crick은 생명체에서 유전정보는 DNA → RNA → 단백질의 방향으로 흐른다고 주장하였다.
⑴ 4-1. 단백질을 암호화하는 DNA 염기서열 상의 돌연변이가 단백질의 아미노산 서열의 변화에 반영되지 않는 경우가 있다. 이 같은 현상에 작용하는 유전암호의 특성과 그에 의해 생명체의 생존에 유리할 수 있는 점을 문제 4-2의 관점을 제외하고 설명하시오.
○ 아미노산은 약 20개인데 코돈은 총 64가지가 나올 수 있어 서로 다른 코돈이 동일한 아미노산을 암호화할 수 있다. 이를 축퇴(degeneration)라고 한다. 축퇴로 인해 염기서열 상의 돌연변이가 나타나도 아미노산 서열의 변화에 반영되지 않을 수 있는데, 이를 침묵 돌연변이라고 한다. 이 경우 돌연변이 전과 후의 생존율이 달라지지 않기 때문에 생존에 대한 리스크 없이 유전적 다양성을 확보할 수 있다는 점에서 생명체의 장기 번영에 기여할 수 있다.
⑵ 4-2. 61개의 유전암호들은 단백질 번역과정 중에 해당 아미노산에 대한 tRNA의 anticodon에 의해 해석되는데 실제 세포 내에 존재하는 tRNA의 종류는 종에 따라 다르지만 대개 45개 내외뿐이다. 이런 현상을 설명하기 위해 Francis Crick이 제안한 가설을 염기쌍의 실례를 들어 설명하시오.
○ tRNA는 이론상 61개이나 실제로는 약 45개가 존재하는데 이를 워블가설이라고 하며 tRNA의 안티코돈의 첫 번째 염기와 mRNA의 코돈의 세 번째 염기사 엄격하게 결합하기 때문에 발생한다. 예를 들면 코돈표에서 CUU, CUC, CUA, CUG 모두 Leu를 암호화하는데 이 경우 세 번째 염기가 달라져도 암호화하는 아미노산이 달라지지 않았음을 알 수 있다.
입력: 2021.01.17 11:28
'▶ 자연과학 > ▷ 변리사 풀이' 카테고리의 다른 글
| 【분자생물학】 2023년 제60회 변리사 2차 국가자격시험 (0) | 2023.11.09 |
|---|---|
| 【유기화학】 2023년 제60회 변리사 2차 국가자격시험 (1) | 2023.10.21 |
| 【분자생물학】 2021년 제58회 변리사 2차 국가자격시험 (0) | 2022.12.06 |
| 【분자생물학】 2022년 제59회 변리사 2차 국가자격시험 (0) | 2022.12.06 |
| 【유기화학】 2022년 제59회 변리사 2차 국가자격시험 (8) | 2022.09.18 |




최근댓글