8강. 중심학설(central dogma)
추천글 : 【생물학】 생물학 목차
1. 개요 [본문]
2. 유전자와 유전자 발현 [본문]
3. DNA 복제 [본문]
4. 전사 [본문]
5. 번역 [본문]
6. 번역 후 단백질의 배달 [본문]
7. 번역 후 단백질의 가공 [본문]
8. 저해제 [본문]
a. DNA의 교정 및 수선
b. 미생물학의 중심학설
1. 개요 [목차]
⑴ 중심학설(생명 중심 원리, central dogma) : 유전정보의 흐름, 즉 DNA → RNA → 단백질
⑵ 레트로 바이러스(RNA 바이러스)에서 역전사 효소가 알려지고 나서 중심학설을 수정
⑶ 정보의 전달관계
① DNA 복제 : DNA → DNA
② 전사(transcription) : DNA → RNA
③ 번역 : RNA → 아미노산 서열(1차구조)
④ 트리플렛 코드(DNA) → 코돈(mRNA) → 안티코돈(tRNA) → 아미노산
2. 유전자와 유전자 발현 [목차]
⑴ 유전자와 염색체
⑵ 1 유전자 多 폴리펩티드 설
① 1 유전자 1 효소설 : 비들과 테이텀 실험(Beadle & Tatum's experiment)
○ 돌연변이체를 이용한 물질의 생합성 경로 유추 : 오르니틴, 시트룰린, 아르기닌
○ 노하우 1. 가장 많이 자라게 하는 물질일수록 생합성 경로의 뒷 단계 물질
○ 노하우 2. 돌연변이체에서 자라는 게 많을수록 앞 단계의 대사에 관여
○ 노하우 1이나 노하우 2 중 하나만 선택해서 문제를 풀자!
② 1 유전자 1 단백질설 : 효소로서 기능을 하지 않는 단백질이 밝혀짐
③ 1 유전자 1 폴리펩타이드설 : 4차 구조를 형성하는 단백질이 밝혀짐
④ 1 유전자 多 폴리펩타이드설 : 하나의 유전자로부터 여러 폴리펩타이드를 생성하는 경우가 밝혀짐
⑶ 유전자 발현의 조절단계
① 유전자 발현의 개요
○ 첫째, 우리 몸의 체세포는 동일한 DNA를 가짐
○ 둘째, 각 조직세포는 특정한 유전자만 전사시켜 유전자 발현양상이 달라 구조와 기능이 달라짐
② 전사의 조절
○ 전사 억제에 의한 조절 (원핵세포)
○ 전사 활성화에 의한 조절 (원핵세포, 진핵세포)
○ 염색체 응축에 의한 조절 : RNA 중합효소의 출입 방지(바소체)
③ mRNA 분해에 의한 조절
④ 번역의 조절
⑤ 단백질 분해의 조절(단백질 분해 효소)
3. DNA 복제 [목차]
⑴ 1st. DNA 나선을 품
① 복제는 복제 원점(ori)에서부터 양방향으로 진행 (= 복제분기점이 복제 원점에서 양방향으로 이동)
○ 복제 원점 : 수소결합이 약한 A=T 결합이 많은 지점(A=T rich site)
○ 복제분기점(replication fork) : DNA가 수소결합을 이루는 가닥과 그렇지 않은 가닥 사이의 접점
○ 원핵생물 : DnaA가 복제원점(1개)을 인식
○ DnaA
○ 공통서열. 9개 염기 × 4 + 13개 염기 × 3
○ 20-50 monomer
○ 여러 AT-rich site에 동시에 결합
○ 1분자의 ATP를 소모하여 DNA가 melt됨
○ 진핵생물 : ORC(복제원점복합체, origin recognition complex)가 복제원점을 인식
○ 복제 속도가 느려 복제원점이 여러 개
○ 효모 : 250 ~ 400개의 복제원점
○ 포유동물 : 25,000개의 복제원점. 사람의 경우 6,500여 군데
○ A-T 서열이 풍부함
○ 복제원점에 따라 결정되는 복제의 단위를 replicon이라고 함
○ ARS(자동성 복제서열, autonomic replicatoin sequence)
○ DnaA 및 ORC는 복제기포를 생성하여 수십 nt의 ssDNA(single strand DNA)를 노출시킴
② 효소 : 헬리케이스(helicase), DNA 회전효소(DNA topoisomerase), 단일사슬결합단백질이 관여
○ 헬리케이스 : 두 상보적 가닥의 수소결합을 풀어줌
○ 원핵생물 : DnaB, PcrA
○ 진핵생물 : MCM (minichromosome maintenance complex)
○ DnaB
○ 헬리케이스 특성 실험 : 결론 1, 결론 2를 얻을 수 있음
○ 결론 1. DnaA 혹은 ORC가 생성한 복제기포가 있어야 DNA에 결합
○ 결론 2. 일반적으로 DNA 복제 분기점에서 지연가닥의 주형에 결합. 5' → 3'으로 이동 (반례 존재)
○ ATP 사용. 복제기포의 최소 ssDNA 길이 : 16 뉴클레오티드 이상
○ 100 turns / sec = 6,000 rpm
○ PcrA : A1, A2, B1, B2 domain과 P-loop로 구성
○ 헬리케이스의 작용으로 DNA의 회전하중(torsion)이 심해져 구조가 불안정해짐
Figure. 1. 크기가 210 bp인 원형 DNA의 위상 이성질체(topological isomer)
2번째 이중나선은 1회전 당 nt가 적음(하중 小). 3번째 이중나선은 1회전 당 nt가 많음(하중 大)
○ L (linking number) : 고리수. 회전하중 계산시 보존되는 양으로 반시계 방향이 (+)
○ T (twist number) : 회전수. DNA는 기본적으로 우나선성으로 반시계 방향 (+)의 회전하중 존재
○ W (writhe number) : 초나선꼬임수
○ 초나선(supercoil) : DNA 분자가 자체적으로 고리를 이루는 것. 회전하중의 강화 및 완화와 관련
○ 양성초나선 : 왼나선 방향의 초나선(위 그림에서 3번째). 초나선꼬임수가 양의 값
○ 음성초나선 : 오른나선 방향의 초나선(위 그림에서 2번째). 초나선꼬임수가 음의 값
○ 직관적 이해. 양성초나선은 왼나선 방향의 초나선을 그리면서 그만큼 이중나선의 오른나선 방향의 회전하중을 강화
○ 직관적 이해. 음성초나선은 오른나선 방향의 초나선을 그리면서 그만큼 이중나선의 오른나선 방향의 회전하중을 완화
○ 호열성 세균이 아닌 대부분의 생명체는 음성초나선 구조의 DNA를 가짐
○ DNA 회전효소 : DNA의 회전하중을 완화하는 효소
○ 복제분기점에서 먼 앞쪽 부위에서 작용
○ DNA 회전효소 Ⅰ (type 1 DNA topoisomerase)
○ DNA 가닥 중 한 지점을 끊어 자연스럽게 풀리게 한 뒤 다시 결합시키는 효소
○ strand-cutting and strand-releasing activites : nuclease, ligase activities
○ ATP를 요하지 않음
○ E. coli에서 negative super coil을 완화함
○ step 1. 티로신이 인산 골격을 공격하여 nick을 만듦
○ step 2. DNA가 힘을 받아서 한 바퀴 회전
○ step 3. resealing이 진행되어 결국 supercoil이 해소됨
○ DNA 회전효소 Ⅱ (DNA gyrase, type 2 DNA topoisomerase)
○ DNA 가닥 중 두 지점을 끊은 뒤 회전(위상 변경)시키고 다시 결합시키는 효소
○ 양성초나선으로 있던 DNA 분자를 음성초나선으로 바꾸어줌
○ ATP hydrolysis가 일어남
○ fluoroquinolones (ciprofloxacin)이라는 항생제의 타겟이 됨
○ 단일사슬결합단백질
○ 원핵생물 : 단일사슬결합단백질(single strand binding protein; SSBP). monomer이고 domain이 두 개
○ 진핵생물 : RPA(replication protein A)
○ 기능 1. 수소결합이 풀린 각 DNA 단일가닥에 일시적으로 결합하여 단일가닥들이 다시 쌍을 이루지 못하게 함
○ 기능 2. DNA도 안정화하는 단백질
○ 기능 3. DNA가 꼬이지 않게 보호함
○ 실험상 SSBP가 있어야 헬리케이스의 작용이 활성화
③ 원핵세포 : 환상 DNA, 복제 원점이 1개
④ 진핵세포 : 선형 DNA, 복제 원점이 다수
○ 진핵세포의 유전체의 크기는 원핵세포의 유전체보다 크므로 많은 복제 원점을 가짐
⑵ 2nd. 프라이머 부착
① 프라이머 : DNA 중합에 틀이 되는 짧은 RNA 단편
○ PCR의 프라이머는 DNA 단편임
○ 프라이머는 대략 10 nt 정도의 크기
② 프라이머는 DNA 중합에 필요한 최초의 3’-OH기 말단을 제공
③ 효소 : 프리메이스(primase)가 관여. 프리메이스는 프라이머 합성 및 부착에 관여
○ 헬리케이스가 100 turns / s의 속도를 가지므로 프리메이스도 상당히 빠르게 작용함
○ 진핵생물 : Pol a/primase
④ primosome : RNA 프라이머가 생성되기 전에 몇 개의 단백질들이 형성하는 prepriming complex
⑶ 3rd. DNA 중합
① 효소 : DNA 중합효소(DNA polymerase)가 관여
○ 대장균의 DNA 중합효소는 DNA pol Ⅰ, Ⅱ, Ⅲ가 존재
○ DNA pol Ⅰ : 틈 번역(nick translation). 프라이머 제거 및 RNA 프라이머 자리를 dNTP로 교체
○ 10 nt / s
○ T4 연결효소는 DNA pol Ⅰ의 소단위체와 유사한 기능을 수행함
○ DNA pol Ⅱ : 외부 요인에 의해 손상된 DNA 수리, DNA 합성이 중단되면 활성화
○ DNA pol Ⅲ : DNA 신장, 이합체로 작용하며 실제로는 자기가 이동하지 않고 DNA 가닥이 움직임
○ 특징 : very high potency, fidelity, processivity
○ 1000 nt / s
○ 비대칭성 : 지연가닥과 선도가닥을 신장시킬 때 다르게 진행
○ 약 10 종류의 서로 다른 폴리펩티드 체인으로 구성됨
○ 전체 구조 : αεθβ2τ2 - αεθβ2γ2(δδ'χψ)2
○ α subunit : polymerase
○ ε subunit : 3' → 5' exonuclease
○ β2τ2 : prcessivity, star-shaped ring, 35 Å hole
○ β2 : slinding DNA clamp (활주 클램프)와 관련
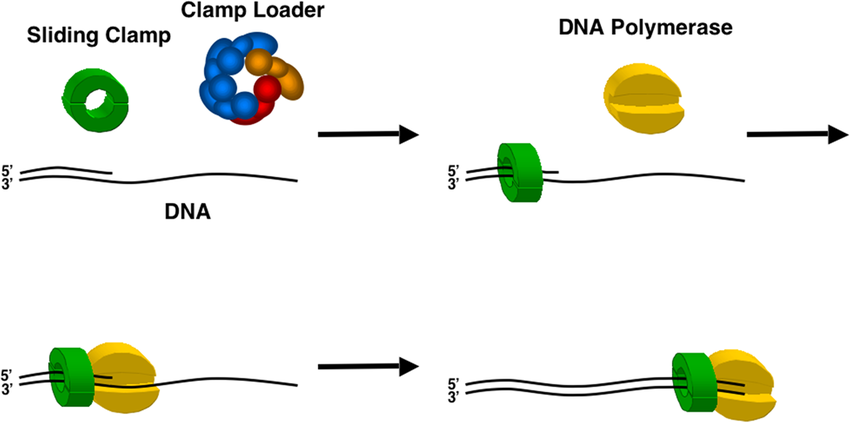
○ 활주 클램프(sliding clamp)
Figure. 2. 활주 클램프
○ 모가닥과 딸가닥이 잘 나열되도록 하여 DNA 중합효소가 실패 없이 DNA를 잘 합성하도록 함 : 정확성, 효율성 증대
○ 진핵생물 : 활주 클램프는 PCNA(proliferating cell nuclear antigen)가 관여. Ki-67 staining과 관련됨
○ 원핵생물 : 활주 클램프는 클램프 로더(clamp loader)에 의해 일시적으로 개방되어 닫힌 원형 구조의 대장균 DNA를 중앙통로에 삽입할 수 있으며, 이후 다시 닫혀 DNA 복제 과정에서 중합효소의 활동을 가능하게 함
○ 클레노우 절편(클레나우 조각, Klenow fragment)
○ (참고) DNA pol Ⅰ : 5' → 3' 중합, 3' → 5' 엑소뉴클레아제, 5' → 3' 엑소뉴클레아제
○ (참고) DNA pol Ⅰ의 대단위체 : 5' → 3' 중합, 3' → 5' 엑소뉴클레아제
○ (참고) DNA pol Ⅰ의 소단위체 : 5' → 3' 엑소뉴클레아제
○ (참고) 5' → 3' 엑소뉴클레아제 : 말단 핵산 제거 기능
○ (참고) 3' → 5' 엑소뉴클레아제 : 교정(proofreading) 기능
○ DNA pol Ⅰ의 대단위체이자 DNA pol Ⅲ와 동일
○ T4 파지의 DNA 중합효소
○ DNA 재조합에서 제한절편의 점착성 말단을 비점착성 말단으로 바꿈
○ 5' → 3' 엑소뉴클레아제 활성이 없어 주형 DNA의 프라이머를 분해하지 못함
○ 클레노우 절편을 이용한 시험관 중합반응 예시
Figure. 3. 클레노우 절편을 이용한 시험관 중합반응 예시
○ 진핵생물은 DNA pol α, β, γ, δ, ε가 존재
○ DNA pol α : 프리메이스를 포함하고 있음. proof reading X
○ DNA pol β : 수선(repair) 기능. proof reading X
○ DNA pol δ : 지연 가닥의 오카자키 절편을 신장시킴. proof reading O
○ DNA pol ε : 선도 가닥을 신장시킴. proof reading O
○ DNA pol γ : 미토콘드리아 DNA를 복제. proof reading O
② 기질 : dNTP(dATP, dTTP, dCTP, dGTP), 5’ 탄소에 3개의 인산기를 가짐
③ 주형에 상보적인 뉴클레오티드의 5' 인산기를 합성가닥의 3‘-OH기에 붙이면서 피로인산 2개 방출
○ DNA 이중나선 구조에서 3'-OH기가 정확하게 α 인산기를 공격
○ 피로인산(pyrophosphate)
④ A는 T와, G는 C와 상보적 (퓨린-피리미딘 → DNA 두 가닥 사이의 거리는 항상 일정)
⑤ 합성 방향 : 5‘ → 3’
⑥ 선도가닥(leading strand)
○ 연속적으로 합성되는 사슬
○ 복제 방향과 복제분기점의 이동방향이 같은 경우
⑦ 지연가닥(lagging strand)
○ 불연속적인 절편(오카자키 절편)으로 합성되는 사슬
○ 복제 방향과 복제분기점의 이동방향이 반대인 경우
⑷ 4th. 사슬 종결
① 종결 기작이 존재하긴 하나 기본적으로 100% 복제하기 때문에 중요하지 않음
⑸ 5th. 프라이머 제거
① RNA 프라이머 제거 효소
○ 원핵생물 : DNA 중합효소 Ⅰ
○ 진핵생물 : RNAase H, FEN1(flap endonuclease)
② RNA 프라이머가 제거되면 DNA 연결효소(리게이스, ligase, DnaG)가 DNA 사슬 연결반응을 수행
○ 리게이스는 ATP 사용
○ DnaB와 DnaG를 통틀어 프리모좀(primosome)이라고 부름
○ step 1. E + ATP (or NAD+) → E-AMP + PPi (or NMN)
○ 즉, phosphoamide 결합을 통해 ATP가 제공한 activated AMP와 DNA ligase의 lysine 잔기의 e-amino 그룹이 결합함
○ step 2. E-AMP + ⓟ-5'-DNA → E + AMP-ⓟ-5'-DNA
○ step 3. DNA-3'-OH + AMP-ⓟ-5'-DNA → DNA-3'-O-ⓟ-5'-DNA + AMP
○ 즉, activated 5' phosphorous atom이 3'-OH에 의해 친핵체 공격을 당함
③ 제거 속도
○ 1 / sec
○ 1 / 10,000,000 error
④ 딸 사슬 5' 말단에 있는 RNA 프라이머 부분은 복제되지 못함 → DNA 복제를 거듭할수록 DNA 길이가 짧아지는 이유
○ 노화와 관련
⑹ 6th. DNA 복제의 교정 및 수정 : 엑소뉴클레아제(exonuclease) 활성
① 원본 표식 : 주형가닥의 A 염기를 메틸화하여 원본임을 표시
○ Dna 메틸레이스(methylase)가 관여. ori C 부위에 있는 5'-GATC-3' 염기서열의 메틸화
② 교정 : DNA 중합효소는 교정 기능이 있음
○ (주석) single nucleotide를 교체하는 느낌
○ 3' → 5' 엑소뉴클레아제 활성
○ DNA pol I, II, III 모두 가능
○ 중합 시 복제 착오율이 1/107이 되도록 함
○ step 1. DNA 합성과정 중에 잘못된 염기를 연결 : 이 단계에서는 그 염기의 3' 말단이 노출된 상태
○ step 2. structural fluctuation이 크게 발생
○ step 3. 수소결합이 약해져서 exonuclease site로 잘못된 염기가 이동
○ step 4. hydrolysis를 통해 잘못된 염기를 제거
○ 5' → 3' 엑소뉴클레아제 활성
○ DNA pol I만 할 수 있음
○ single nucleotid씩 교체를 하지만 결국 한 번에 대략 10 nt 길이의 RNA 프라이머를 교체하는 과정
○ step 1. 오카자키 말단의 3' 말단과 바로 다음 프라이머(RNA)의 5' 말단 사이에 중합효소가 위치함
○ step 2. 한 번에 한 뉴클레오티드씩 다음 단계를 진행함
○ step 2-1. 가수분해를 통해 RNA primer를 제거
○ step 2-2. 각 RNA를 dNTP로 바꾸고 (5' → 3' polymerase activity) 교정 (3' → 5' exonuclease activity)
○ step 3. gap이 DNA로 모두 채워질 때까지 위 스텝들을 반복
○ HIV 바이러스는 수선기작을 가지고 있지 않음
③ 수선 : 수선 효소가 수정을 하면 복제 착오율은 1/109
○ (주석) multiple nucleotides를 교체하는 느낌
○ 종류 1. 이량체 회복
○ 종류 2. 절제수선 : 화학적 손상으로 발생한 비정상 염기 제거
④ DNA 교정 및 수선이 실패할 경우 돌연변이 속출
⑺ DNA 복제의 특성
① 보존적 복제 : 복제 후 기존의 DNA와 새로운 DNA가 1개씩 존재 (기각)
② 분산적 복제 : 복제 후 완전히 새로운 DNA 2개가 생성 (기각)
③ 반보존적 복제 : 복제 후 각 DNA 내 하나의 주형가닥과 하나의 복제가닥이 존재 (채택)
④ 상보성 : 주형가닥과 복제가닥은 상보적인 정보를 가짐
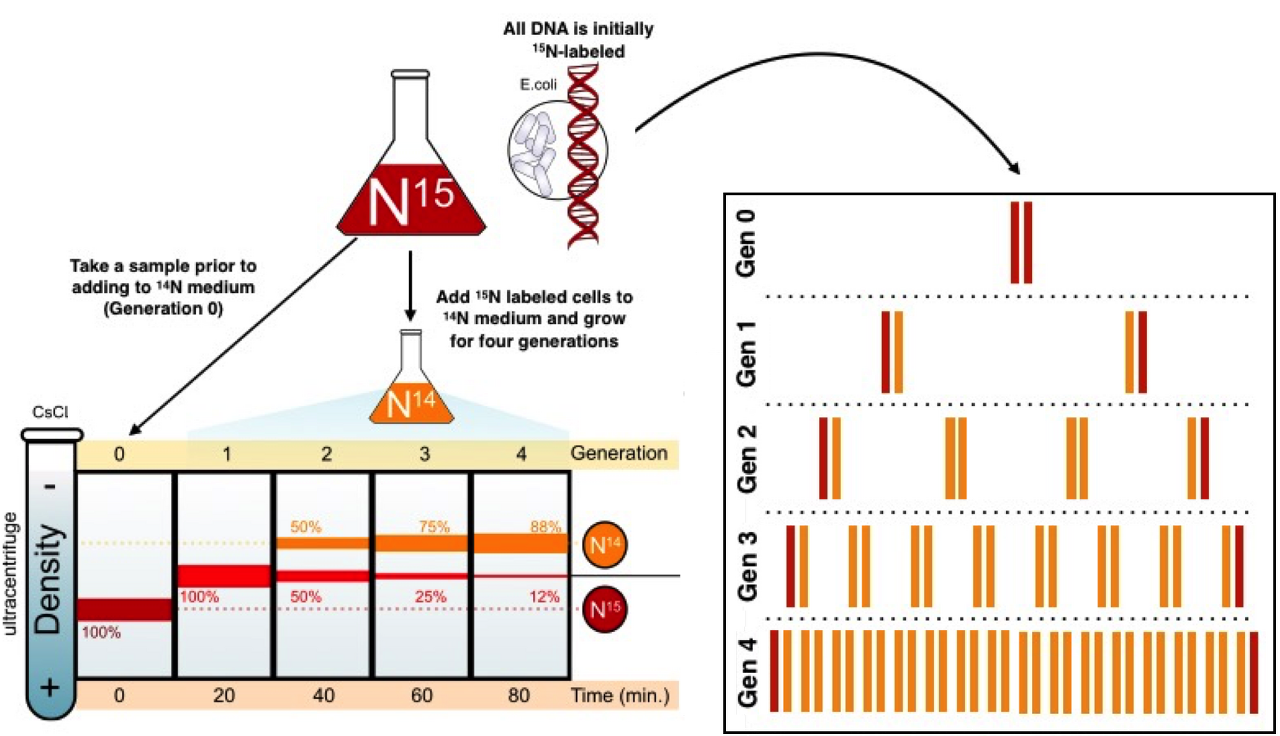
⑤ 메셀슨과 스탕의 실험(Meselson-Stahl's experiment)
Figure. 4. 메셀슨과 스탕의 실험
○ 염화세슘을 이용한 밀도 구배 원심분리 → 밀도가 다른 DNA 분리
○ 15N으로 표지한 대장균을 보통의 질소(14N)가 포함된 배지에서 매 세대마다 DNA 밀도를 분석
⑻ 텔로머레이스(telomerase) : 2009년 노벨생리의학상
① 딸 사슬 5' 말단에 있는 RNA 프라이머 부분은 복제되지 못함 → DNA 짧아짐 → 노화
○ 헤이플릭 한계(Hayflick limit) : 더 이상 분열할 수 없을 때까지의 분열 횟수
② 텔로미어(telomere)
○ DNA의 짧아짐으로부터 어느 정도까지 보호하는 역할
○ 무의미 반복서열. 6개 nt가 반복됨. 반복수는 대략 300 ~ 5000번
○ 텔로미어 상의 tetra G가 t-loop를 형성하여 노출된 텔로미어 3' 말단을 헤어핀 구조로 변형하여 말단 보호
○ 사람, 쥐, 조류, 붉은빵곰팡이, 누에나방 : TTAGGG
○ 애기장대 : TTTAGGG
○ 클라미도모나스 : TTTTAGGG
○ 효모 : TTAC(A)(C)G(1-8)
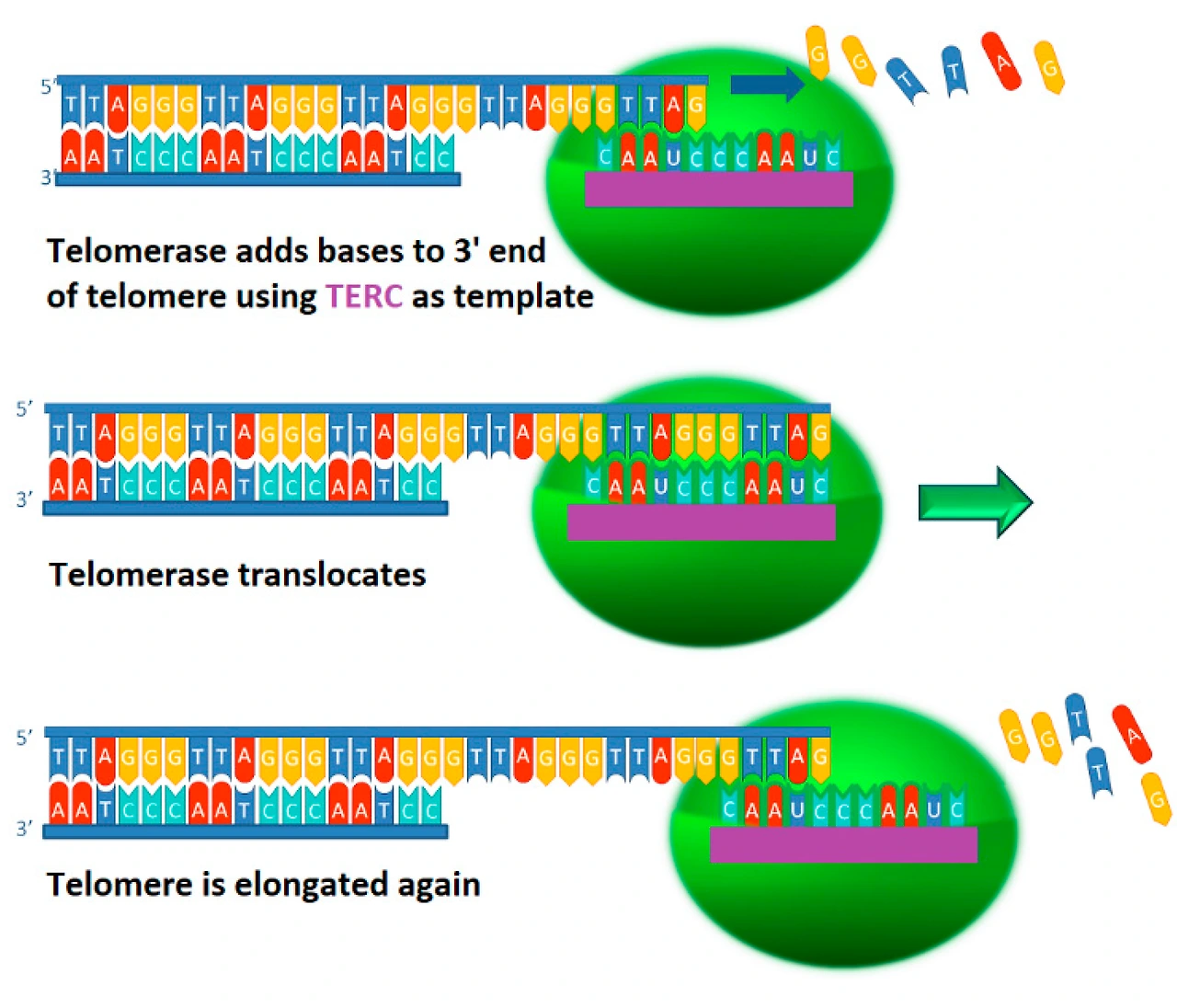
③ 텔러머레이스 : 역전사효소, RNA 의존성 DNA 중합효소
○ 분자 내 짧은 RNA가 텔로미어를 다시 길게 함
○ 분자 내 짧은 RNA : 3'-CCCAAUCCC-5' RNA 주형
Figure. 5. 3' 말단이 노출된 텔로미어와 텔로머레이스의 원리
④ 생식세포, 증식하는 정상세포, 암세포에서 발견
⑤ 마우스는 사람과 달리 텔로머레이스가 흔하게 발견됨
⑥ 환형 DNA(예 : 미토콘드리아 DNA)는 텔로머레이스가 필요하지 않음
⑼ RCR(rolling-circle replication) : 환형 DNA에서만 해당
Figure. 6. rolling-circle replication
⑽ multi-fork replication
① 상황 : 극도로 좋은 환경에서, E. coli의 번식 주기는 20분
② 의문 : DNA의 복제 속도는 1000 nt / 초이기 때문에 염색체를 모두 복제하는 데 번식 주기보다 더 긴 시간이 소요됨
③ 해결 : multi-fork replication. 이미 복제가 진행중인 딸 가닥에서 복제가 시작되는 현상으로 하나의 세포주기가 완성되기 전에 다음 세포주기를 준비하는 것
4. 전사(transcription) [목차]
⑴ DNA와 RNA 비교
① 가닥 수 : DNA는 이중가닥, RNA는 단일가닥
② 당 : DNA는 디옥시리보오스(5탄당), RNA는 리보오스(5탄당)
○ 디옥시리보오스는 2번 탄소가 -H기, 리보오스는 2번 탄소가 -OH기
③ 염기 : A, T, G, C (DNA) ↔ A, U, G, C (RNA)
④ DNA 종류
○ gDNA (게놈 DNA, genomic DNA) : 전형적인 DNA
○ 이동성 DNA
○ satellite DNA : 진핵생물 게놈에서 랜덤하게 반복되는 비암호화 DNA
○ 플라스미드(plasmid) : 박테리아 한정
○ ecDNA(extrachromosmal DNA) : 염색체 밖에 환형 DNA로 존재. 암세포에서 특정 유전자의 많은 copy number에 기여함
○ pseudogene : protein-coding 능력이 결여된 유전자 유사 그룹
○ 종류 1. 레트로트랜스포존으로 복제가 됐지만 인트론과 프로모터가 결실된 경우
○ 종류 2. 돌연변이 누적으로 유전자가 disable 된 경우
⑤ RNA 종류
○ mRNA (전령 RNA, messenger RNA)
○ tRNA (운반 RNA, transfer RNA)
○ rRNA (리보솜 RNA, ribosome RNA)
○ SRP RNA
○ snRNA : 스플라이싱과 관련
○ miRNA, siRNA
○ lncRNA 및 ncRNA : 비암호 RNA (예 : pseudogene은 전사는 되지만 번역되지 않음). lncRNA는 엑손이 적어 isoform이 적고, 발현이 적고, 조직특이성이 높음
○ sciRNA
○ ASO(antisense oligonucleotide)
○ snoRNA (small nucleolar RNA), small nuclear RNA
○ glycoRNA
○ piRNA
○ eRNA
○ lincRNA
⑵ 원핵생물과 진핵생물의 mRNA
① 폴리시스트론, 모노시스트론
○ 원핵생물 : 폴리 시스트론(= 오페론)
○ 한 mRNA 분자가 서로 다른 몇 개의 폴리펩타이드 사슬을 암호화하는 경우
○ 각 폴리펩타이드가 시작하는 부위는 리보솜 결합부위(RBS, ribosome binding site)를 가짐
○ 하나의 프로모터로 여러 유전자의 발현을 조절할 수 있다는 장점
○ 폴리리보솜(polyribosome, polysome) : 여러 리보솜이 한 mRNA에 결합된 형태
○ 진핵생물 : 모노 시스트론
○ 진핵생물에서도 폴리리보솜이 관찰되지만 극히 소량
○ rRNA : 진핵생물에서도 폴리리보솜이 관찰
② 인트론의 유무 : 진핵생물만 가지고 있음
○ 히스톤 DNA : 인트론이 없음
③ 전사와 번역의 동시성 : 원핵생물만 동시 진행
④ 전사 후 변형 과정 : 진핵생물만 존재
⑶ 1st. RNA 사슬 개시 : RNA 중합효소(RNA polymerase)가 유전자의 프로모터에 결합
① RNA는 closed promoter complex를 open promoter complex로 풀어 버림
② DNA 사슬 두 가닥이 모두 유전자를 암호화하지만, 임의의 한 유전자는 한쪽 DNA 가닥만 주형으로 사용
○ 좁은 범위에서 보면 한쪽 DNA 가닥만 주형으로 사용된다고 간주할 수 있음
○ sense 가닥 (암호 가닥, (+) 가닥, 비주형 가닥) : RNA 중합효소가 붙지 않는 사슬, 코돈 분석시 용이
○ anti-sense 가닥 (비암호 가닥, (-) 가닥, 주형 가닥) : RNA 중합효소가 붙는 사슬
○ 예 : 효모의 갈락토오스 유전자(총 6개)
○ 2번 염색체에 4개(GAL7, GAL10, GAL1, MEL1), 12번 염색체에 1개(GAL2), 4번 염색체에 1개(GAL3)
○ GAL7, GAL10, GAL1은 한 가닥에, MEL1은 다른 한 가닥에 위치
③ 프로모터(promoter)
○ 전사 개시 부위를 표시하는 약 40 base pairs의 DNA
○ RNA 중합효소가 결합하는 부위
○ 원핵생물 프로모터의 공통서열(consensus)
○ 예 1. Pribnow box (-10 box) : TATAAT (or TATGTT, ⋯)
○ 예 2. -35 box : TTGACA (or TTTACA, ⋯)
○ 진핵생물 프로모터의 공통서열(consensus)
○ 예 1. TATA box (골드버그-호그니스 박스, -25 box) : TATA
○ 예 2. CAAT box (-75 box) : CAAT
○ 예 3. GC box
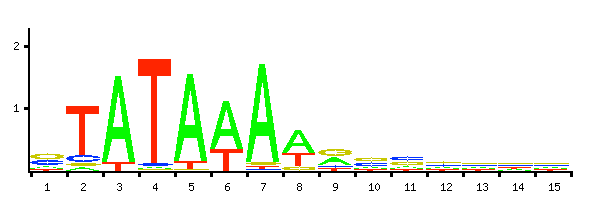
Figure. 8. TATA box motif
○ core promoter : 프로모터부터 전사가 시작되기 전까지의 약 300 bp에 해당하는 부분
④ 상류(upstream) / 하류(downstream)
○ RNA로 전사되는 첫 번째 뉴클레오티드를 +1이라 함
○ +1을 기준으로 5'쪽을 상류(-), 3'쪽을 하류(+)라 함
⑤ 대장균의 시그마 인자 : 모두 -35 상류를 인식
○ σ70 (σD) : TTGACA를 인식. 정상 생장 동안 대부분의 유전자에 관여하는 주요 시그마 인자
○ σ54 : TTGGCACA를 인식. 질소 동화에 관여
○ σ38 : CCGGCG를 인식. 정지 상태, 산화적 스트레스, 삼투적 스트레스 시 관여하는 주요 시그마 인자
○ σ32 : TNTCNCCTTGAA를 인식 (단, N은 아무 뉴클레오타이드). 열 충격 스트레스 시 관여
○ σ28 : TAAA를 인식. flagella를 합성하는 유전자들에 관여
○ σ24 : GAACTT를 인식. periplasm의 misfolded protein에 대한 반응 시 관여
○ σ19 : AAGGAAAAT를 인식. 철 이온 운반을 위한 유전자들에 관여
⑷ 2nd. 사슬 신장 (elongation of the RNA chain) : RNA 중합효소가 유전자의 DNA 염기(주형 가닥)에 상보적인 RNA 염기를 5‘ → 3’으로 중합시켜 전사의 산물로 RNA 생산
① transcription bubble = RNA transcript + DNA + RNA polymerase
② RNA-DNA hybrid
③ RNA polymerase : primer 필요 없음, topoisomerase 필요 없음, 엑소뉴클레아제 활성 없어 전사 오류율↑
④ 대장균의 DNA dependent RNA polymerase는 1가지
○ 전효소(holoenzyme) 형태 : α2ββ'ωσ
○ core enzyme 형태 : α2ββ'ω. 프로모터를 인식하는 σ70 인자의 도움으로 프로모터에 결합
○ σ70 : 프로모터 인식, 합성 개시. 재사용됨
○ β : phosphodiester bond 형성, rNTP와 결합
○ β' : DNA 주형과 결합
○ core enzyme : α 소단위체만 관여하는 경우
○ no specific binidng
○ tight non-specific DNA binding
○ Kd ≒ 5 × 10-12 M
○ holoenzyme : σ70 소단위체도 관여하는 경우
○ specific promotor binding
○ weak non-specific DNA binding
○ Kd ≒ 10-7 M
○ finds promotor 10,000 times faster
⑤ 진핵생물의 DNA dependent RNA polymerase는 3가지
○ RNA pol Ⅰ : 인에서 작용. 28S rRNA, 18S rRNA, 5.8S rRNA 합성
○ RNA pol Ⅱ : 핵질에서 작용. mRNA의 합성
○ RNA pol Ⅲ : 핵질에서 작용. tRNA, 5S rRNA, ncRNA, snRNA 합성
⑸ 3rd. 사슬 종결 (termination of the RNA chain) : terminator 서열에서 RNA 중합효소는 DNA를 떠나고 DNA는 다시 꼬임
① 원핵생물 종결자
○ 내재적 종결자 : 머리핀(hairpin-oligo-U 구조)을 형성하여 전사 종결
○ 2가 대칭서열(inverted repeat sequence)에 의해 머리핀(hairpin)이 형성
○ 2가 대칭서열에서 멀지 않은 지점에 A-T rich site가 있어 A를 주형으로 U 염기를 전사
○ 불안정한 결합인 A=U 결합 사슬이 연속되어 자연스럽게 전사체가 분리
○ Rho-의존성 종결자 : Rho 인자는 DNA-RNA 헬리케이스로 작용하여 합성 중인 RNA의 이탈을 촉진
② 진핵생물 종결자
○ 전사체의 폴리 A 형성 신호서열(5'-AAUAAA-3')이 오면 엔도뉴클레아제 활성
○ 엔도뉴클레아제가 10 ~ 35 정도 하단 부위를 절단한 후 이탈
○ 절단된 3‘ 말단에서 A 염기가 다수 연결되는 폴리 A 꼬리 형성
⑹ 4th. mRNA processing (진핵세포만 해당) : 성숙 mRNA (mature mRNA)는 핵공을 통해 세포질로 이동, 분해↓, 번역↑
① 1st. capping
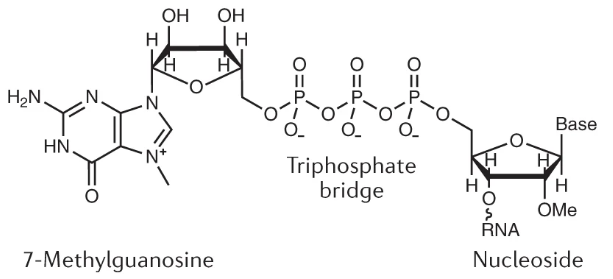
Figure. 9. 5'-cap
○ 전사와 동시에 시작
○ pre-mRNA의 5‘ 말단에 메틸화된 구아노신 유도체인 7-MeG가 5’-5‘ 결합(5' 탄소 - 3개 인산기 - 5' 탄소)으로 연결
○ 5'-MeG는 CBC(cap binding complex)와 결합하여 5'-cap을 형성 : 5'-cap을 m7GpppN이라고도 함
○ 모자(cap) : 핵공 탈출을 도와줌. mRNA를 뉴클레아제로부터 보호. 단백질 합성기구에 의해 인지되는 자리
○ IVT(in vitro transcription)를 위한 인위적인 5'-cap
○ 1세대 : mCap (Cap 0). yeast에서 유래. 방향성이 없다는 단점
○ 2세대 : ARCA(anti-reverse cap analog) (Cap 0 mimic). yeast에서 유래. 메틸기를 도입하여 방향성 부여
○ 3세대 : trinucleotide cap analog (e.g., CleanCap) (Cap 1). 생체 내 5'-cap과 가장 유사하여 우수함
② 2nd. poly A 꼬리 형성
○ poly A 꼬리는 진핵생물 전사 종결자와 관련
○ PBP(poly A binding protein)가 5'-AAUAAA-3' 뒤에 A 염기를 붙임
○ poly A 꼬리는 mRNA가 핵에서 세포질로 이동하는 것을 도와주고 mRNA의 안정성을 증가시킴
③ 3rd. 스플라이싱 : 스플라이소좀에 의해 엑손 부위만 연결. ATP 필요
○ 엑손(exon) : 실질적인 유전정보와 UTR(untranslated region)(번역 조절)을 암호화하는 부위
○ 인트론(intron) : 무의미한 정보를 암호화하는 부위
○ 무의미한 반복서열을 가지는 것은 염색질에서 DNA 간 거리를 유지시키기 위한 목적
○ 그러나 보다 심오한 의미가 있을 것으로 추정
○ 스플라이소좀(snRNP) : snRNA로 구성
○ u1 snRNP + u2 snRNP + u4 snRNP + u5 snRNP + u6 snRNP → u1 snRNP + u4 snRNP 제거
○ 스플라이싱을 위한 pre-mRNA에서 보존 서열
○ splice donor, splice acceptor : 스플라이스 접합 시작과 끝은 각각 GT, AG 모티프가 많이 관찰됨
○ 즉, 5'-GU---AG-3' : 5' splice site의 GU + 3' splice site의 AG
○ 스플라이싱 수용부 : 피리미딘 rich site, branch point A
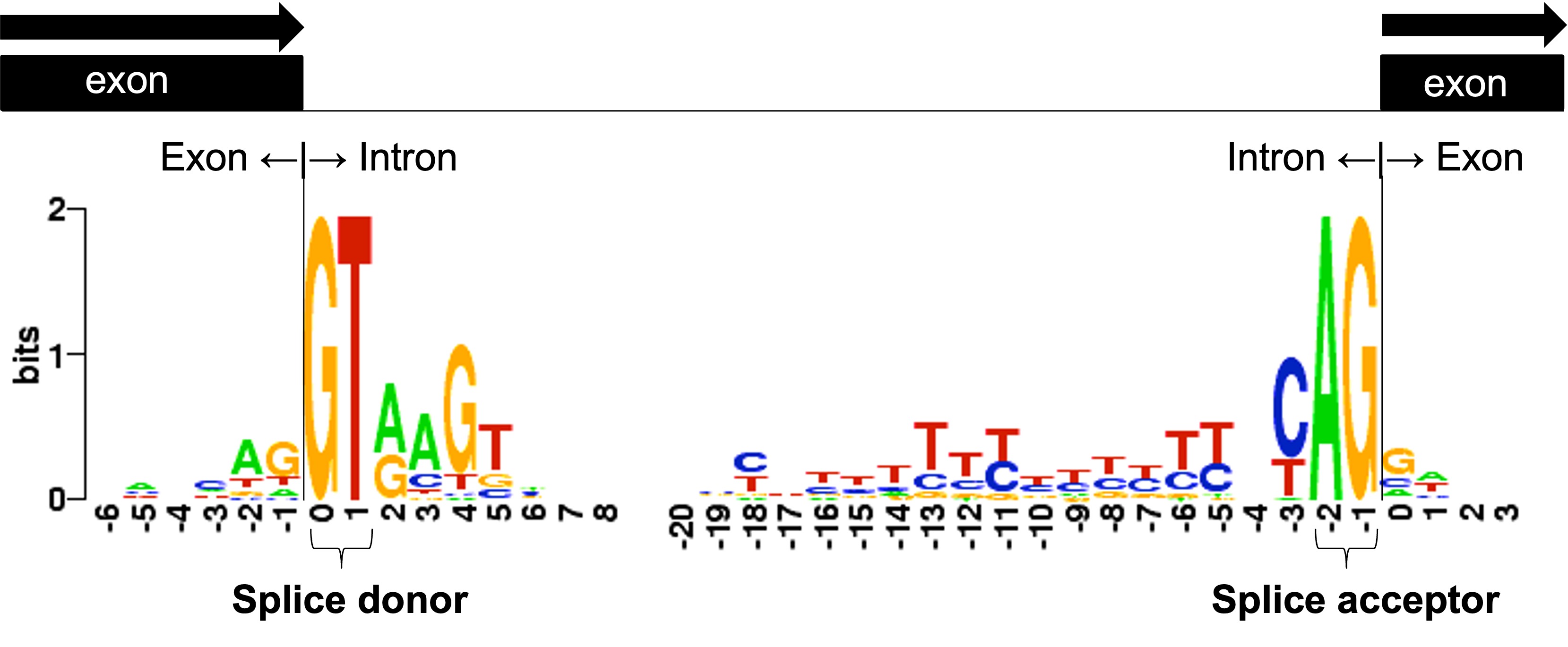
Figure. 10. 스플라이싱 공급부, 스플라이싱 수용부
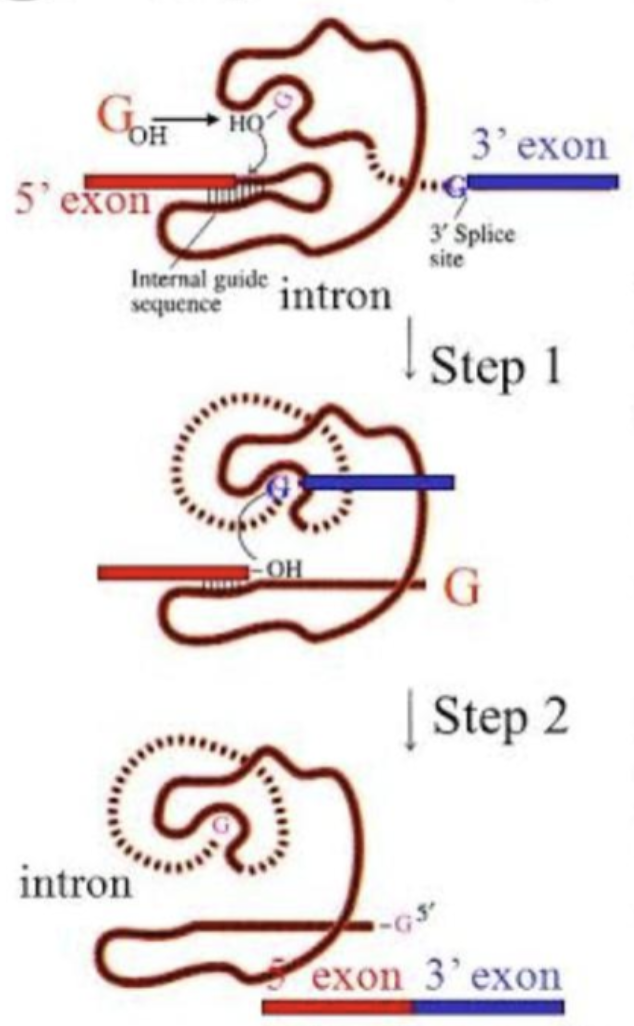
○ group I self-splicing
Figure. 11. group I self-splicing
○ 1단계 : 외부의 구아노신 분자가 5' splice site를 공격 : 이로 인해 구아노신 분자가 인트론과 연결되고 5' 엑손이 3' OH 그룹을 가짐
○ 2단계 : 5' 엑손의 3' 말단이 3' splice site를 공격 : 5' 엑손이 3' 엑손과 연결되고 그 사이의 인트론은 분리됨
○ 이런 식으로 엑손들끼리 점점 연결됨
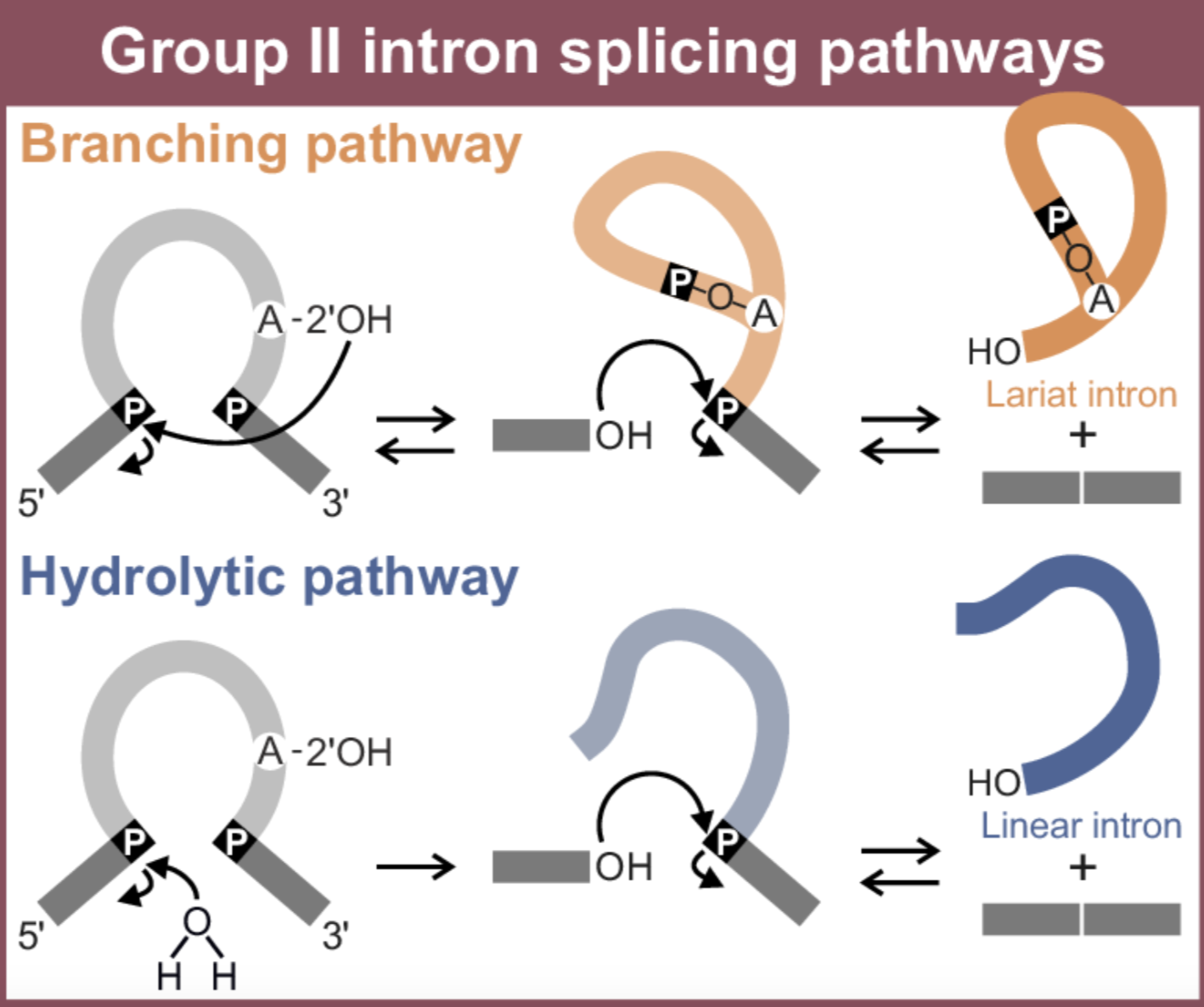
○ group II self-splicing
Figure. 12. group II self-splicing
○ 1단계 : 내부의 아데노신 분자가 5' splice site를 공격 : 라리아트(lariat) 구조를 형성
○ 2단계 : 5' 엑손의 3' 말단이 3' splice site를 공격 : 5' 엑손이 3' 엑손과 연결되고 그 사이의 인트론은 분리됨
○ 이런 식으로 엑손들끼리 점점 연결됨
○ 차이점 1. 최초의 친핵체 : group I은 외부의 구아노신 분자이고, group II는 내부의 아데노신 분자
○ 차이점 2. 구조 : group I은 복잡한 루프 및 헬릭스 구조를 특징으로 하고, group II는 Lariat 구조를 특징으로 함
○ 차이점 3. 분포
○ group I의 분포 : 박테리아의 rRNA, mRNA, tRNA; 하등 진핵생물의 미토콘드리아 게놈, 엽록체 게놈; 하등 진핵생물의 핵 게놈의 rRNA; 고등 식물의 엽록체와 미토콘드리아의 일부 몇몇 tRNA, mRNA
○ group II의 분포 : 균류, 식물, 원생생물의 세포소기관의 rRNA, tRNA, mRNA; 박테리아의 mRNA
○ 요약 : group I은 하등 생물(예 : 박테리아)에 비교적 많이 분포하고, group II는 고등 생물(예 : 균류, 식물)에 비교적 많이 분포
④ 4th. mRNA 편집(RNA editing) : RNA 서열을 바꾸는 것
○ capping, poly A 꼬리 형성, 스플라이싱보다 상대적으로 드묾
○ guide RNA가 템플릿(template)으로 사용됨
○ 유형 1. 단백질의 길이가 변하지 않는 경우
○ 유형 2. 단백질의 길이가 변하는 경우 : apolipoprotein B 단백질이 대표적
○ 사람의 apolipoprotein B 단백질은 간에서는 100 kDa의 Apo-B100이 됨
○ 사람의 apolipoprotein B 단백질은 소장에서는 48 kDa의 Apo-B48이 됨
○ cytidine deaminase가 RNA 내 5'-CAG-3'을 5'-UAG-3'으로 탈아민화 → 종결코돈이 새로운 위치에 생성
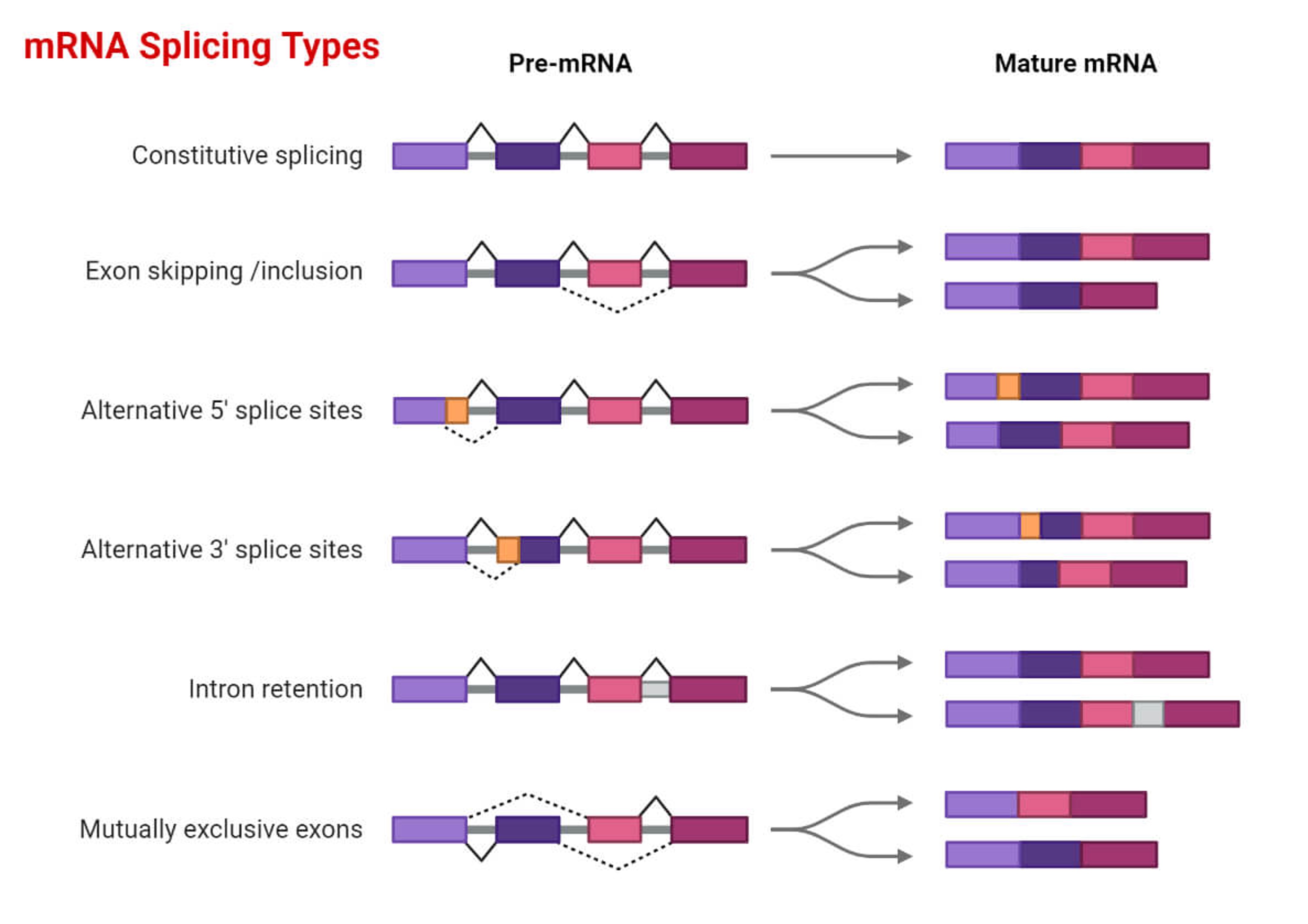
⑤ 선택적 스플라이싱(alternative splicing) : 하나의 pre-mRNA 상의 일부의 엑손만이 선택되어 여러 조합의 mRNA가 형성
○ 인간에서 인트론이 있는 유전자의 95 % 이상이 선택적 스플라이싱 有
○ 결과 1. 스플라이싱 잘못으로 정상 엑손 제거 → 암 유발 가능
○ 결과 2. 스플라이싱 결과 종결코돈이 빨리 나옴 → NMD(nonsense-mediated mRNA decay)로 비정상 mRNA 제거
○ alternative splicing event
Figure. 13. mRNA splicing 종류
○ SE(skipped event) : ~40%. 특정 exon 전체가 포함되거나 포함되지 않는 경우
○ A5SS(alternative 5' splice site) : ~7.9%. exon 전체가 아닌 exon의 5'의 splice junction이 다르게 사용
○ A3SS(alternative 3' splice site) : ~18.4%. exon 전체가 아닌 exon의 3'의 splice junction이 다르게 사용
○ RI(retained intron) : <5%. 아미노산 서열을 코딩하지 않는 intron이 유지되거나 splicing이 되는 경우
○ MXE(mutually exclusive exon) : 하나의 exon이 포함되는 경우에는 다른 exon은 splice가 되고, 그 하나의 exon이 splice 되는 경우에는 다른 exon이 포함되는 배타적 스플라이싱. MXE는 뇌 조직에서 주로 관찰됨
○ alternative promoter
○ alternative poly-A
○ 다양한 헤모글로빈의 종류는 선택적 스플라이싱이 아니고 시기 특이적 발현에 불과함을 유의
⑥ 엑손 셔플링(exon shuffling) : 서로 다른 유전자간 교차로 인해 새로운 조합의 엑손으로 구성된 유전자가 생성
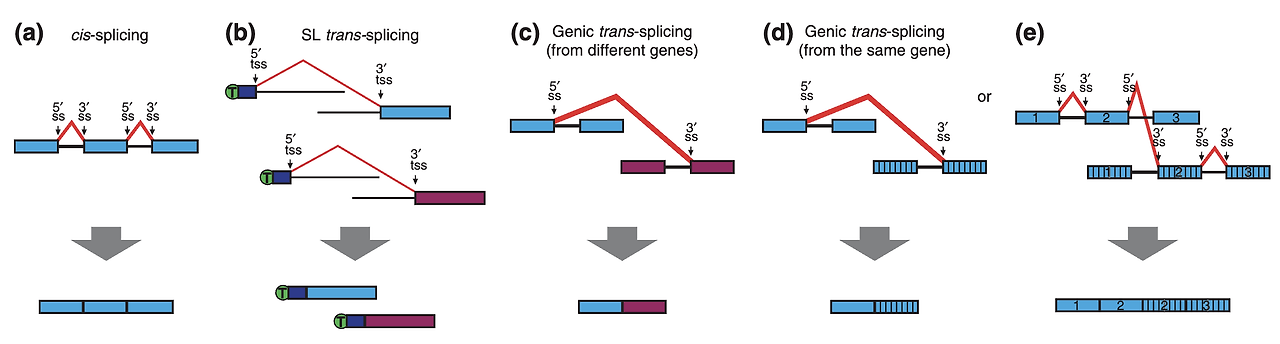
⑦ 트랜스 스플라이싱(trans-splicing)
Figure. 14. cis/trans splicing
○ 종류 1. cis-splicing : 한 pre-mRNA 내의 엑손 간에 5' ss (splice site) to 3' ss 반응, 즉 연결이 진행되어 mRNA를 만드는 과정
○ 종류 2. trans-splicing : 두 개의 서로 다른 pre-mRNA가 하나의 mRNA 분자로 연결되는 과정
○ 2-1. SL trans-splicing : 5' tss (trans splice site) 측 spliced leader (SL)와 3' tss 측 pre-mRNA가 연결되는 과정
○ 2-2. 서로 다른 유전자 간의 trans-splicing
○ 2-3. 같은 유전자 간의 trans-splicing : 주형의 방향이 다르거나 모계/부계 origin이 다를 수 있음. exon duplication도 가능
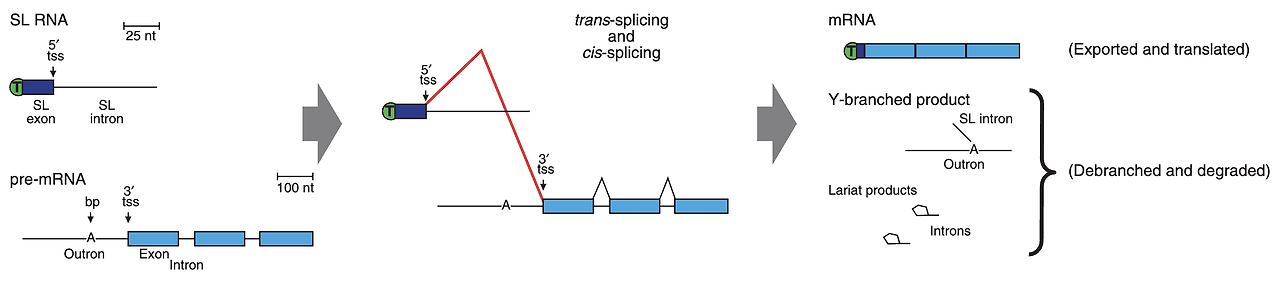
○ SL trans-splicing 과정
○ 1단계. SL RNA에 있는 5' tss와 pre-mRNA에 있는 3' tss가 결합
○ 2단계. 성숙 mRNA는 SL exon, 5' 말단에 있는 TMG cap을 가지고 있고, 세포질에서 번역됨
○ 3단계. splicing 결과 Y형 생성물 (SL intron + pre-mRNA outron)이 생성 : cis-splicing에서 생성된 lariat intron 생성물과 유사
○ 4단계. Y형 생성물은 빠르게 분해됨
Figure. 15. SL trans-splicing 과정
⑧ mRNA의 안정성 조절 (수명 조절)
○ 수명이 긴 mRNA는 더 많은 단백질로 해독
○ 원핵생물 mRNA는 수명이 2 ~ 3분 정도
○ 진핵생물 mRNA는 수명이 수 일 ~ 수 주 정도
○ 예 1. 헤모글로빈의 mRNA는 적혈구가 살아 있는 한 분해되지 않고 계속 남아 있음
○ 글로빈 mRNA는 적혈구에 상당히 많이 발현하며 전체 blood RNA의 2/3을 차지함
○ 예 2. 트랜스페린(transferrin) : 철 도입-수송 단백질
○ 철 반응요소(IRE, iron response element) : 암호화 부위 하류에 존재. AT-rich sequence
○ Fe3+ ↑ : IRE-BP가 불활성 → 트랜스페린 mRNA의 poly A 서열 제거 → mRNA가 분해됨
○ Fe3+ ↓ : IRE-BP가 활성 → 트랜스페린 mRNA의 poly A 서열 보호 → mRNA가 안정화 → 발현
⑺ 5th. rRNA, tRNA 전사
① rRNA는 RNA pol Ⅰ에 의해 인에서 전사됨
② mRNA, microRNA는 RNA pol Ⅱ에 의해 핵질에서 전사됨
③ tRNA는 RNA pol Ⅲ에 의해 핵질에서 전사됨
④ rRNA의 프로모터는 유전자 상류 멀찍이 존재
⑤ mRNA, microRNA의 프로모터는 유전자 상류 가까이 존재
⑥ tRNA의 프로모터는 유전자 하류 가까이 존재
⑦ TBP(TATA-binding protein)는 모두 필요
⑻ 6th. rRNA processing, tRNA processing
① rRNA processing
○ 종류 1. 인에 있는 45S rDNA → 18S rRNA + 5.8S rRNA + 28S rRNA
○ 종류 2. 비메틸화된 염기를 절단
② tRNA processing : pre-tRNA에서 3'과 5'을 약간 절단하는 것
⑼ 원핵생물 전사의 조절
① 발현조절 요소
○ 프로모터(P), 작동유전자(O)(억제자 자리) 공통 서열
○ 활성자 자리
○ 예 : 대장균 cAMP-CAP(Catabolite Activator Protein) 복합체 인식 부위
② 오페론(operon) : 여러 유전자를 하나의 mRNA에 의해 전사되도록 하여 통합 조절을 꾀한 것
③ 레귤론(regulon) : 여러 오페론이 하나의 단백질에 의해 조절되도록 한 것
○ 예 : 대장균의 maltose regulon
④ 전사 억제자에 의한 조절
○ 작동 유전자에 억제인자가 결합 시 RNA 중합효소의 프로모터 결합을 억제
○ 유도성 오페론 : 유도자(inducer)가 있는 경우 작동유전자에 결합한 억제인자를 분리시켜 RNA 중합효소가 프로모터에 결합할 수 있게 하여 전사 개시
○ 예 1. 분해 오페론 : 젖당 오페론(3 genes)
○ 억제성 오페론 : 유도자가 없는 경우 작동유전자에 결합한 억제인자를 분리시켜 RNA 중합효소가 프로모터에 결합할 수 있게 하여 전사 개시
○ 예 1. 합성 오페론 : 트립토판 오페론
⑤ 전사 활성자에 의한 조절
⑽ 오페론(operon) : 하나의 프로모터·작동부위 아래에 몇 개의 구조유전자에 대한 ORF가 존재하여 동시에 발현되는 것
① 개요
○ 유도성 오페론 : 관심 물질이 최종적으로 오페론의 발현을 촉진하는 것
○ 억제성 오페론 : 관심 물질이 최종적으로 오페론의 발현을 억제하는 것
○ 양성 조절 : 활성자에 대한 조절, 활성자를 유도성 오페론은 관심물질이 활성화하고 억제성 오페론은 억제
○ 음성 조절 : 억제자에 대한 조절, 억제자를 유도성 오페론은 관심물질이 억제하고 억제성 오페론은 활성화
② 젖당 오페론(lac 오페론) : 유도성 오페론
○ lac 오페론의 구성 : (조절자) - CAP 결합 부위 - 프로모터 - 작동부위 - lac Z - lac Y - lac A
○ 조절자 (lac I) : 억제자를 암호화하는 부위. 자체 프로모터가 있기 때문에 오페론에 포함시키지 않음 (항시 발현)
○ 작동부위(operating site)
○ 구조유전자 1. 베타갈락토시다아제(β-galactosidase, lac Z)
○ 베타갈락토시다아제 : 젖산 분해효소, 젖당을 알로젖당으로 전환
○ lac Z는 사실 베타갈락토시다아제의 N 말단 부분인 α 펩티드를 만듦
○ α 상보성(α complementation) : α 펩티드와 숙주의 다른 펩티드가 베타갈락토시다아제를 형성하는 것
○ 구조유전자 2. 베타갈락토시드 투과효소(premease, lac Y) : 젖당수송체, 세포막에 발현
○ 구조유전자 3. 베타갈락토시드 아세틸기전이효소(transacetylase, lac A)
○ 베타갈락토시드 아세틸기전이효소 : 부산물 제거
○ lac 오페론의 음성적 조절
○ lac 오페론의 억제자는 작동부위에 결합할 수 있음
○ lac 오페론의 억제자는 lac I 유전자에서 만들어지며 RNA pol의 프로모터 결합을 억제
○ lac 억제물질이 젖당의 변이체인 알로락토스(allolactose)와 결합하면 작동부위와 결합 못함 (젖당이 유도자)
○ lac 오페론의 양성적 조절
○ 배경 : lac 오페론의 프로모터는 RNA pol과 친화력이 약해 억제물질이 없어도 전사가 잘 안 일어남
○ 아데닐고리화효소(AC)는 ATP로부터 cAMP를 만듦
○ 대사활성자단백질(CAP)은 cAMP와 결합하여 복합체 형성
○ CAP-cAMP 복합체는 RNA pol의 프로모터 결합을 도움
○ 포도당은 알로스테릭 억제부위를 통해 아데닐고리화효소를 억제할 수 있어 위 도움 기전이 작동하지 못하게 함
○ 결과 : lac operon 작동보다 효율적인 포도당을 우선적으로 사용하여 lac operon 상의 불필요한 효소를 합성하지 않음
○ lac 오페론은 포도당과 젖산이 있는 배지에서의 이중영양적 생장(diauxic growth)의 메커니즘을 제공
○ 포도당 O, 젖당 O : cAMP 감소 → 양성적 조절 감소 → 젖산 분해효소 생성 ×
○ 포도당 O, 젖당 X : cAMP 감소 → 양성적 조절 감소 → 젖산 분해효소 생성 ×
○ 포도당 X, 젖당 O : 알로락토오스↑ → 억제자의 작동부위 결합↓ → 젖산 분해효소 생성
○ 포도당 X, 젖당 X : 알로락토오스↓ → 억제자의 작동부위 결합↑ → 젖산 분해효소 생성 ×
○ lac 오페론에서 파생된 오페론
○ tac 오페론
○ lac I- : 억제자에 결함이 있어 작동부위에 결합하지 못함. 항상 활성
○ lac IS : 젖당이 억제자에 결합하지 못함. 항상 비활성
○ lac Oc : 작동부위에 결함이 있어 억제자가 작동부위에 결합하지 못함. 항상 활성
○ lac Z- : 결함이 있는 lac Z를 암호화함. 오페론과 무관하게 β-갈락토시다아제 발현 ×
③ 트립토판 오페론(trp 오페론) : 억제성 오페론
○ trp 오페론의 구성 : (조절자) - 프로모터 - 작동부위 - 구조유전자
○ 조절자 : 억제자를 암호화하는 부위. 자체 프로모터가 있기 때문에 오페론에 포함시키지 않음(항시 발현)
○ 트립토판의 생합성 과정에 필요한 5가지 효소를 암호화한 5개의 구조유전자 trp E, D, C, B, A
○ 각각 ξ 폴리펩티드, δ 폴리펩티드, indole glycerolphosphate synthase, β 폴리펩티드, α 폴리펩티드
○ 조절기작 1. 음성적 조절
○ trp 프로모터는 RNA 중합효소에 대해 상대적으로 높은 결합력 → 활성화 단백질 필요 ×
○ 조절유전자(trp R)에 의하여 trp 억제물질은 trp과 결합하여 작동부위에 붙음 (corepressor 작용)
○ 조절기작 2. 감쇄 조절기작(attenuation)
○ trp mRNA는 상보적으로 염기쌍을 형성할 수 있는 4개의 부분이 있음. 각각 trp 암호화 부위가 근처에 위치
○ 선도서열 : 단백질로 번역되지만 트립토판의 생합성과는 무관
○ (주석) 선도펩티드에서 '선도'의 의미는 단순히 트립토판 효소보다 먼저 만들어진다는 의미
Figure. 18. trp 오페론의 DNA 서열
Figure. 19. trp 오페론의 선도펩티드 mRNA 서열
폴리 U 이후에 trp E, D, C, B, A 서열이 존재함
○ 경우 1. 트립토판이 부족한 경우 : RNA pol이 4번까지 전사를 해도 트립토판 tRNA가 부족하여 trp 유전암호를 번역 속도가 느림 → 리보솜이 1번 서열에 위치 (선도펩티드 합성 중) → 2번과 3번이 헤어핀 형성 → RNA 중합효소와의 거리가 멀어 전사 종결기작(내재적 종결자)이 일어나지 않음 → trp mRNA 2가 생성
○ 경우 2. 트립토판이 풍부한 경우 : RNA pol이 4번까지 전사를 하면 번역속도가 빨라 리보솜이 1-2 서열에 위치 → 3번과 4번이 헤어핀 형성 → 전사 종결 (내재적 전사종결) → trp mRNA 1이 생성
○ 경우 3. 트립토판이 전무한 경우 : RNA pol이 4번까지 전사를 해도 mRNA에 결합한 리보솜이 전무 → 3번과 4번이 헤어핀 형성 → 전사 종결 (내재적 전사종결) → trp mRNA 1이 생성
Figure. 20. trp 오페론의 감쇄조절기작에서 헤어핀 형성
○ trp L에서 트립토판의 코돈이 결실된다면 리보솜이 트립토판 아미노산을 기다릴 필요가 없음 : 리보솜이 트립토판 아미노산을 기다리는 시간이 짧은 경우인 경우 2와 동일한 논리로 trp mRNA 1이 생성됨
④ tac 오페론
○ IPTG(isopropylthiogalactoside) : 알로락토스와 같이 lac 및 tac 프로모터를 활성화하는 인공 물질. 분해되지 않아 일정한 농도를 유지하며 항상 프로모터 활성화
○ lac 프로모터처럼 IPTG가 있어야 발현이 되는 인공 프로모터
○ trp 프로모터보다 발현이 강력함
○ 억제자는 lac I 또는 lac IQ에서 만들어짐
○ lac IQ : lac I보다 더 강하게 발현되도록 돌연변이 된 억제자 유전자
⑤ PL 오페론
○ cI repressor(cI 857) 유전자 : 저온에서 cI repressor를 만들어 PL 프로모터를 비활성화
⑥ T7 오페론
⑦ 부분이배체 : cis-trans 이배체
○ 부분이배체 : 대장균의 유전자 일부만 F 플라스미드가 가지고 있다면, F 플라스미드에 있는 유전자들은 이배체 상태
○ 부분이합체 : 성선모를 통한 접합. 부분이배체로 간주
○ cis-acting element : 부분이배체에서 자기 오페론에만 영향을 주는 프로모터. 상시 동일 작용
○ 예 : lac 파생 오페론에서 Oc
○ trans-acting element : 부분이배체에서 2배체에 모두 영향을 주는 프로모터. 조건부 작용
○ 예 : lac 파생 오페론에서 I-, Is
⑧ 재조합 단백질과 유도성 프로모터
○ 재조합 단백질을 계속 만드는 세포는 대사적 부담이 있음 : 세포 성장 저해, 플라스미드 불안정, 생산 수율 저하
○ 발현 전략 : 세포 농도를 어느 정도 올려준 후 유도물질을 첨가하여 발현을 유도
○ 결론 : 생산 수율을 높이기 위해 발현조절이 가능한 유도성 프로모터가 널리 사용됨
⑾ 진핵생물 전사의 조절
① 전사인자(transcription factor)
○ 개요
○ 진핵생물의 RNA 중합효소는 단독으로 프로모터에 결합할 수 없음
○ 유전자 상류의 다양한 조절 서열(예 : TATA box, CAAT box)에 보편전사인자와 특수전사인자가 결합했을 때 RNA 중합효소가 전사 시작
○ 전사인자 개수는 대략 1600개
○ pioneer factor : 염색질(chromatin)의 닫힌 상태에서도 결합할 수 있는 특수한 전사인자. 먼저 결합하는 전사인자. 훨씬 적음
○ CTCF 전사인자는 다른 TF보다 길게 (~수 분) DNA에 결합
○ 보편전사인자(general TF) : 각 세포에 보편적으로 존재 (예 : TFIIA, TFIIB, TFIID, TFIIE, TFIIF, TFIIH, TFIIJ 등)
○ TFIID : 13개의 서로 다른 단백질 complex
○ TFIIH : 10개의 서로 다른 단백질 complex
○ 특수전사인자(specific TF) : 특이적으로 존재
○ 예 1. DBD(DNA-binding domain)
○ 예 2. AD(activation domain)
○ 예 3. SP1 : zinc finger. 5'-GGGCGG-3'을 인식. monomer
○ 예 4. AP-1 : basic zipper. 5'-TGA(G/C)TCA-3'을 인식. dimer
○ 예 5. C/EBP : basic zipper. 5'-ATTGCGCAAT-3'을 인식. dimer
○ 예 6. heat shock factor : basic zipper. 5'-XGAAX-3'을 인식. trimer
○ 예 7. ATF/CREB : basic zipper. 5'-TGACGTCA-3'을 인식. dimer
○ 예 8. c-Myc : basic helix-loop-helix. 5'-CACGTG-3'을 인식. monomer
○ 예 9. Oct-1 : helix-turn-helix. 5'-ATGCAAAT-3'을 인식. monomer
○ 예 10. NF-1 : novel structure. 5'-TTGGCXXXXXGCCAA-3'을 인식. dimer
○ 근거리 조절요소 : 프로모터 근거리에 존재
○ 1st. TFIID가 TFIID 상의 TBP 서브유닛을 통해 TATA 박스에 결합
○ 2nd. TFIIA와 TFIIB가 전체 복합체를 안정화
○ 3rd. TFIIF와 RNAPII (RNA polymerase II)가 복합체에 결합
○ RNAPII recruting에 GTF가 관여함
○ 4th. TFIIE와 TFIIH가 추가로 복합체에 결합
○ TFIIH는 인에서 RNAPII의 CTD(C-terminal domain)를 인산화 → TFIID 방출 및 헬리케이스 활성
○ RNAPII는 α2ββ'ω와 달리 헬리케이스 활성이 없음
○ 5th. 전사가 개시됨 : PIC(pre-initiation complex) 형성
○ 전사 개시에도 GTF가 관여함
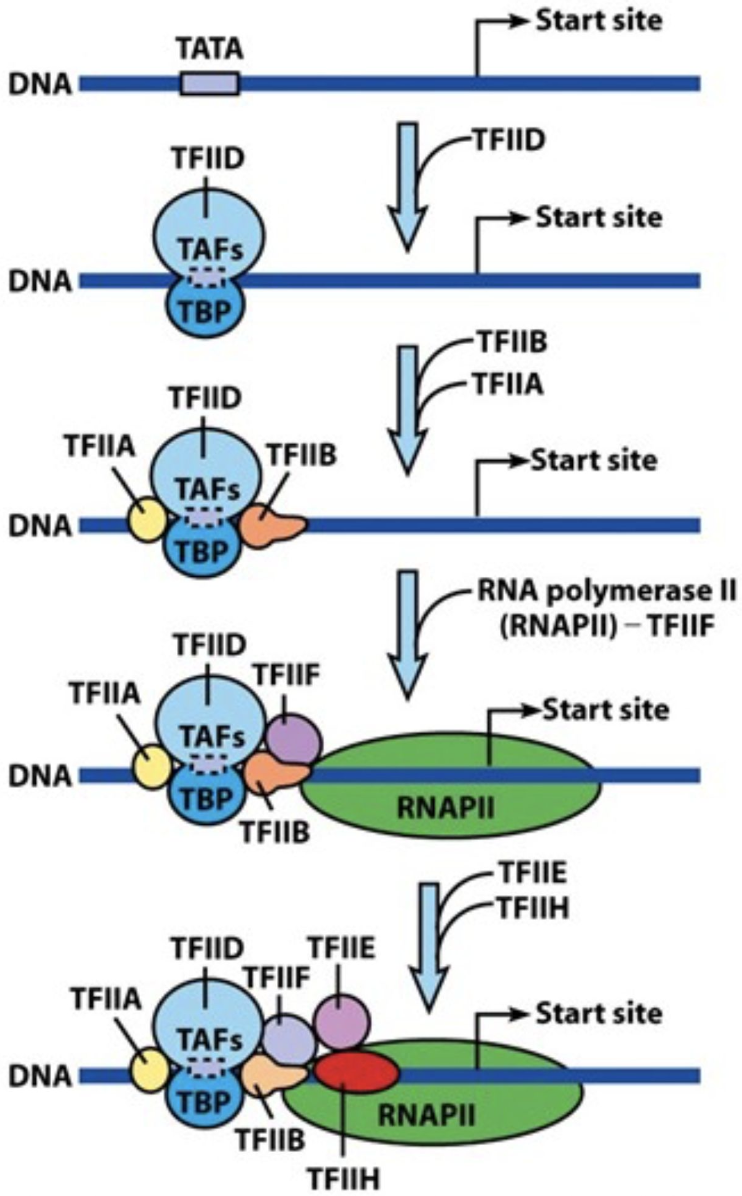
Figure. 21. PIC 형성 과정
○ 원거리 조절자 : 프로모터 상류 부위에 주로 분포
○ 인핸서(증폭서열, enhancer) : RNA 중합효소의 프로모터 결합 촉진. 상류나 하류 모두에 있을 수 있음. 길이는 50-1500 bp 정도
○ 사일렌서(침묵서열, silencer) : RNA 중합효소 무조건 억제. 프로모터 앞에 위치
○ 인슐레이터(insulator) : 인핸서를 억제. 인핸서와 프로모터 사이에 존재함
○ 스트레스 반응요소(SRE, stress response element)
○ DNA 결합 단백질
○ HMG 단백질 : DNA pol Ⅱ가 작용할 수 있도록 DNA를 구부리는 역할을 함
○ DNA 결합 단백질의 공통구조(DNA binding motif)
○ helix-turn-helix motif : 두 개의 알파나선, CAP·오페론 작동 부위 등
○ zinc finger motif : 스테로이드 호르몬에서 관찰
○ leucine zipper motif (예 : AP-1는 포유동물의 세포생장과 분열에 관여)
○ helix-loop-helix motif
○ 마우스와 인간 게놈은 10,000개 이상의 루프로 접혀 있음
② 수식 과정(pre mRNA → mature mRNA)의 조절
③ DNA 메틸화 (DNA Methylation) : 후성유전학(epigenetics)의 대표적인 개념
○ methyl-CpG binding domain containing protein (MBD)
○ MBD1 ~ MBD6
○ MeCP2 : Rett syndrome과 관련 있음
○ CpG dinucleotide는 거의 항상 메틸화돼 있음
○ 전사개시부위(TSS) 근처 프로모터의 약 70%가 CpG island와 교차 : 유전자가 발현 시 CpG island는 unmethylated
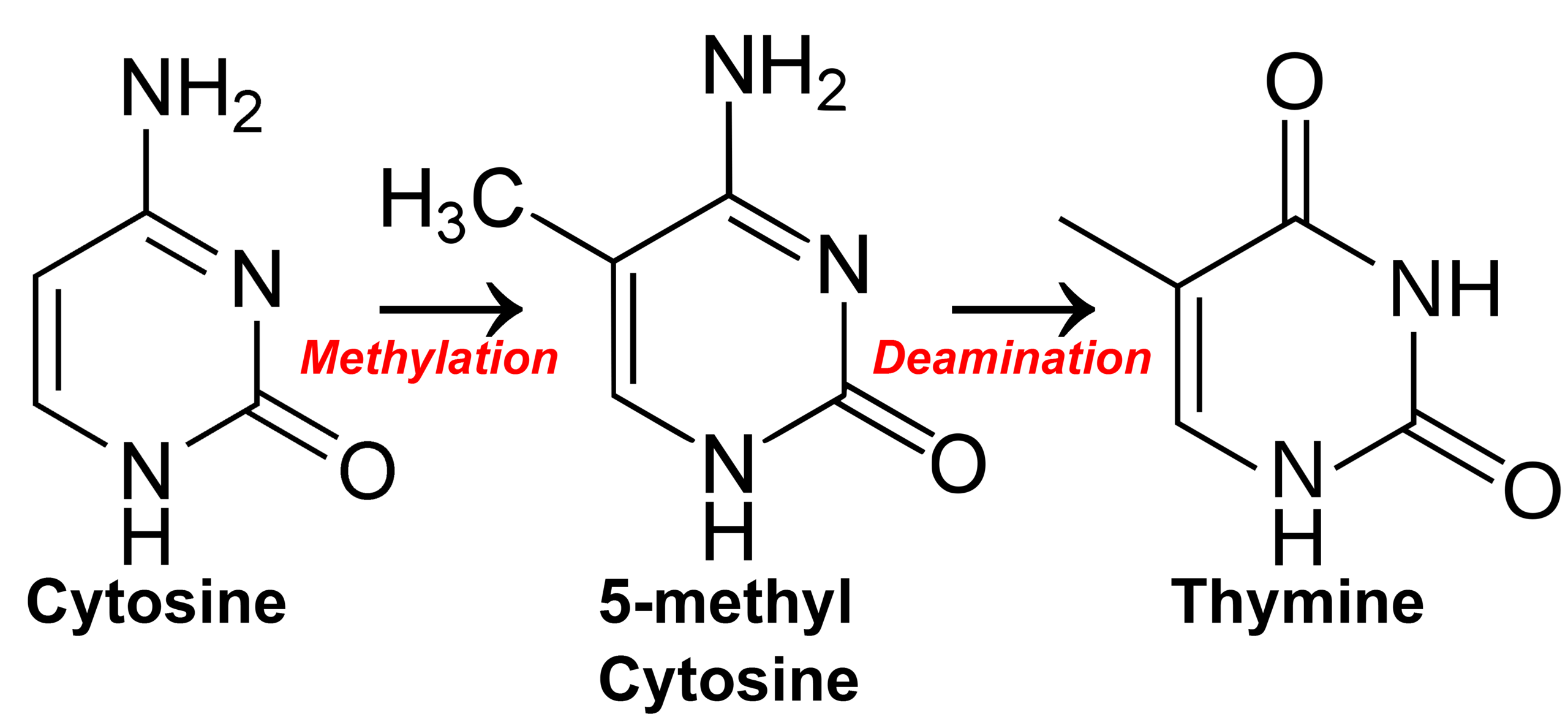
○ 사이토신의 탈아미노화 : 메틸기는 C 염기에 붙음 → C 염기는 deamination을 거쳐 T 염기가 됨 → CpG는 게놈에서 드묾
Figure. 22. 사이토신의 탈아미노화
○ 인간 유전체의 70 ~ 80%가 메틸화
○ global methylation map
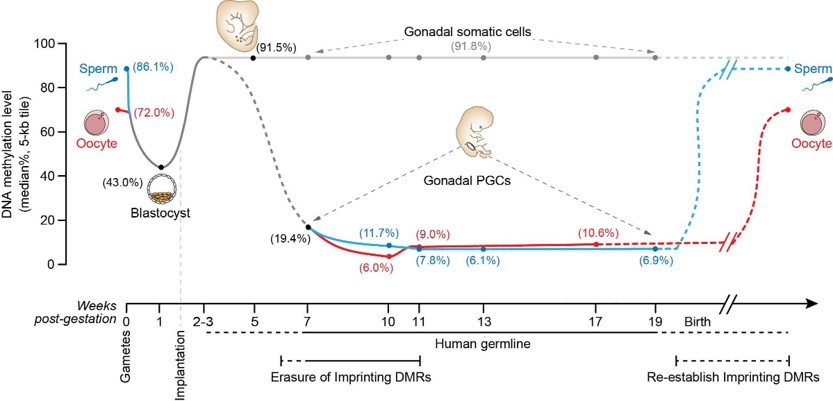
Figure. 23. global methylation map
○ 메틸기가 붙어 있는 염색질은 응축하여 전사가 불활성화
○ 발현정도 : 진정염색질 > 이질염색질
○ 프로모터 및 전사인자 결합부위에 C 염기가 많음 (예 : GC box)
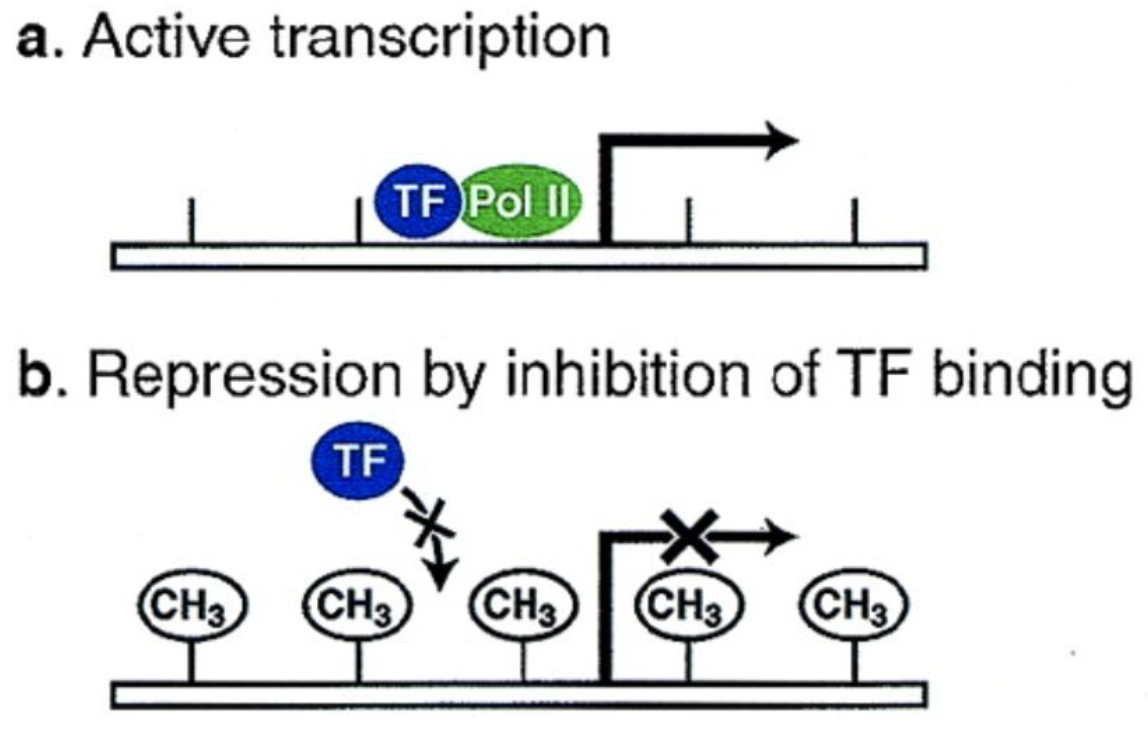
Figure. 24. DNA 메틸화와 전사 불활성화
④ 히스톤 수식
○ 히스톤 중 H3가 가장 수식이 많이 일어날 수 있음 : H2A, H2B는 훨씬 드물게 수식됨
○ 히스톤 수식은 히스톤 꼬리에서 일어남
○ 히스톤 아세틸화 : 아세틸기는 히스톤 꼬리의 리신 잔기에만 결합함
○ 아미노산인 리신의 작용기의 NH2기가 염기로 작용하여 양전하를 띰 → 히스톤의 응축에 핵심적인 역할 수행
○ 리신의 NH2기에 아세틸기(-COCH3)가 결합하면 히스톤 중성화가 이루어짐
○ 히스톤 중성화 → DNA-히스톤 간 정전기적 결합 약화 → 진정염색질화 및 전사 활성화
○ 히스톤 H3 리신 9번 잔기에서의 아세틸화가 대표적
○ 히스톤 아세틸 전달효소(histone acetyltransferase, HAT)
○ 종류 1. HAT A
○ 핵 안에 위치한 HAT A는 뉴클레오좀 안에 있는 히스톤을 아세틸화시킴
○ 히스톤을 아세틸화하면 양전하를 띠는 리신의 NH2기가 중성이 되어 뉴클레오솜 응축이 풀림
○ 종류 2. HAT B
○ 세포질에 위치한 HAT B는 세포분열 중에 일어나는 chromatin assembly에 필요한 free histone을 아세틸화함
○ HAT B는 세포질에 있는, 새로 합성된 히스톤 H4 리신 잔기와 상호작용을 함 : HAT1/HAT2/H4
○ 그 뒤 새로 합성된 히스톤 H3가 히스톤 H4와 결합한 뒤 karyopherin 도움으로 핵 내로 이동함 : HAT1/HAT2/H3/H4
○ HAT1/HAT2/H3/H4 구조가 heterodimer인지 heterotetramer인지 아직 결정되지는 않았음
○ HAT1/HAT2/H3/H4 복합체는 DNA 상에 H3/H4 complex를 전달함
○ 히스톤 탈아세틸화 : 히스톤 아세틸화의 반대
○ 발프로익산 : 히스톤 탈아세틸화 효소의 저해제
○ 히스톤 메틸화
○ 히스톤 꼬리의 리신 혹은 아르기닌에서 일어남 : 히스톤 아세틸화가 리신에서만 일어나는 것과 대비됨
○ 히스톤 양전하 증가 → DNA-히스톤 간 전기적 결합↑ → 이질염색질화 및 전사 불활성화
○ 3번까지 같은 자리를 메틸화 할 수 있음 : mono-methylation, di-methylation, tri-methylation
○ 히스톤 메틸화가 유전자 전사를 활성화하기도 함
○ 히스톤 인산화 : 인산화 작용기마다 전사 활성·불활성이 다름
○ 히스톤 수식에 따른 활성 / 억제
○ 오직 리신 아미노산만 mono-, di-, tri-methylation을 할 수 있음
○ 그 결과 전사 활성 / 억제가 실제로는 단순하지 않음
| Type | Histone | ||||||
| H3K4 | H3K9 | H3K14 | H3K27 | H3K79 | H4K20 | H2BK5 | |
| mono- methylation |
activation | activation | activation | activation | activation | activation | |
| di- methylation |
repression | repression | activation | ||||
| tri- methylation |
activation | repression | repression | activation, repression |
repression | ||
| acetylation | activation | activation | activation | ||||
Table. 2. 히스톤 수식에 따른 활성 / 억제
예를 들어, H3K27은 H3 히스톤의 27번째 K 아미노산 (리신, Lys)을 의미함
⑤ chromatin remodeling protein
○ remodeling protein a : ATP 사용
○ remodeling protein b : ATP 미사용
⑦ RNA 간섭(RNAi, RNA interference) : RNA silencing이라고도 함
○ 종류 1. miRNA (마이크로 RNA, micro RNA) : 핵과 세포질을 거쳐 가공된 작은 단일가닥 RNA
○ 개요
○ 시작이 단일가닥인 경우
○ small non-coding RNA (약 22 nucleotide)
○ RNA pol Ⅱ에 의해 전사됨
○ endonuclease에 의해 생성됨
○ well conserved
○ 사람에게 약 2000 종류 이상의 miRNA가 존재함
○ simple ternary interaction과 그것의 알려진 determinant로는 오직 10-20%의 miRNA 타겟팅만 설명 가능
○ 사람의 protein-coding gene 중 60% 이상이 miRNA의 direct target임
○ 1st. gene → pri-miRNA : RNA pol Ⅱ가 관여
○ 2nd. pri-miRNA → pre-miRNA : Drosha, Dgcr8, Pasha가 관여. pre-miRNA는 헤어핀 구조
○ 3rd. pre-miRNA가 핵외로 방출 : RNA-GTP, Exportin-5가 관여
○ 4th. pre-miRNA → miRNA/miRNA* duplex : Dicer가 관여. miRNA/miRNA는 dsRNA
○ miRNA/miRNA* duplex는 20 bp 정도
○ 5th. miRNA/miRNA* duplex → mature miRNA : Ago, RISC가 관여. miRNA는 ssRNA
○ Ago(Argonaute)
○ RISC(RNA-induced silencing complex)
○ 6th. mature miRNA가 mRNA와 결합 : Ago가 관여
○ miRNA와 mRNA가 완전 상보적인 경우 : mRNA 분해
○ miRNA와 mRNA가 부분 상보적인 경우 : mRNA 번역 중지
○ miRNA targeting
○ CST(canonical site type) : incomplete sequence
○ 6mer site : NNNNNN-5'
○ 7mer-A1 site : NNNNNNA-5'
○ 7mer-m8 site : NNNNNNN-5'
○ 8mer site : NNNNNNNA-5'
○ NST(noncanonical site type)
○ offset 6mer : 6mer site, 7mer-A1 site, 7mer-m8 site, 8mer site
○ centered site
○ pivot pairing site
○ single mismatch site
○ AGO CLIPSeq-based analysis
○ MIRZA site
○ miRNA targeting 인헨서 : Pumilio, PCBP2, FUS, PTBP1
○ miRNA targeting 서프레서 : Dnd1, RBM38, HuR, IGF2BP1, PTBP1
○ 종류 2. siRNA(소형간섭 RNA, small interfering RNA, short interfering RNA)
○ 1998년 처음으로 소개됨
○ miRNA와 거의 유사하나 시작이 이중가닥인 경우
○ small non-coding RNA라고도 함 : 약 20-27 nucleotide 정도로 인위적으로 합성
○ siRNA : 바이러스 RNA 혹은 실험 목적의 도입 RNA. pre-miRNA(shRNA) 혹은 dsRNA를 지칭
○ blood half life : 4분. 벡터가 있으면 수 시간 단위로 증가할 수 있음
○ 1st. pre-miRNA 형태로 도입된 경우 : pre-miRNA → miRNA/miRNA* duplex. Dicer가 관여
○ 2nd. miRNA/miRNA* duplex → miRNA : Ago, RISC가 관여
○ 3rd. miRNA가 mRNA와 결합 : Ago가 관여
○ 4th. 어떤 유전자든지 발현을 억제할 수 있음
○ 종류 3. lncRNA : long non-coding RNA
○ 200 nucleotide 이상
○ RNA pol Ⅱ에 의해 전사됨
○ 대부분 5'-capping, 부분적인 polyadenylation, splicing이 돼 있음
○ poorly conserved
○ 사람에게 약 30,000 종류 이상의 lncRNA가 존재함
○ 종류 4. sciRNA : small circular interfering RNA
○ 종류 5. ASO(antisense oligonucleotide)
○ 1978년 처음으로 소개됨
○ 기능 1. RNaseH recruitment → target mRNA degradation
○ 기능 2. splicing modification → exon inclusion / exclusion
○ 기능 3. miRNA targeting → miRNA sequestration
○ miRNA와 siRNA의 비교
○ miRNA는 hairpin 구조가 있고 siRNA는 hairpin 구조가 없음
○ 도메인별 RNA 간섭
○ 동물 : RNAi가 있음
○ 식물 : PTGS 현상이 있음
○ 원핵생물, 고세균 : RNA 간섭 현상이 없음. 대신 제한효소가 있음
5. 번역 [목차]
⑴ tRNA
① tRNA : 20종류, 안티코돈으로 mRNA의 코돈에 결합
○ 약 80 뉴클레오티드로 구성
② 아미노아실 tRNA 합성효소 : 20종류
○ 특정 아미노산을 해당 tRNA에 결합시켜 활성 아미노산 (aa-tRNA) 생성
○ 아미노아실 tRNA 합성효소가 tRNA 부위 중 주요하게 인식하는 부위
○ tRNA anticodon loop (mRNA 측 요소) : 정확한 tRNA를 선택하기 위해 tRNA의 안티코돈이 있는 loop를 인식
○ 3′ acceptor stem base (아미노산 측 요소) : 특정 tRNA가 특정 아미노산과 결합하는, tRNA의 3' 말단 부위
○ 아미노산은 tRNA의 3‘ 말단에 결합 (ATP 사용) : 3'-CCA가 아미노산 결합자리
○ 3' 말단에 있는 5탄당의 3'-OH기와 아미노산의 -COOH의 탈수축합반응
○ 단계 1. -COO- → -CO(AMP) : ATP가 분해되는 에너지를 이용하여 아미노산의 카르복실기 말단이 AMP와 결합
○ 단계 2. -CO(AMP) → -CO(tRNA) (효소 : 아미노아실 tRNA 합성효소)
○ 이중체 기전(double sieve model) : 아미노아실 tRNA 합성효소가 특정 아미노산을 받아들이는 기전
○ 첫 번째 체 : 활성 부위(activation site)라고도 함. 이소류신보다 큰 아미노산(예 : 페닐알라닌)은 배제됨. 크기 및 구조에 특이적으로 결합하는 효소가 관여함
○ 두 번째 체 : 편집 부위(editing site)라고도 함. 이소류신보다 작은 아미노산(예 : 알라닌)은 배제됨. 아미노산-AMP 중에서 정확한 아미노산만이 tRNA에 결합되도록 보장함
○ 아미노산과 안티코돈이 맞지 않을 경우 교정 가능하나 이후의 번역 단계에서는 교정 불가
○ L형 아미노산만 사용. (참고) 생체 내 포도당은 D형 포도당만 존재
③ tRNA의 1차 전사체는 전사 후 변형과정을 거쳐 성숙 tRNA가 됨
○ 성숙 tRNA : 약 80 ~ 90개의 뉴클레오티드로 구성된 단일가닥 RNA
○ tRNA의 특이한 자리에 있는 염기 몇 개는 화학적으로 변형(methylguanin, ribothymidine, dihydrouridine 등)되는데, 이런 변형은 안정성 부여
○ tRNA의 3‘ 말단에 3개의 뉴클레오타이드(-CCA)를 첨가
○ 엑소뉴클레아제의 일종인 RNase D가 관여
④ 성숙한 tRNA는 3개의 고리를 가짐
○ 염기와 리보오스의 OH기 사이에서 수소결합이 일어나기 때문
○ 2차원적 모양은 평면적 클로버 잎 모양
○ 3차원적 모양은 L자형
○ 5' 말단이 더 짧음 (∵ tRNA의 3'-OH기에 아미노산이 결합하므로)
⑤ 단백질 합성기구에 의한 tRNA의 인식 특이성에 관한 실험
⑵ rRNA
① 진핵생물 : 네 가지 rRNA(5S rRNA, 5.8S rRNA, 18S rRNA, 28S rRNA)가 필요
○ 인에 있는 45S rDNA → 45S rRNA (by RNA pol Ⅰ) → 18S rRNA + 5.8S rRNA + 28S rRNA
○ 인에 있는 45S rDNA → 5S rRNA (by RNA pol Ⅲ)
② 원핵생물 : 세 가지 rRNA(5S rRNA, 16S rRNA, 23S rRNA)가 필요, 한 개의 RNA pol 존재
③ 리보솜의 조립
○ 세포질 내 리보솜에서 합성된 단백질이 핵공을 통해 인으로 전달
○ 단백질과 rRNA가 인에서 조립 → 2개의 리보솜 서브유닛
○ 세포질로 이동한 후 리보솜 작은 소단위체와 큰 소단위체가 조립 (번역 개시복합체)
○ 미생물은 시간당 약 100,000개의 리보솜을 생성
④ 진핵생물의 리보솜 : 80S
○ 60S 큰 소단위체 : 28S, 5.8S, 5S rRNA + 46개의 서로 다른 단백질. tRNA 결합부위 존재
○ A site (amino site)
○ P site (peptide site)
○ E site (exit site)
○ 40S 작은 소단위체 : 18S rRNA + 33개의 단백질. mRNA 결합부위
⑤ 원핵생물의 리보솜 : 약 55가지 단백질 포함, 70S(고세균도 70S 리보솜)
○ 50S 큰 소단위체 : 5S, 23S rRNA
○ 30S 작은 소단위체 : 16S rRNA
⑥ rRNA 가공 시 snoRNA가 관여
○ 박테리아 : rRNA 절단을 통한 rRNA 가공, RNase Ⅲ, RNase P, RNase E 관여
⑶ 1st. 개시(initiation) : 리보솜이 리보솜 결합 부위(ribosome binding site; RBS)에 결합
① 원핵생물의 개시
○ 5'-UTR : 5' 말단부터 개시코돈 바로 전까지의 비번역 부위
○ SD서열(Shine-Dalgarno sequence) : 5'-AGGAGGU-3'의 purine-rich sequence
○ Shine-Dalgarno 서열에 30S 소단위체(16S rRNA + 단백질)가 결합한 후 mRNA의 AUG가 나올 때까지 이동
○ 1970년대 중반에 E.coli에서 16S rRNA와 상호작용하는 서열로서 처음 발견됨
○ TSS(start codong)의 upstream 위치 : TSS로부터 50 bp 이내. 주로 5-10 이내로 TSS와 가깝게 위치
○ 개시 tRNA(formyl-Met-tRNA)가 mRNA의 개시 코돈을 인식한 뒤 P자리에 결합 (GTP 사용)
○ 개시인자(IF-1, IF-2, IF-3)들과 대단위체가 결합하여 번역 개시복합체 형성
② 진핵생물의 개시
○ mRNA의 5‘-cap 말단에 소단위체가 결합하면서 시작 : 18S rRNA가 결합
○ 5'-UTR : 5'-cap부터 개시코돈 바로 전까지의 비번역 부위
○ 개시코돈 바로 전에 코작 서열(Kozac sequence)이라는 공통 서열 존재
○ Marilyn Kozac이 최초로 발견함
○ 코작 서열 : 5'-A/GCCACC-3'
○ 코작 서열 바로 다음으로 5'-AUGG-3'과 같이 이어짐
○ 즉, 진핵생물의 5'-UTR은 코작 서열을 포함함
○ 리보솜이 5'-cap에서 5'-AUG-3'까지 이동할 때 ATP를 가수분해하는 helicase를 필요로 함
○ Met-tRNA가 mRNA의 개시 코돈을 인식한 뒤 P자리에 결합 (GTP 사용)
○ 개시인자들과 대단위체가 결합하여 번역 개시복합체 형성
⑷ 2nd. 신장(elongation)
① A site, P site, E site
○ A site : aa-tRNA가 번역 개시복합체에 들어가는 자리
○ P site : 펩티드 결합이 형성되어 아미노산 사슬이 길게 늘어지는 자리
○ E site : tRNA가 번역 개시복합체에서 나가는 자리
○ 5' - E - P - A - 3' 순. 헷갈리지 말자
② 신장인자 : EF-Tu, EF-Ts, EF-G
③ codon recognition : 상보적인 tRNA가 A site의 코돈 인식, 2 GTP 필요, EF-Tu와 EF-Ts 관여
④ peptide bond formation : P site의 펩티드 말단의 카르복실기가 A site 아미노산의 아미노기와 펩티드결합 형성
○ 아미노기의 질소가 카르복실기의 카르보닐 탄소를 친핵체 공격함
○ 대단위체의 펩티드 전이효소활성에 의해 이루어짐
○ 아미노산 합성방향 : N → C
⑤ ④ 과정으로 P site의 아미노산 사슬이 전부 A site의 tRNA에 매달리게 됨
⑥ translocation : 리보솜이 다음 코돈으로 이동 (P자리 → E 자리 방출, A자리 → P 자리). 1 GTP 필요. EF-G 관여
⑸ 3rd. 종결(termination)
① 종결코돈(UAA, UAG, UGA)이 A 부위에 위치하게 되면 종결인자들이 붙어 번역이 종결됨
○ mycoplasma 코드 (code 4) : 종결코돈은 UAA, UAG
○ ciliate 코드 (code 6) : 종결코돈은 UGA
② 방출인자(releasing factor; RF) : P자리에 있던 tRNA와 폴리펩타이드의 마지막 아미노산 사이의 결합을 가수분해
○ 방출인자는 재활용됨
○ RF-1 : UAA와 UAG 인식
○ RF-2 : UAA와 UGA 인식
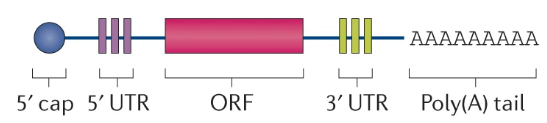
③ 3'-UTR : 종결코돈 바로 다음부터 3'-poly (A) tail 직전까지
⑹ 유전암호(genetic code)
① 트리플렛 코돈(triplet codon) : 개시코돈부터 시작하여 3개씩 코돈을 묶어서 번역의 기초가 되는 틀을 구성함
○ 그러한 틀을 번역틀이라고 함
② 중복 번역 불용 : 한 코돈은 단 한 번씩만 번역
③ 암호의 방향성 : mRNA를 기준으로 5'에서 3' 방향으로 코돈을 읽음 → 코돈표 해석
④ 개시코돈 : 번역의 시작은 항상 mRNA의 AUG(Met; 개시코돈)에서 시작
⑤ nonsense codon : 64개의 코돈 중 3개의 코돈은 종결코돈(UAA, UAG, UGA)이므로 아미노산 지정 ×
⑥ 워블 가설 (wobble hypothesis) : tRNA는 이론상 61개이나 실제로는 약 45개 정도 존재. mRNA → tRNA
○ A 염기가 이노신산(I)으로 변하는 탈아미노 과정과 관련
○ 특정 코돈이 상보적이지 않은 tRNA와 결합을 형성하는 경우 존재
○ 세 번째 코돈의 서열을 워블 서열(wobble sequence)이라 하며, tRNA와의 결합을 wobble pairing이라고 함
○ 가설 : 안티코돈의 첫 번째 염기와 코돈의 세 번째 염기는 엄격하게 결합하지 않음
⑦ 축퇴 (degeneration) : 아미노산은 약 20개이므로 2 이상의 코돈(45개)에 대응, tRNA → 아미노산
○ 코돈 사용의 발생빈도(codon usage) : 같은 아미노산을 생산하더라도 tRNA에 따라 번역 속도가 상이
○ 풀 내의 tRNA의 양을 확인하고 적절한 코돈으로 대체하여 번역 속도를 높이는 연구도 있음
⑧ 명확성(unambiguity) : ⑥, ⑦에 의해 하나의 코돈은 하나의 아미노산과 대응, mRNA → 아미노산
○ 반례 : 하나의 코돈이 여러 개의 안티코돈과 대응되는 사례가 있음
⑨ universal codon
○ 유전암호는 생명의 역사 초기에 진화된 것
○ 최소한의 변화마저도 치명적일 수 있기 때문에 변화 없이 잘 유지 (frozen accident)
○ 일부 원생동물, 미토콘드리아에서 예외가 발견
⑩ overlapping gene
○ 바이러스는 유전체의 양이 적으므로 정보의 중첩, 즉 번역틀 위반이 관찰
○ 하나의 염기를 최대 6번 해석 : 각 번역틀 별 세 번과 sense 가닥인지 anti-sense 가닥인지의 두 번을 곱함
○ 하나의 돌연변이가 큰 영향을 줌
⑺ 유전코드의 해독 : 1968년 노벨 생리의학상
① 아미노산 : 20 종류
○ 4∏2 = 16 < 20 < 4∏3 = 64
○ 초기 과학자는 암호의 단위를 3 뉴클레오티드라고 추론
② 유전암호의 종류
○ 무의미한 암호 : 3개의 종결코돈, 아미노산 서열의 마침표 역할
○ 유의미한 암호 : 20 아미노산 지정
○ 개시코돈 : 메싸이오닌(AUG)
③ 유전암호의 해독
○ 시험관 내 번역체계를 이용
○ 원핵생물의 발현체계와 동일
○ AUG부터 시작하지 않아도 됨
○ 폴리뉴클레오티드 가인산 분해효소만 있으면 만들 수 있음
○ 방법 1. Marshall W. Nirenberg & J. H. Mattaei 실험(1961)
○ 인공합성 RNA와 무세포단백질합성계(in vitro translation system) 이용
○ UUU, AAA, CCC, GGG에 대한 아미노산 해독
○ 방법 2. H. Gobind Khorana 실험
○ 인공합성 RNA를 이용하여 추가적으로 유전암호의 판독
○ 예 : 5'-CACACACACAC-3', ATP : CTP = 1 : 1인 혼합물을 이용시 트레오닌 : 히스티딘 = 1 : 1
○ 방법 3. Marshal W. Nirenberg & Ochora 실험
○ UTP와 GTP를 3 : 1로 섞음
○ UUU 코돈이 나올 확률은 0.753 → 검출 비율을 통해 무슨 아미노산과 대응되는지 알 수 있음
○ 확률이 비슷한 두 아미노산은 어떤 코돈과 대응되는지 확실히 알 수 없음
○ 예 : 5'-CACACACACAC-3', ATP : CTP = 5 : 1인 혼합물을 이용시 리신 100, 트레오닌 26, 아스파라긴 24, 글루타민 24, 프롤린 7, 히스티딘 6
○ 방법 4. tRNA 혼성화 + 여과법 : 정확한 코돈표 작성 가능, 실제로도 방법 4가 사용됨
④ 유전암호표
⑻ 해독틀(reading frame; RF)
① 정의 : 아미노산 서열로 번역할 수 있는, 3염기씩 구분돼 있는 프레임
② 구성 : 개시코드(ATG; met)부터 시작하는 약 100여 개 아미노산
③ 열린 해독틀(open reading frame; ORF) : 어떤 코돈을 개시코돈, 종결코돈으로 보는지에 따라 생성되는 여러 해독틀
④ 해독틀은 종결코돈(3종류가 있음)을 포함하지 않음
⑤ mRNA 내 해독틀의 위치
6. 번역 후 단백질의 배달 [목차]
⑴ 개요
① 합성된 단백질에는 특정 아미노산 순서로 구성된 신호서열이 있어서 단백질이 세포의 특정 위치로 운반됨
② 2013년 노벨 생리의학상을 받은 분야
⑵ 세포내 구획
① 세포내 화학반응은 서로 상반된 것이 많아 세포내 구획 필요
② 특정한 반응과정을 공동으로 촉매하는 여러 효소들을 하나의 거대 단백질 복합체로 조립하는 방식(원핵, 진핵)
③ 하나의 막구조물 내에 구획화시키는 방식(진핵) : 막성세포소기관 존재 의의
④ 새로 합성된 단백질을 해당 막성세포소기관으로 이동시키는 방식(단백질 표적화)
⑶ 신호서열
① N 말단 부위에 존재하는 일반적으로 약 20개의 서열
② 소포체 신호서열 : 약 10개의 소수성 아미노산
○ 소수성인 이유 : 신호펩티드가 친수성이면 막을 통과하기 어렵기 때문
③ 핵 배치 신호서열 : 연속적(4개 이상)인 (+) 전하를 가지는 아미노산
④ 엽록체 배치 신호서열 : 두 개의 다른 신호서열이 관여함
⑤ 예 : Hsp90의 신호서열

Figure. 27. Hsp90의 신호서열
○ 모든 Hsp90 단백질은 10개의 아미노산으로 구성된 신호서열을 포함
○ Y (1), F (7), R (9)가 모든 종에서 보존돼 있음
○ 하단의 그래프에 각 아미노산 기호의 크기는 빈도에 비례함
○ 이론적으로 2번째 아미노산은 아무 아미노산이 될 수 있지만 Ser이 대부분 들어감
⑷ 유리 리보솜(자유 리보솜, free ribosom)
① 세포질에 떠다니면서 세포 내부에서 작용하는 단백질을 합성
② 특징
○ 일반적으로 단백질 배달 기작 ×
○ 유리 리보솜에 의해 생성된 단백질은 이황화결합이 없음
○ post-translation : 번역이 완료된 후 단백질이 이동
③ 예 1. 핵 단백질
○ 안 → 밖으로 핵공을 통해 단백질 출입 시 : nuclear export signal + exportin
○ 밖 → 안으로 핵공을 통해 단백질 출입 시 : nuclear localization signal (NLS) + importin
○ 단백질의 신호서열에 수용체 단백질이 결합 후 같이 운반되기 때문에 신호서열 및 3차 구조 보존
○ 핵공을 통해 단백질 출입 시 접힌(folding) 상태로 이동
○ importin, exportin의 원 위치로 위치시킬 때 GTP 사용
○ 효모 단백질의 약 45%가 classic NLS 패턴을 갖는 아미노산 서열을 가지고 있음
④ 예 2. 퍼옥시좀 단백질
○ 퍼옥신에 의해 3차 구조 보존, 신호서열 보존
⑤ 예 3. 미토콘드리아 및 엽록체 단백질
○ 단백질이 막을 이동시 3차 구조가 깨짐. 신호서열 절단
○ 엽록체로 배달되는 단백질은 신호서열이 두 개임
⑥ 예 4. 표재성 단백질
⑦ 예 5. 세포질 단백질
○ 세포골격 단백질 등
⑸ 결합 리보솜(bound ribosom)
① 예시
○ 막단백질
○ 외분비 단백질
○ 리소좀 단백질
○ 액포 단백질
② 단백질 배달 기작
○ 1st. 번역 개시
○ 2nd. co-translation : 번역 중인 아미노산 서열상에 있는 신호펩티드가 세포질에 있는 SRP와 결합
○ 3rd. 먼저 제거 후 이동 : 신호펩티다아제가 신호서열 제거
○ 4th. 먼저 제거 후 이동 : 이동복합체를 통해 소포체 내부로 유입
○ 5th. 번역 재개
7. 번역 후 단백질의 가공(post-translational modification, PTM) [목차]
⑴ 개요
① 목적 1. 활성 조절
○ PTM은 활성을 증가시킬 수 있음
○ PTM은 활성을 감소시킬 수 있음
○ PTM은 다양한 기능을 부여할 수 있음
② 목적 2. protein-protein interaction
○ PTM에 의해 수식되는 부위가 binding interface일 수 있음
③ 목적 3. subcellular interaction
○ PTM이 타겟팅 신호일 수 있음
○ PTM이 membrane anchor일 수 있음
④ 목적 4. 수명 조절
○ PTM이 protein degradation을 촉진할 수 있음
○ PTM이 protein scanvege를 촉진할 수 있음
⑵ 화학적 변형
① 당화(glycosylation) : 막단백질, 신호단백질 등의 대다수가 당단백질
○ 분류 1. 도메인별 당화 과정의 차이
○ 대장균 : glycosylation 과정이 없어서 glycoprotein이 없음
○ 효모 : glycoprotein이 만노오스(mannose)가 다량 붙는 형태로 나타남
○ 효모의 구조 : 세포질 - 세포막 - 키틴 - β 글루칸 - 만노오스
○ 동물세포 : glycoprotein이 sialic acid가 붙는 형태로 나타남
○ 분류 2. 세포소기관 내 당화 과정 : N 당화와 O 당화로 구분
○ 2-1. N 당화(N-linked glycosylation)
○ 주로 세포막 단백질을 대상으로 N-glycan이 결합하는 것
○ 장소 : 소포체에서 아스파라긴(Asn) (N)-결합
○ 여러 개의 당을 붙임
○ 알부민은 N 당화가 안 돼 있음
○ PNGage F : N 당화를 분해함
○ 2-2. O 당화(O-linked glycosylation)
○ 주로 핵 및 세포질 단백질을 대상으로 O-glycan이 결합하는 것
○ 장소 : 골지체에서 세린(Ser), 트레오닌(Thr) (O)-결합
○ 1개의 당을 붙임
○ 종류 1. O-N-acetylgalactosamine (O-GalNAc)
○ 종류 2. O-N-acetylglucosamine (O-GlcNAc)
○ N-acetylglucosamin의 생성
○ Glc
○ Glc-6-ⓟ
○ Fruc-6-ⓟ
○ GlcN-6-ⓟ
○ GlcNAc-6-ⓟ
○ GlcNAc-1-ⓟ
○ UDP-GlcNAc
○ 세린 및 트레오닌의 -OH기와 N-acetylglucosamin의 β-glycoside 결합을 특징으로 함
○ 종류 3. O-mannose (O-Man)
○ 종류 4. O-galactose (O-Gal)
○ 종류 5. O-fucose (O-Fuc)
○ 종류 6. O-glucose (O-Glc)
○ glycocapture method : N 당화가 일어나는 자리를 알 수 있음
○ 1st. glycan chain을 산화
○ 2nd. hydrazide resin을 공유결합으로 부착시킴
○ 3rd. PNGase F로 N 당화를 제거
○ 4th. 동시에 Asn이 Asp로 변함
○ 5th. LC/ESI-MS/MS로 확인하면 0.984 Da만큼 증가한 peak을 확인함
② 인산화(phosphorylation)
○ 신호전달에 중요하게 관여
○ 예 : serine → phosphorylated serine. ATP가 사용됨
○ 예 : threonine → phosphorylated threonine. ATP가 사용됨
○ 예 : tyrosine → phosphorylated tyrosine. ATP가 사용됨
③ 이황화결합 : 조면소포체에서 형성. PDI(protein disulfide isomerase)가 관여
④ 프레닐화(prenylation) : farnesylation, geranyl-geranylation 등
⑤ 유비퀴틴화(ubiquitination) : 아래 참고
⑥ 아세틸화(acetylation)
○ 예 : lysine → acetyl-lysine
⑦ 황화(sulfation)
| phosphorylation | sulfation |
| HPO3- (+79.9663 Da) | SO3- (+79.9568 Da) |
| Ser, Thr, Tyr | Tyr |
| loss of 98 Da (pST), 80 Da (pY) | loss of 80 Da (sY) |
| reversible by kinase and phosphatase | sulfotransferase |
| 70-80% of proteome | secreted or membrane proteins (all proteins : 487, mammalian : 155, human : 54) |
| intracellular signaling | extracellular signaling |
| triester (pKa1 = 12, pKa2 = 7.2, pKa3 = 2.1) |
monoester (pKa = 2.1) |
Table. 4. 인산화와 황화 비교
⑧ 기타
○ 메틸화(methylation)
○ 나이트로화(nitration)
○ 아민화(amidation)
○ 포르밀화(formylation)
○ 팔미토일화(palmitoylation)
○ 히드록실화(hydroxylation)
○ SUMOylation
○ lipid anchoring
⑶ 샤프롱(샤페론, chaperon)
① 번역된 단백질 전구체는 샤프롱의 도움으로 적절한 접힘구조를 형성하여 3차, 4차 구조를 가지게 됨
② 예 1. 부분적 분해
○ 예 : 신호서열의 절단, 엔도펩티다아제에 의한 단백질 활성화 등
○ 예 : 인슐린 - 아미노산이 절단돼야 인슐린 활성을 가짐
○ 조면소포체에서 prepro-insulin의 N-signal이 절단되어 pro-insulin이 됨
○ 골지체에서 pro-insulin이 절단
○ 인슐린과 C peptide가 외분비
○ 인슐린 및 C peptide는 임상학적 진단에 유용
○ 예 : 소장벽에 붙어 있는 엔테로펩티다아제
○ 트립시노겐 내 결합 일부를 잘라 트립신으로 변형
○ 트립신은 키모트립시노겐 내 결합 일부를 잘라 키모트립신으로 변형
③ 예 2. 칼넥신(calnexin) : 정교한 3차 구조. 낭포성 섬유증과 관련
④ 예 3. 열 충격 단백질(heat shock protein)
⑷ 자이모겐(zymogen)
① 불활성인 효소 전구체
② 번역 후 가공으로 활성을 획득
③ 대부분의 소화효소가 자이모겐 형태
⑸ 단백질 합성량 조절
① 예 1. 페리틴(ferritin) : 철 저장 단백질
○ 철 반응요소(IRE, iron response element)가 암호화 부위 상류에 존재
○ Fe 수준 ↑ : IRE-BP가 불활성 → 단백질 번역 개시 → 발현
○ Fe 수준 ↓ : IRE-BP가 활성 → mRNA RBS에 IRE-BP가 결합 → 단백질 번역 억제
○ (참고) Dps : 원핵생물에서 페리틴의 역할을 하는 것. E. coli에서 발견됨 (Almiron et al., 1992)
⑹ 수명 조절
① 목적
○ 단백질이 잘못된 구조를 가지거나 변성된 경우 체내 독성을 가질 수 있음
○ 체내 단백질의 활성을 조절하기 위해 정상 단백질이라도 분해하는 경우가 굉장히 많음
② 예 1. 유비퀴틴
○ 76개의 아미노산으로 이루어진 작은 단백질
○ 1st. 분해할 단백질 및 폴리펩티드에 샤페로닌(예 : hsp70)이 와서 유비퀴틴 부착
○ 2nd. 유비퀴틴으로 표지된 단백질은 프로테아좀에 의해 인식된 뒤 가수분해
③ 예 2. 리소좀과 mannose pathway
○ 표적 분자, 손상된 수용체, 손상된 세포소기관 소화
④ 예 3. 프로탁 기술(protac)
○ 개요
○ 정의 : 유비퀴틴-프로테아좀 시스템을 이용하여 기존에 억제되지 않았던 단백질까지도 분해할 수 있는 기술
○ 프로탁은 인위적으로 E3 ligase에 결합하는 화학물질과 분해될 단백질에 결합하는 화학물질을 인위적으로 결합시킨 것
○ 생리적 활성을 가진 약물을 찾다가 우연히 프로탁을 구성하는 분자접착제 종류들이 발견됨
○ E3 ubiquitin ligase
○ 분해될 단백질에 유비퀴틴을 달아줌
○ 600여 가지 종류가 있으나 2~3개 정도만 프로탁으로 활발히 이용되고 있음
○ 구성
○ E3 ubiquitin ligase에 결합하는 화합물
○ 분해하려고 하는 단백질에 결합하는 화합물
○ 두 개의 화합물을 서로 연결하는 링커
○ 예시
○ VHL(von Hippel-Lindau tumor suppressor) : 프로탁 기술에서 널리 이용됨
○ CRLs·Cullin-RING E3 ligase : 여러 종류의 단백질에 유비퀴틴을 결합시켜 분해를 유도
○ SD-36 : 암세포의 성장과 증식, 침투, 전이 등에 관여하는 전사인자인 STAT3을 선택적으로 분해하는 프로탁
○ 풀베스트란트(Fulvestrant) : 에스트로젠 수용체(ER)을 억제할 뿐만 아니라 소수성 잔기를 붙여 ER 단백질 분해를 유도
○ 탈리도마이드(thalidomide) : 대표적인 분자접착제. 탈리도마드는 CRBN과 결합하고, CRBN은 유비퀴틴을 타겟에 붙임
○ 레날리도마이드(lenalidomide), 포말리도마이드(pomalidomide) : 탈리도마이드 유도체이자 분자접착제
8. 저해제 [목차]
⑴ 중합 저해제
① acyclovir (acycloguanosine) : 항바이러스제. 바이러스의 티미딘 인산화효소(tk)와 반응하여 바이러스의 DNA 중합 저해
② araC (cytosine arbinoside, cytarabine) : 항암제
③ araA (adenine arabinoside, vidarabine) : 항바이러스제
④ Antinomycin D : groove에 결합하여 DNA 복제를 방지함. 진핵세포에도 작용할 수 있음
⑤ fluoroquinolones (ciprofloxacin) : DNA gyrase를 억제함
⑥ 카보플라틴(carboplatin) : DNA 합성을 억제하는 항암제
⑦ topoisomerase Ⅰ inhibitor : 다음 예시들은 type ⅠB topoisomerase를 억제함
○ irinotecan
○ topotecan
○ camptothecin (CPT-11: Camtosar)
○ diflomotecan
○ lamellarin D
⑧ topoisomerase Ⅱ inhibitor
○ etoposide
○ teniposide
○ doxorubicin
○ daunorubicin
○ mitoxantrone
○ amsacrine
○ ellipticines
○ aurintricarboxylic acid
○ HU-331
○ ofloxacin (제품명 : Tarivid)
○ levofloxacin (제품명 : Cravit)
⑵ 전사 저해제
① antinomycin D : 전사억제제
⑶ 번역 저해제
① 사이클로헥시마이드(cycloheximide) : 진핵세포의 번역 억제제
② 세균의 번역에 작용하는 억제제 : 항생제로 작용할 수 있음
○ Chloromycetin : 펩티드 결합 형성 억제
○ Erythromycin : mRNA 상에서 리보솜 이동 억제
○ Neomycin : tRNA와 mRNA 간의 상호작용 억제
○ Streptomycin : 번역 개시 억제
○ Tetracycline : 리보솜에 tRNA의 결합 억제
⑷ 역전사효소 억제제 (nucleoside reverse transcriptase inhibitor, NRTI)
① AZT (Zidovudine)
② Combivir (AZT + Epivir)
③ Emtriva (emtricitabine)
④ Epivir (Lamivudine)
⑤ Epzicom (Abacavir + Epivir)
⑥ Hivid (ddC)
⑦ Trizivir (Abacavir + AZT + Epivir)
⑧ Videx & Videx EC (ddI) : 2', 3'-dideoxynosine. 3'-OH를 제거함
⑨ Zerit (D4T)
⑩ Ziagen (Abacavir)
입력: 2015.07.02 17:56
수정: 2025.11.27 13:25
'▶ 자연과학 > ▷ 일반생물학' 카테고리의 다른 글
| 【생물학】 9강. DNA 테크놀로지 (0) | 2019.02.16 |
|---|---|
| 【생물학】 5-1강. 유전자의 수평적 전달 (0) | 2019.02.16 |
| 【생물학】 7강. 유전과 유전학 (4) | 2019.02.16 |
| 【생물학】 4-7강. 지질 합성 (0) | 2019.02.16 |
| 【생물학】 4-6강. 지질 분해 (0) | 2019.02.16 |
















최근댓글