9강. DNA 테크놀로지(DNA technology)
추천글 : 【생물학】 생물학 목차
1. DNA 재조합 [본문]
2. 유전자 도서관 [본문]
3. PCR [본문]
4. DNA 지문법 [본문]
5. 혼성화 [본문]
6. 유전자 결손 [본문]
7. 핵치환 [본문]
8. DNA-단백질 상호작용 연구 [본문]
9. 단백질-단백질 상호작용 연구 [본문]
10. 유전자 치료 [본문]
b. 웨스턴블롯팅 프로토콜
c. 로마노프 왕가와 법의학
1. DNA 재조합 : DNA 합성 [목차]
⑴ 1st. mRNA로부터 표적유전자 생성 (진핵생물 유래 DNA의 경우)
① mRNA를 역전사하여 cDNA(complementary DNA to mRNA)로 합성
② RT-PCR : mRNA 용액에 DNA 프라이머를 첨가하고 역전사 효소(reverse transcriptase)를 1회 처리
○ oligo dT 프라이머 : 진핵생물 유래 mRNA에는 꼭 있는 poly A를 주형으로 완전한 cDNA를 합성할 수 있음
○ random 프라이머 (주로 hexomer) : 다양한 cDNA 풀을 형성할 수 있음
③ RT-PCR : 일반 PCR처럼 cDNA의 양을 증폭시킴
⑵ 2nd. 재조합 플라스미드
① 제한효소(restriction enzyme) : 엔도뉴클레아제(endonuclease)의 일종. DNA를 자르는 가위
○ 세균에서 외래 DNA를 공격하기 위한 목적
○ 공격받을 수 있는 자신의 DNA는 메틸화시켜서 보호
○ 평활 말단(blunt end) : 제한효소에 잘린 뒤에 다시 결합할 수 없는 말단
○ 점착성 말단(sticky end) : 제한효소에 잘린 뒤에 다시 상보적 수소결합을 형성할 수 있는 말단
○ DNA 재조합시 점착성 말단을 형성하는 제한효소를 사용
○ 회문구조(palindrome) 인식 : 5'에서 3' 순으로 읽으면 sense 가닥과 anti-sense가닥의 문장이 동일한 경우
○ TATCGTACGAAC → TATC + GTACGAAC
○ ATAGCATGCTTG → ATAGCATG + CTTG
○ 위의 예는 점착성 말단의 예
○ 제한효소 예
○ AgeⅠ : 5'-A▼CCGGT-3'
○ AluⅠ : 5'-AG▼CT-3'
○ BamHⅠ : 5'-G▼GATCC-3'
○ BglⅡ : 5'-A▼GATCT-3'
○ DpnⅠ : 5'-G metA ▼TC-3'
○ DpnⅡ : 5'-▼GATC-3'
○ EcoRⅠ : 5'-G▼AATTC-3'
○ FspEⅠ : 5'-CmC(N)12▼-3'
○ HindⅢ : 5'-A▼AGCTT-3'
○ HpaⅡ : 5'-C▼CGG-3'은 절단, 5'-CmC▼GG-3'은 절단하지 못함, 단 mC는 메틸화된 시토신
○ LpnPⅠ : 5'-CmCDG(N)10▼-3'
○ McrBC : 5'-PumC(N40-3000)PumC-3'
○ MspⅠ : 5'-C▼CGG-3', 5'-C▼mCGG-3', 단 mC는 메틸화된 시토신
○ MspJⅠ : 5'-mCNNR(n)9▼-3'
○ MSTⅡ : 5'-CCTN▼AGG-3', 단 N은 n개의 회문구조의 염기서열
○ NcoⅠ : 5'-C▼CATGG-3'
○ PstⅠ : 5'-CTGCA▼G-3'
○ SalⅠ: 5'-G▼TCGAC-3'
○ Sau3AⅠ : 5'-▼GATC-3'
○ SmaⅠ : 5'-GGG▼CCC-3', 평활 말단 형성
○ BamHⅠ에 의한 재조합 서열은 Sau3AⅠ에 의해서도 절단됨, 반대는 특정 경우만 성립
○ BglⅡ에 의한 절편과 BamHⅠ에 의한 절편은 서로 결합하며 제한효소에 의해 다시 잘리지 않음
○ 선형 DNA가 제한효소로 한 번 잘리면 2조각이 되는 반면, 환형 DNA는 제한효소로 한 번 잘리면 1조각이 됨
○ 제한지도 작성법(restriction mapping)
② 2nd - 1st. 표적 DNA와 플라스미드 DNA를 같은 제한효소로 잘라 같은 점착성 말단 형성
○ 2nd - 1st - 1st. 한 개의 제한효소만 처리 시 자가연결(self-ligation)을 막기 위해 alkiline phospatase(예 : CIP(calf intestinal phosphatase)) 처리
○ 2nd - 1st - 2nd. 2종류 이상의 제한효소로 처리하여 표적 DNA에 결합하는 플라스미드 DNA에 방향성 부여
③ 2nd - 2nd. 잘려진 유전자와 플라스미드를 한 시험관에 투입 → 상보적인 점착성 말단이 서로 결합
④ 2nd - 3rd. DNA 연결효소로 연결시켜 재조합 플라스미드 생성
⑶ 3rd. 형질전환 : 재조합 유전자를 숙주 세포(박테리아)에 넣어 형질전환시킴
① 형질전환 과정
○ 3rd - 1st. CaCl2 : Ca2+는 인지질에 결합하여 세포막 안정화
○ 3rd - 2nd. 열충격 42 ℃ : 일시적으로 세포막에 천공이 생겨 재조합 플라스미드를 숙주 내 삽입
② 숙주(host) : 대장균, 효모, 곤충세포 등
○ 재조합 유전자의 산물을 얻기 위해서는 대사 기능이 있는 숙주 세포에 넣어주어야 함
○ 조건 1. 1세대 기간이 짧을 것
○ 조건 2. 배양이 용이하고 값싼 배지에서 잘 자랄 것
○ 조건 3. 병원성이 없을 것
○ 조건 4. 항생제 비내성, 선별 표식 목적
③ 벡터(vector) : 재조합 DNA를 운반 및 복제하는 플라스미드 또는 박테리오파지 DNA
④ 클로닝 벡터(cloning vector) : 유전자 복제를 목적으로 하는 벡터
○ 4대 조건 : 복제원점(ori), 프로모터, 제한효소인식서열, 선별표식 마커(항생제 저항성 유전자)
○ 부가조건 : 작은 크기, copy number ↑
○ 클로닝 부위(cloning site)
⑤ 발현벡터 : 발현체계가 다른 숙주에 외래 유전자를 발현시키기 위한 벡터
○ 원핵세포 숙주 시 조건 : 프로모터, SD 서열, 70S 리보솜 결합자리, 전사 종결자, 클로닝 유전자는 cDNA
○ 3차 구조(예 : 이황화결합) 가공 단백질도 필요
○ 진핵세포 숙주 시 조건 : 프로모터, poly A 서열
○ 예 : lac 오페론의 촉진유전자 다음에 프로모터를 붙여준 경우
⑥ 벡터 예시
○ 예 1. pBR322 플라스미드
○ 4361 bp, ori 존재
○ EcoRI, BamHI, HindIII 등의 제한효소 인식서열을 가지고 있음
○ amp r : 플라스미드 pBR322에 있는 암페실린 내성 유전자, PstⅠ 인식
○ tet r : 플라스미드 pBR322에 있는 테트라사이클린 내성 유전자, HindⅢ, BamHⅠ, SalⅠ 인식
○ 재조합 균주 : tet 비저항성 amp 저항성 균주를 선택
○ 예 2. pUC19 플라스미드
○ 2686 bp, ori 존재
○ ApaLI 등의 제한효소 인식서열을 가지고 있음
○ lac Z : X-gal을 기질로 하여 푸른색 생성물을 생성
○ amp r
○ 예 3. Ti 플라스미드
○ Agrobacterium tumefaciens(Rhizobium radiobacter, 근두암종균) : 식물 근두암종(양성종양)의 병원균

Figure. 1. 당근세포에 부착하고 있는 근두암종균
○ Ti 플라스미드 : 근두암종균의 플라스미드, T-DNA 포함
○ T-DNA : 20 kb DNA, 오파인과 옥신(auxin)·시토키닌(cytokinin)(근두암종 형성) 합성 효소를 암호화
○ 오파인(opine) : 근두암종균의 영양분 제공원
○ 경과 : 근두암종균 식물 감염 → vir (virulance DNA, 25 bp)가 T-DNA 양단을 잘라 식물 핵염색체에 무작위 삽입
○ DNA 재조합 : 표적 DNA를 T-DNA 영역에 삽입, 식물에 상처를 낸 뒤 재조합 근두암종균을 감염
○ 재조합 근두암종균은 옥신, 시토키닌을 합성하지 않아 근두암종을 형성하지 않음
○ 예 4. pBHA
○ 예 5. pBIC-A
○ 예 6. 유전자 도서관 벡터 : BAC 도서관, YAC 도서관
○ 예 7. 박테리아 플라스미드 : F 플라스미드. R 플라스미드 (항생제 저항성 유전자 삽입). 대략 1 ~ 10 kb 정도
○ 예 8. 바이러스 벡터
○ 세균감염, 증식, 용균, 방출, 절단 과정을 거치면서 세균 DNA 내에 유전정보가 삽입
○ λ 파지를 주로 이용하며 대략 25 kb 정도
○ 레트로바이러스를 이용하기도 함
○ 예 9. cosmid : 파지의 cos end 도입, in vitro packaging 방법으로 대략 50 kb 정도
○ 예 10. 베타락타미다아제 유전자는 베타-락탐 링을 만들어 페니실린 저항성을 부여
⑷ 4th. 선별(screening) : 재조합 유전자를 가진 세포 또는 개체군을 선별
① 항생제 저항성 유전자 (재조합 플라스미드 정상 삽입에 따른 선별)
○ 예 : 암피실린 저항성 유전자, 테트라사이클린 저항성 유전자
○ 1st. replica 형성 : 콜로니 전체를 배양한 뒤, 배양접시 전체를 거름종이에 묻힘
○ 2nd. 거름종이에 묻은 콜로니의 일부에 항생제를 첨가
○ 3rd. 항생제로 인해 죽은 콜로니는 형질전환되지 않았다는 의미
○ 4th. 거름종이 상의 항생제로 인해 죽지 않은 콜로니와 대응되는 콜로니 선별
② 항생제 저항성 유전자 (재조합 성공여부에 따른 선별)
○ 1st. replica 형성 : 콜로니 전체를 배양한 뒤, 배양접시 전체를 거름종이에 묻힘
○ 2nd. 거름종이에 묻은 콜로니의 일부에 항생제를 첨가
○ 3rd. 항생제로 인해 죽은 콜로니는 제한효소가 항생제 저항성 유전자를 잘랐다는 의미
○ 4th. 거름종이 상의 항생제로 인해 죽은 콜로니와 대응되는 콜로니 선별
③ X-gal (재조합 성공여부에 따른 선별)
○ 클로닝 유전자는 lac Z 유전자 내에 삽입
○ 1st. X-gal은 원래 흰색이지만, lac Z에 의해 푸르게 변색된다.
○ 2nd. lac Z 함유 플라스미드를 넣어준 어떤 콜로니에서 X-gal이 흰색이면 형질전환이 됐음을 의미
④ 보고자 유전자(reporter gene)
○ 숙주세포에서 발현되지 않은 것을 이용
○ 예 : eGFP, td tomato
⑤ 서던 블로팅
⑸ 5th. 클로닝 증폭 : 대량 배양(fermentation) 및 정제과정(purification)으로 산물을 다량 수득
① 대량 배양시 항생제 내성 플라스미드와 비선별 목적으로 항생제를 쓰는 이유
○ 1st. 균주 내에 들어있는 플라스미드가 균주가 분열할 때마다 딸세포에 무작위로 들어감
○ 2nd. 분열이 진행되다 보면 어떤 세포는 플라스미드가 하나도 없게 됨
○ 3rd. 플라스미드가 있는 세포는 플라스미드를 만드는데 많은 에너지를 사용하므로 플라스미드가 없는 세포와 생장속도가 차이 남
○ 4th. 궁극적으로는 플라스미드가 없는 세포가 더 많아짐
② 봉입체(inclusion body)
○ 형질전환 시 숙주가 원래 가지고 있던 유전자가 아니므로 단백질이 만들어질 때 제대로 folding 되지 않고 뭉치는 것
⑹ 예시
① 호르몬 및 생리활성 물질 : 생장 호르몬, 인슐린, 세크레틴 등
② 치료 및 진단 시약 : B형 간염 백신, HIV 감염 진단, 인터페론, 네오엔돌핀, 진통제, 혈액 응고인자 등
③ 농업 : 황금쌀, 제초제 내성 작물, 해충 내성 작물
④ Ti 플라스미드 형질 전환 신기능성 작물 : 제초제 내성, 항 탄저병 작물(씨감자 등)
⑤ 재조합 균주 개발 전략
○ feedback / antibiotics resistant mutant
○ 영양요구성 돌연변이주(auxotrophic mutant)
○ 과발현 돌연변이주(overexpression mutant)
2. 유전자 도서관(DNA library, 인공염색체) : DNA 저장 [목차]
⑴ 유전자 도서관용 벡터
① BAC(Bacterial Artificial Chromosome)
○ 장점 : 교차가 일어나지 않음
○ 단점 : 원핵생물의 발현체계를 맞춰야 함
○ 인간 유전체 프로젝트에서 사용됨
○ 현재에도 사용되고 있음
② YAC(Yeast Artificial Chromosome)(pYAC3)
○ 인공 염색체 into yeast (~ 1 Mb), ARS 보유, 동원체와 텔로미어 사이에 DNA 삽입
○ 장점 : 진핵생물을 이용하기 때문에 인간 단백질을 만들기 용이
○ 단점 : 제1감수분열 전기에서 발생하는 교차
③ 바이러스 유전체 도서관
○ 단점 : 용량이 작음, 번식을 제어하기 어려움
⑵ 필요한 DNA 요소
① 동원체(centromere) : 세포분열 시 방추사에 붙어 각 딸 세포에 염색체를 적절하게 분배하도록 하게 하는 GC-rich 서열
② 텔로미어(telomere) : 염색체의 끝에 있어 세포분열로 인해 염색체가 짧아지는 것을 대비하기 위한 염기서열 부분
③ 복제원점(origin of replication) : DNA 복제가 개시되는 부위
④ 선택마커(selection marker) : 인공염색체가 세포 내에 제대로 삽입됐는지를 확인하기 위한 염기서열 부분
⑤ 기타 : 프로모터, 유전자 발현 조절 기구
⑶ 종류
① gDNA library
○ 1st. 페놀을 처리하여 히스톤을 침전시킴
○ 2nd. 핵산은 유전체 DNA 상층액에 존재함
○ 3rd. 상층액 분리 → Tri chloroacetic acid → 핵산 응집체
○ 4th. gDNA를 제한 효소로 절단한 후 모두 클로닝 → 벡터(BAC)에 삽입
○ 5th. 선별을 통해 해당 대장균 증식
○ 6th. 인트론 연구 시 사용
○ Triton X-100은 양친매성으로 DNA 구조를 변성시키지 않음
○ SDS-PAGE는 DNA 구조를 변성시킴
② cDNA library
○ 1st. 친화성 크로마토그래피 : 올리고 dT(poly T)를 비즈에 부착
○ 2nd. reverse transcriptase로 분리한 mRNA를 역전사시킴
○ 3rd. 저농도의 RNA 분해효소를 처리함 : 프라이머로 작용
○ 4th. DNA pol I를 처리
○ 5th. ligase를 처리
○ 6th. 제한효소를 처리
○ 7th. 벡터에 삽입
○ 8th. 클로닝 증폭
○ 9th. 유전자의 발현을 연구 시에 사용
③ 도서관 검색(library screening)
○ 도서관 클론들을 필터에 옮기고 탐침자와 활성화시키면 해당 탐침자와 동일한 서열을 가진 클론들의 위치 파악 가능
3. PCR (중합효소 연쇄반응, polymerase chain reaction) : DNA 증폭 [목차]
⑴ 개요
① 정의 : DNA 복제 원리를 응용한 DNA 증폭 기술
② 1983년 Kerry Mullis에 의해 개발됨 : 그는 1993년 노벨 화학상을 수상
⑵ 시료 : DNA 채취 시료, DNA 프라이머(2 종류), dNTP, Taq 중합효소, Buffer(pH 안정화), MgCl2(DNA 안정화)
① DNA 프라이머
○ 조건 1. GC content > 50%
○ 예 : 18개 중 10개(= 18/2 + 1)가 G 또는 C
○ 예 : 20개 중 11개(= 20/2 + 1)가 G 또는 C
○ 조건 2. 5' 말단은 AT로 끝나고 3' 말단은 GC로 끝남
○ 이유 : self-ligation으로 인해 hair pin이 생기는 것을 막기 위함
○ 예 : 5'-ATGCCTATGCG-3'
○ 예 : 5'-ATGCAGGCTAAT-3'은 self-ligation을 일으킬 수 있음
○ 조건 3. 3' 말단에서 2 ~ 3개의 G, C가 겹치게 함
○ 이유 : A=T 결합은 이중결합이고 G≡C 결합은 3중 결합이기 때문에 G, C가 겹치면 더욱 결합이 잘 됨
○ 예 : 5'-ATGCCTATGCG-3'
② dNTP
○ NTP는 RNA 중합의 재료임을 유의
○ colony PCR에서 DNA template 대신 대장균 colony를 사용할 수 있는 이유 : 열을 가하는 denaturation 때문
○ 즉, 대장균 colony가 denaturation 하에 조각남으로써 DNA 중합을 위한 재료를 제공함
③ Taq 중합효소
○ Taq 중합효소는 고열의 온천수에 서식하는 고세균 Thermus aquaticus로부터 얻어짐
○ 최초의 발견은 1960년대 옐로스톤 국립공원에서 이루어짐
○ Taq 중합효소 55℃ 최적의 중합반응, 92 ~ 95 ℃ 열변성
○ Taq 중합효소는 3' 말단까지 중합을 한 뒤에도 추가적으로 A를 첨가
○ 주형을 필요로 하지 않으므로 평활말단의 DNA의 재조합에 응용할 수 있음
○ Taq 중합효소는 3' → 5' proofreading 능력이 없어서 에러율이 1/104
○ 보통 DNA 중합효소는 복제 착오율이 1/107
④ MgCl2
○ DNA는 인산골격의 음이온 반발이 존재하므로 불안정한 구조 → Na+나 Mg2+가 DNA를 안정화할 수 있음
⑤ IPP(inorganic pyrophosphatase)
○ NTP들의 결합을 증진시킴
⑶ 단계
① 1st. DNA 변성(denaturing) : DNA 두 가닥을 분리하기 위해 가열하는 단계. 94 ℃. 30 ~ 60 초
② 2nd. 프라이머 부착(annealing) : 혼합물이 식음에 따라 프라이머들이 DNA 주형에 결합. 50 ~ 55 ℃. 1 분
③ 3rd. DNA 중합(elongation) : 중합효소는 프라이머에서 합성을 개시. 72 ℃. 1000 bp 당 1분 정도의 시간
④ 4th. ① ~ ③ 과정을 반복하면 이론적으로 2의 지수적으로 DNA 채취 시료가 복제
⑤ 5th. DNA 연결효소(ligase) 작용(40 ℃) 및 안정화(4 ℃)
⑥ 6th. 몇 사이클 후 대장균 클로닝 증폭으로 전환 : 실제로는 인공 뉴클레오티드의 양이 부족해 훨씬 적은 DNA가 생산
⑷ Tm vs annealing 온도
① DNA melting curve : 온도에 따라 DNA denaturation이 일어나는 정도를 도식화한 그래프
○ 즉, 온도에 따른 선형 이중가닥 DNA의 변성 정도를 흡광도(A260)로 나타낸 것
○ A260 값 비교 : 이중가닥 DNA < 단일가닥 DNA < 절편화된 핵산
○ 이유 : 가리움이 적을수록 흡광도 값이 더 높음
② Tm : DNA의 50 %는 이중가닥을 유지하고, 50 %는 단일가닥으로 변성된 온도
○ 통상적으로 (A + T) × 2 + (G + C) × 4에 비례
○ Tm 값과 반복서열은 무관함
○ 동일한 서열의 dsRNA가 dsDNA보다 Tm 값이 15 ℃ 정도 더 높음 : RNA의 -OH기가 dsRNA를 안정화
③ annealing
○ 보통 annealing 온도는 Tm에서 5 ~ 10 ℃ 정도 낮게 설정
○ annealing이 너무 높으면 hybridization이 잘 안 일어나고, 너무 낮으면 mis-hybridization이 많아짐
○ 금속이온의 유무가 최적 annealing 온도에 영향을 줌
⑸ Ct (threshold cycle)
① 정의 : 역치값 이상이 될 때의 cycle
② Ct와 copy number는 음의 상관관계가 있음
⑹ 한계 : template DNA의 양 말단의 염기서열을 알아야 함
① 가정 : 만약 중간에 알려진 서열이 있고, template DNA의 양 말단이 blunt end이면, 한계 극복 가능
② 해결책 1. inverse-PCR
○ 1st. 주형 DNA로부터 DNA 연결효소를 통해 환형 DNA 생성
○ 2nd. 알려진 서열 내부를 자르는 제한효소로 선형 DNA로 생성
○ 3rd. 양 말단의 염기서열을 알고 있으므로 PCR 가능
③ 해결책 2. anchored-PCR
○ 1st. 새 프라이머를 주형 DNA와 DNA 연결효소로 연결하여 반쪽을 증폭
○ 2nd. 나머지 반쪽도 같은 과정을 진행
④ 해결책 3. oligonucleotide-directed mutagenesis
○ 경우 1. 몇 개의 염기만 mismatch가 되는 프라이머로 주형 DNA를 복제하는 방법
○ 경우 2. 중간에 헤어핀 구조가 형성되도록 염기서열을 추가한 프라이머로 주형 DNA를 복제하는 방법
○ 경우 3. 주형 DNA가 헤어핀 구조를 형성하도록 염기서열을 제거한 프라이머로 주형 DNA를 복제하는 방법
⑺ 종류
① RT-PCR : 역전사 효소를 사용하여 RNA에서 cDNA를 만드는 과정이 포함됨
② real time PCR(qPCR, quantitative PCR) : 증폭된 DNA의 양을 실시간으로 확인 가능
○ 처음에 주어진 염기서열 절편은 형광 reporter와 quencher 사이의 거리가 가까워 형광이 나타나지 않음
○ PCR 상에서 중합반응이 일어나면 형광 reporter와 quencher 사이의 거리가 멀어져 형광이 나타남
○ Taqman qPCR : 타겟 DNA에 결합하는 Taqman 프로브(fluorophore, quencher 포함)가 DNA 합성 중 분리되어 형광 생성
③ multiplex PCR : 한 PCR 장치 내에 여러 종류의 DNA 시료를 증폭하는 것
④ 대립유전자 특이 중합효소연쇄반응법(AS(allele-specific)-PCR)
4. DNA 지문법 : DNA 구분, 일란성 쌍생아가 아닌 어떤 두 사람도 유전적으로 동일하지 않다는 것 [목차]
⑴ 목적 : 친자 확인, 유전병 예측, 법의학적 범인 식별
⑵ 전기영동(electrophoresis) : 크기에 따른 물질 분리 및 양 추정 가능
① 정지상, 이동상
○ 정지상(압축용 겔, stacking gel)
○ 겔의 밀도가 높아 DNA 시료나 단백질이 느리게 이동
○ 겔 밀도가 높은 이유 : DNA 시료나 단백질의 크기에 상관없이 이동속도를 일정하게 하기 위함
○ 이동상(분리용 겔, running gel)
○ 겔의 밀도가 낮아 DNA 시료나 단백질이 빠르게 이동
○ 겔 밀도는 6-15 %
② 핵산 전기영동 : 아가로스 겔(agarose gel)
③ 단백질 전기영동 : 폴리아크릴아마이드 겔 + 비스아크릴아마이드 겔, SDS-PAGE, TEMED
○ 재료 1. 아크릴아마이드(acrylamide) : 젤에서 중합체를 형성
○ 재료 2. 리파 버퍼(ripa buffer) : 등장액. 세포를 용해시킬 때 사용하는 버퍼
○ 재료 3. 로딩 버퍼(loading buffer) 또는 Laemmil sample buffer : 이황화결합을 파괴하는 버퍼
○ SDS-PAGE를 포함하고 methanol을 포함하지 않음
○ SDS-PAGE : 더 효율적인 분리를 위해 사용. 변성겔에 사용
○ SDS-PAGE는 SDS(음전하 계면활성제)와 β-ME(β-mercaptoethanol)로 구성
○ SDS : 이온결합, 소수성 상호작용 등 제거. 단백질의 질량당 음전하 밀도를 일정하게 만들어줌
○ β-ME : 이황화결합 제거
○ SDS-PAGE가 아미노산마다 붙으면 음하전 간의 반발력으로 모든 단백질이 동일한 전하밀도를 가지는 2차 구조가 됨
○ (참고) DTT : 시스테인 간의 이황화결합을 파괴하는 첨가제
○ 재료 4. 트랜스퍼 버퍼(transfer buffer) : gel에서 membrane으로 transfer하는 데 사용
○ methanol을 포함하고 SDS-PAGE를 포함하지 않음
○ native-PAGE : 비변성겔에 사용
○ methanol : transfer하는 데 발생한 열을 cooling하는 역할을 수행함
○ 재료 5. 과산화황산암모늄(APS, ammonium persulfate) : 폴리아크릴아마이드 겔과 비스아크릴아마이드 겔의 교차결합을 형성하는 효소의 역할
○ 재료 6. TEMED(N,N,N',N'-tetramethylethylenediamine) : APS를 안정화하는 역할. 젤이 빨리 굳도록 함
④ 일반적으로 위가 음극(anode), 아래가 양극(cathode)이 되도록 함
⑤ 이동거리 = Δt × (a × log M + b), a < 0, M : molecular weight
○ SDS 등의 특수물질 처리시 분자량은 크기와 선형 비례하므로 이동상에서 저항을 많이 받아 이동거리가 작아짐
○ 분자량은 이동거리에 가장 큰 영향을 줌
○ 이동거리는 시간(Δt)에 비례
○ DNA ladder(size marker) : 크기에 따른 이동거리를 제시하여, 미지의 물질의 크기를 추측할 수 있음
○ 초나선 DNA는 전기영동에서 저항이 작음
⑥ 종류 1. 일반적 전기영동
○ 분자량이 작은 핵산·단백질이 더 많이 이동
○ 단백질 전기영동에서 SDS-PAGE에 의해 단백질은 (-)극에서 (+)극으로 이동
⑦ 종류 2. 등전점 전기영동 : 단백질의 고유한 값인 등전점을 이용한 전기영동
○ pH 구배가 있는 상태에서 전기영동하면 전체 전하의 합이 0이 되는 지점(등전점)까지 단백질 이동
○ 전기영동 후에 쿠마시블루나 질산은과 같은 단백질 염색약으로 염색

⑧ 종류 3. 2차원 전기영동
○ 오파렐(O'Farrel) 법이 일반적
○ x 축 : 요소 존재 하의 등전점 전기영동이나 미변성 조건 하의 전기영동에 의해 하전에 따라 분리
○ 즉, 등전점에 따라 분리
○ 요소 : 이황화결합을 제외한 모든 R-R 상호작용 제거
○ y 축 : SDS 존재 하의 전기영동에 의해 분자량에 따라 분리
○ 1,000 종류 이상의 단백질을 분리하는 것이 가능
○ 응용 1. x 축과 y 축을 동일 방법으로 분리하되, y 축 분리시 특정 물질을 첨가 → 대각선을 벗어나는 물질은 해당 물질과 상호작용한다는 증거
○ 응용 2. tRNA , 소형 RNA 분자의 경우 : 1차원 10 %, 2차원 20 % 폴리아크릴아마이드 겔 농도 → 각종 RNA를 효과적으로 분리
⑶ 지문법 1. RFLP (제한효소 조각 길이 다형성, restriction fragment length polymorphism) : 친자 확인, 유전병 분석
① 과정 : 유전자를 대상으로 함
○ 1st. 조직으로부터 DNA 분리
○ 2nd. PCR : DNA의 양을 증폭
○ 3rd. 제한효소를 사용하여 DNA를 조각으로 절단
○ 4th. 동일한 제한효소 인식 부위를 가지는 서로 다른 크기의 DNA 조각 생성
○ 5th. 전기영동 : 조각들을 크기 차이로 분리시켜 볼 수 있도록 함
② 원인 : SNP(단일 염기 다형성, single nucleotide polymorphism)
○ 정의 : 염기 하나가 점돌연변이에 의해 달라서 생기는 사람 간의 차이
○ 사람 기준 1000 bp당 1개씩의 SNP가 나타남
○ 유전자나 인트론 모두 SNP가 일어날 수 있음
○ 제한효소 인식부위에 SNP가 일어나면 RFLP가 일어날 수 있음
○ 돌연변이와의 차이 : 전체 인구의 1%가 안 되면 돌연변이, 그렇지 않으면 다형성(polymorphism)
○ SNP의 개수 : 2017년 기준 약 10000개
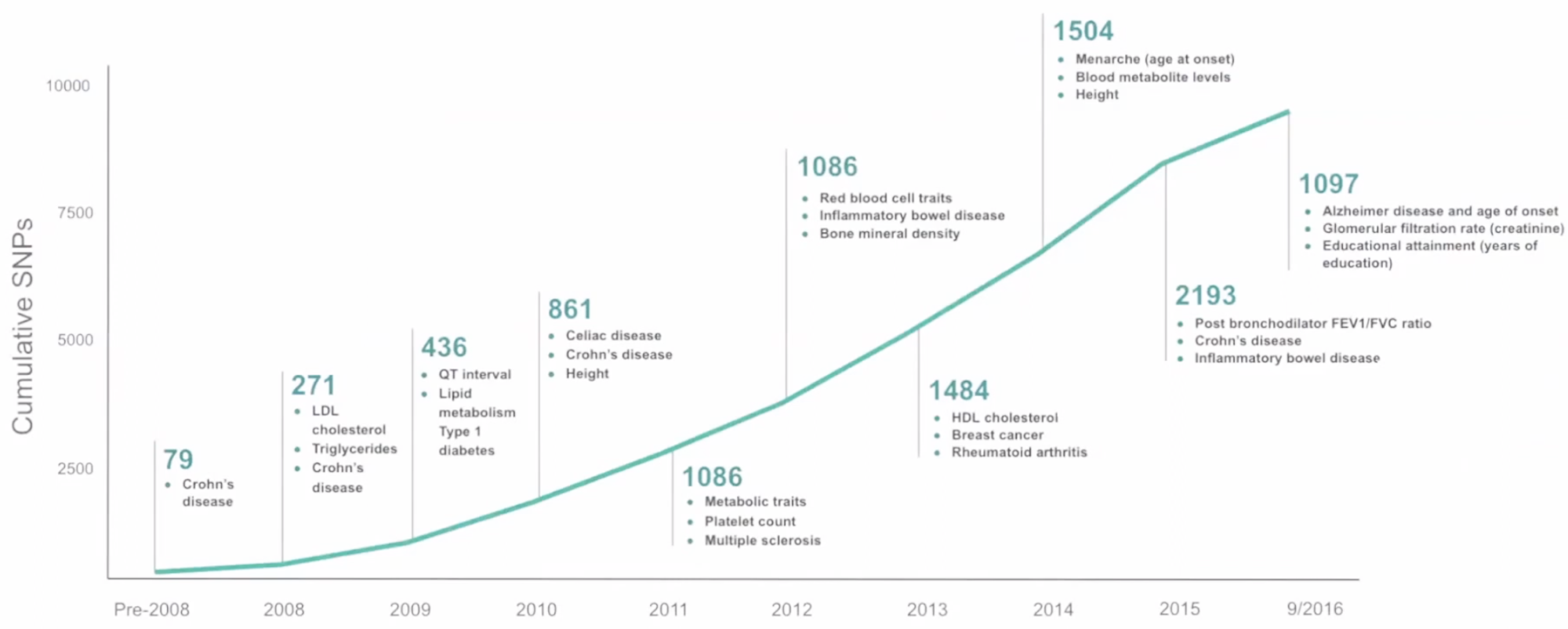
Figure. 3. SNP의 개수
③ 한계 : SNP가 하필 제한효소 인식부위에 일어난다고 할 수 없으므로 RFLP가 사람마다 차이가 크지 않음
○ 범인 식별 등에 활용하기 곤란
⑷ 지문법 2. 반복변수(tendom repeat) : 범인 식별. RFLP보다 강력한 유전자 지문. 반복서열을 대상으로 함
① 직렬반복변수(VNTR, variable number tandem repeat) : 15-100 번의 반복서열
○ 원리 : 개인 간의 반복서열의 반복수가 달라 다양한 크기의 토막이 만들어짐
○ 원인 : 상동염색체의 불균등한 교차
○ 1st. 전기영동으로 DNA 조각을 길이에 따라 분리 및 증폭
○ 2nd. 분리된 DNA 조각들을 여과지로 옮김
○ 3rd. 여과지로 옮긴 후 수소결합을 깨기 위해 화학약품(염기성)을 처리 → 한 가닥의 DNA
○ 4th. 방사선 물질로 표지된 VNTR 탐침을 가지고 VNTR 조각들의 위치를 비교
○ 5th. 여과지를 X-선 필름에 노출시키면 밴드가 나타남
② STR(Short Tendom Repeat) : 2-4 번의 반복서열
○ VNTR과 별반 다르지 않음
⑸ 지문법 3. 기타 지문법
① CNV(copy number variation)
② LOH(loss of heterozygosity)
③ genomic rearrangement
④ rare variant
5. 혼성화(hybridization) : DNA 존재여부 확인 [목차]
⑴ 서던 블로팅(Southern blotting) : DNA에 대한 핵산 탐침 혼성화
① 개요
○ RFLP의 일종
○ 1975년 Edward M. Southern에 의해 개발
② 1st. DNA 시료를 제한효소 처리 → PCR 증폭 → 전기영동
○ 전기영동 : 아가로스 겔 사용
○ 전기영동 대신 마스터 플레이트 사용 가능 : 재조합 플라스미드가 도입된 콜로니를 고체배지에 나열
③ 2nd. 겔을 0.5 M HCl 용액에서 진탕
○ HCl 처리에 의해 아가로스 겔에서 막으로의 DNA 전이가 더 빨라짐
④ 3rd. 겔을 1.5 M NaCl, 0.5 M NaOH 용액에서 진탕 (DNA 변성)
○ NaOH 용액 - 스펀지 - 겔 순으로 두어 DNA를 단일가닥으로 분리
○ 스펀지는 NaOH 용액을 빨아들여 겔에 제공하는 역할
○ ssDNA의 부분적인 이중결합 형성을 방지하기 위해 핵산 변성제를 추가로 첨가
○ 핵산 변성제 : 금속이온, 포름아마이드, 포름알데히드 등, 포름 시리즈는 DNA 결합력 약화
⑤ 4th. 겔을 1.5 M NaCl, 1 mM EDTA, 0.5 M Tris-HCl(pH 7.2) 용액에서 진탕
○ Trsi-HCl : 완충용액
⑥ 5th. 모세관 현상 + 블로팅
○ 5th - 1st. 겔 위에 양전하를 띠는 나일론 필터(nylon filter)와 종이 타월 뭉치를 올려놓음
○ 나일론 필터 대신 니트로셀룰로오스 막(nitrocellulose paper)을 사용할 수 있음
○ 5th - 2nd. 겔로 이동한 NaOH 용액은 단일가닥 DNA를 싣고 타월 뭉치까지 이동하여 흡수됨
○ 5th - 3rd. 음전하를 띠는 단일가닥 DNA는 나일론 막에 붙어 버림
⑦ 6th. autoclave : DNA와 결합한 나일론 막은 그 결합을 고정하기 위해 고압·고열처리
○ 막에 자외선을 비추어도 DNA가 막에 결합할 수 있음
○ 일반적으로 121 ℃에서 15분간 멸균을 하면 대부분의 미생물이 사멸
⑧ 7th. 블로킹(blocking) : blocking agent를 첨가하여 선명도를 향상시킴
○ 7th - 1st. 반복실험 혹은 대립유전자별 전기영동을 통해 유의미하지 않다고 여겨지는 DNA를 식별
○ 7th - 2nd. 해당 DNA들에 연어 정자(salmon sperm) DNA 절편을 첨가
○ 목적 : 8th의 혼성화 과정에서 탐침 DNA가 막에 비특이적 결합을 하는 것을 방지 (선명도 향상)
○ 참고로 막은 DNA 또는 단백질과 상호작용이 강해 블로킹 과정을 필요로 함
⑨ 8th. 혼성화
○ 8th - 1st. DNA가 결합한 나일론 막은 seal-a-meal bag에 담김
○ 8th - 2nd. 62 ℃에서 seal-a-meal bag에 방사성 원소가 표지된 DNA 탐침(probe)을 첨가
○ 표지된 탐침은 관심 유전자의 상보적 서열을 가지고 있음
○ 탐침 DNA : 100-500 bp, 단일가닥 DNA
○ 탐침 DNA는 일반적으로 방사성 동위원소인 P32를 표지
○ 탐침 DNA는 EtBr, SYBR Green 등의 염색약으로도 표지 가능
○ 8th - 3rd. 탐침은 상보적 DNA와 결합
○ 반복실험을 통해 유의미하지 않다고 여겨지는 다른 DNA는 블로킹을 통해 연어 정자 DNA로 덮여 있음
⑩ 9th. seal-a-meal bag 내에서 혼성화하지 않은 방사성 탐침을 제거
⑪ 10th. autoradiogram : 나일론 막에 방사선 조사
⑫ 11th. 방사성을 띠는 전기영동 상의 위치(혹은 콜로니 위치)를 식별
⑬ house keeping gene
○ 모든 세포에 있고 지속적으로 발현하고 있는 유전자
○ 서던 블로팅 때 대조군으로 사용
○ 예 : β-액틴(ACTB), 튜불린, GAPDH, B2M, RPL11, 18S rRNA (가장 신뢰성 높음)
⑭ 엄격성(stringency)
○ 엄격성을 낮추는 조건 : 탐지자가 결합하기 수월한 조건을 구성
○ 저온이나 고농도의 염 용액에서는 핵산 간의 혼성화가 수월하여 엄격성↓
⑵ 노던 블로팅(northern blotting) : RNA에 대한 핵산 탐침 혼성화
① 서던 블로팅과 굉장히 유사하나 다음과 같은 차이가 존재
② 차이 1. 시료의 추출
○ 서던 블로팅은 DNA 추출법을 이용
○ 노던 블로팅에서는 올리고 dT를 이용하는 친화성 크로마토그래피로 추출물에서 mRNA를 효과적으로 분리
③ 차이 2. 제한효소 사용 유무
○ 서던 블로팅은 길이가 긺므로 제한효소 사용해야 함
○ 노던 블로팅은 DNA가 아닌 RNA를 이용하므로 제한효소 사용할 수 없음
④ 차이 3. 모세관 현상 시 용액의 종류
○ 서던 블로팅은 단일 가닥 DNA로 변성시키기 위해 염기성 용액을 사용
○ 노던 블로팅에서 염기성 용액을 사용하면 RNA가 분해됨
○ 노던 블로팅은 RNA를 안정화시키기 위해 염 용액을 사용
⑤ 차이 4. 탐침의 종류
○ 서던 블로팅은 gDNA 탐침을 사용
○ 노던 블로팅은 cDNA 탐침을 사용
⑶ 웨스턴 블로팅(western blotting) : 단백질에 대한 항체 탐침 혼성화
① 핵산 블로팅과 차이점이 다수 존재
② 차이 1. 겔의 종류
○ 핵산 블로팅은 아가로스 겔을 이용
○ 웨스턴 블로팅은 폴리아크릴아마이드 겔(polyacrylamide gel), SDS-PAGE 등을 이용
③ 차이 2. 전기영동 전압 조건
○ 핵산 블로팅에서 핵산은 전기영동으로 이동을 잘하므로 저전압을 인가해 주어야 함
○ 웨스턴 블로팅에서 단백질은 전기영동으로 이동을 잘 하지 못하므로 고전압을 인가해 주어야 함
④ 차이 3. 블로팅 과정
○ 핵산 블로팅은 블로팅 과정에서 모세관 현상 이용
○ 웨스턴 블로팅은 블로팅 과정에서 전기장을 이용
⑤ 차이 4. 핵산 전처리
○ 핵산 블로팅은 연어 정자 DNA 단편을 이용
○ 웨스턴 블로팅은 카제인(casein), 스킴 밀크(skim milk), BSA(bovine serum albumin) 등을 이용
○ Tween : 계면활성제인 Tween-20 등이 사용됨
○ Uniqema라는 미국 회사에서 만든 상품명
○ poly(ethylene glycol)의 친수성 그룹과 탄화수소의 소수성 그룹이 섞여 있는 detergent
○ 용도 : 물과 기름을 섞이게 하는 용도, 세포의 막을 파괴하는 용도
○ 뒤에 있는 숫자는 20, 40, 60, 80 등이 있는데 이 값이 커질수록 탄소수가 늘어나 소수성이 커짐
○ BSA는 핵산 블로팅에도 사용할 수 있음
⑥ 차이 5. 탐침의 종류
○ 핵산 블로팅은 DNA 탐침을 이용
○ 웨스턴 블로팅은 항체 탐침을 이용
○ 1차 항체 : 표적 단백질과 특이적인 항체
○ 1차 항체의 특이성을 위하여 표적 동물과 다른 동물에서 추출한 효소를 사용해야 함
○ 2차 항체 : 1차 항체와 특이적인 항체. 일반적으로 발색 기질을 분해하는 효소와 결합
○ 2차 항체의 특이성을 위하여 표적 동물 및 1차 항체 유래 동물과 다른 동물에서 추출한 효소를 사용해야 함
○ 즉, 웨스턴 블로팅에서 1차 항체와 2차 항체는 다른 동물 기원
⑦ 차이 6. 이미징 방법
○ 핵산 블로팅은 EtBr + 자기방사법을 이용
○ 웨스턴 블로팅은 쿠마시블루 염색법을 이용
⑷ DNA chip (microarray)
① 특징
○ 다수의 유전자의 발현양상을 동시에 확인 가능
○ 한 칸에는 하나의 cDNA probe가 박혀 있음
○ cDNA DNA chip을 통한 조직 특이적 유전자 발현 확인
○ 마이크로어레이 데이터는 raw 데이터가 연속형 데이터인데 RNA-seq은 raw 데이터가 count data
○ RNA-seq은 마이크로어레이 데이터에 비해 발현이 너무 낮거나 높은 유전자에 대해서도 robust하게 신호를 얻을 수 있음
○ NGS 기술에 의해 사양기술이 되어가고 있는 추세
② 과정
○ 1st. 유리 슬라이드에 인간 유전자 cDNA 라이브러리를 점적하여 cDNA 칩을 제작
○ 2nd. cDNA 칩에 1% BSA 용액을 처리
○ BSA는 칩과 DNA의 비특이적 결합을 방지하여 오직 상보적인 DNA만 결합하도록 함
○ 3rd. 정상 조직, 비정상 조직(예 : 암)으로부터 mRNA X와 Y를 각각 준비
○ 4th. X와 Y에 각각 oligo-dT를 첨가
○ oligo-dT는 X와 Y에 있는 poly A 서열에 상보적으로 결합
○ 5th. RT-PCR : X에는 dNTP와 cy3(녹색 형광물질)-dTTP, Y에는 dNTP와 cy5(적색 형광물질)-dTTP를 각각 첨가한 후 역전사 반응을 수행하여 형광 표지된 cDNA 생성
○ 6th. X와 Y에 각각 0.1 N NaOH를 넣어 70 ℃에서 10분간 반응시킨 후 0.1 N HCl을 넣어 중화
○ 7th. X와 Y에서 합성된 cDNA를 정제한 뒤 동량으로 섞어 cDNA 칩과 혼성화
○ 8th. cDNA 칩을 완충용액으로 세척
○ 9th. 스캐닝(scanning) : cy3, cy5의 형광 강도를 측정하고 보정
○ 검정 : X와 Y가 둘 다 발현하지 않음
○ 초록 : X만 발현
○ 빨강 : Y만 발현
○ 노랑 : X와 Y 모두 발현
⑸ ISH(in situ hybridization)
① 개요
○ 특정 DNA 서열을 염색체와 혼성화시키는 실험 기법
○ 특정 염기서열의 위치뿐만 아니라 RNA의 위치를 알아내는 데도 사용
○ 염색체 이상을 신속하게 진단하지만 DNA 이상을 진단할 수는 없음
② 종류 1. FISH(fluorescence in situ hybridization)
○ 특정 염기서열에 상보적인 형광성 탐침으로 혼성화한 뒤 형광현미경으로 관찰
○ 1st. 슬라이드 상에 상피 조직 절편을 준비
○ 2nd. RNase를 처리하고 37 ℃에서 1시간 배양 후 씻어줌
○ DNA에 결합하는 probe는 RNA에 결합할 수도 있으므로 RNA를 제거해주는 과정이 필요함
○ 3rd. 배양액을 제거한 후 2% 포름알데히드 용액을 넣고 15분 동안 반응시킴
○ 세포를 고정시키는 과정
○ 4th. 포름알데히드 용액을 제거한 후, 0.2% Triton X-100 용액을 넣고 5분 동안 반응시킴
○ Triton X-100은 계면활성제임
○ 5th. Triton X-100 용액을 제거한 후, 2 M HCl을 넣고 펩신을 처리한 후 37 ℃에서 10분간 반응시킴
○ 펩신은 산성에서 작용하므로 pH 조성을 산성으로 바꿔줌
○ 펩신은 세포 내의 단백질을 제거해 추후 탐침의 침투를 용이하게 함
○ 6th. 세척
○ 7th. 동원체에 결합할 수 있는 probe를 준비, 단 이 probe의 dTTP에는 biotin이 부착돼 있음
○ 8th. 4th의 샘플과 5th의 probe를 혼성화
○ 9th. 세척
○ 10th. 형광물질이 레이블링 된 avidin을 충분히 처리한 후 세척
○ 11th. DAPI로 염색 : DAPI는 핵 염색
○ DAPI(4'-6-diamidino-2-phenylindole)
○ DNA 부홈의 AT-rich 부위에 강하게 결합하는 형광 염료
○ apoptosis marker로도 사용
○ DAPI는 세포막 투과성이 있어 살아있는 세포와 고정된 세포의 염색에 모두 사용 가능
○ DAPI와 결합한 DNA에 UV를 조사하면 파란색 형광을 띰
○ 12th. 관찰 결과 동원체 부위는 avidin의 형광색을 나타낼 것이고, DNA는 파란 형광색을 나타낼 것임
○ 응용 1. RNA ISH
○ rRNA 분자가 개별 세포로부터 분리되는 게 아니라 in situ로 관찰됨
○ labeled roboprobe (complementary RNA sequence)가 transcript와 혼성화됨
○ complementary transcript와 결합하는 probe를 통해 시각화
○ 응용 2. WM ISH (whole-mount ISH) (1989년)
○ atlas를 만들기 위해 수천 마리의 동물이 필요하므로 2010년 이후에는 잘 사용되지 않음
③ 종류 2. radioactive ISH : 최초의 ISH (1969년)
⑹ 시퀀싱 기술(sequencing) : DNA 염기서열 결정
⑺ gene-trap screen
6. 유전자 결손 : DNA 기능 확인 [목차]
⑴ 녹아웃 마우스(knockout mouse) : 특정 유전자의 기능을 연구

① 1st. 배아줄기(ES) 세포에서 유전자 X의 녹아웃 유도
○ 1st - 1st. 플라스미드 벡터(targeting vector) 제작 : neor(neomycin resistant gene) 유전자가 삽입되어 불활성화된 유전자 X와 멀찍이 떨어져 있는 TK(thymidine kinase) 유전자를 포함

○ 1st - 2nd. targeting vector를 ES 세포 내부에 삽입
○ 1st - 3rd. 몇몇 ES 세포는 알아서 기존 유전자 X를 불활성화된 유전자 X로 대체
○ 유전자 재조합이 일어날 수 있도록 유전자 X 옆에 동일한 유전자가 포함되도록 해야 함
○ 1st - 4th. 선별 : 세포들을 G418(geneticin, 네오마이신 유도체), 간시클로비르(gancyclovir)가 포함된 배지에서 배양
○ 1st - 4th - 1st. 형질전환되지 않은 ES 세포 : G418에 의해 사멸됨
○ 1st - 4th - 2nd. 유전자 X 이외의 부위가 대체된 ES 세포 : TK 유전자가 있어서 간시클로비르에 의해 사멸됨 (TK 유전자는 간시클로비르를 분해하여 독성을 유발)
○ 1st - 4th - 3rd. 유전자 X만 대체된 ES 세포만 살아남음
② 2nd. 녹아웃된 ES 세포 선별
③ 3rd. 녹아웃된 ES 세포를 배아에 주입
④ 4th. 키메라 생쥐의 생식세포는 정상 생식세포와 녹아웃된 ES 세포에 의한 생식세포가 생성
⑤ 5th. F1 생쥐는 (+/+) 개체와 (+/-) 개체가 존재
○ 이유 : 키메라 생쥐가 돌연변이 이형접합체라고 볼 수 있기 때문
⑥ 6th. 유전자 검사를 통해 F1 생쥐 중 (+/-) 개체를 선별하여 자가교배
○ 생쥐의 털색을 통해 선별하는 것에는 한계가 있음
⑦ 7th. F2 생쥐 중 25%가 녹아웃 마우스이며 (즉, 동형접합 돌연변이체) 유전자 검사를 통해 선별해야 함
⑵ Cre-Lox
① Cre 유전자는 recombinase를 암호화
② Cre에 의한 DNA 재조합은 동일 lox 염기서열 사이에만 일어남
③ Cre는 lox 염기서열 쌍의 방향이 동일하면 DNA 삭제, 방향이 반대이면 DNA 역위가 일어남
⑶ miRNA, siRNA : 유전자 간섭(RNA interference)을 이용하여 모든 유전자 발현 억제 가능
7. 핵치환 [목차]
⑴ GMO(genetically modified organism) : 유전자 변형 물질, 식품, 생물체
⑵ 방법
① 핵치환
○ 미세주입법 등을 활용
○ 형질전환, 동물복제에 응용할 수 있음
② 벡터를 이용하지 않은 형질전환
○ 유전자총 등을 활용하여 형질전환을 일으킬 수 있음
○ 예 1. 재조합 식물세포 형성 : 재조합 유전자를 입힌 입자들을 식물 세포로 “쏜다.”
○ 예 2. 재조합 식물 세포 → 캘러스(callus) → 개체 형성 (in 영양배지)
○ 식물 세포는 전형성능을 가짐
○ 예 3. 순종의 유지·보급, 유용 식물의 증식 기술
○ 예 4. 생장점 포함 조직 배양, 분화 유도
⑶ 예시
① 예 1. 형질전환 동물 : 유용한 유전자 산물을 대량 생산하는 동물 생산 (예 : 팜잉)
② 예 2. 식용 백신
③ 예 3. 유전자 변형식품 (Genetically Modified Food)
○ 과거 : 선택교배(인공선택)에 의해 특정 대립유전자의 빈도를 증가시킨 유전자 변형 작물
○ 현재 : 유전자 재조합 기술 → 보관기간, 생산율(해충, 잡초, 질병, 가뭄, 추위저항성) ↑
○ 예. 황금쌀 : 베타카로틴(쌀의 영양가↑)을 생산하도록 유전적으로 조작
8. DNA-단백질 상호작용 연구 [목차]
⑴ 유전자 지문법(footprinting assay) : DNA footprinting technology라고도 함

① 단백질과 결합하는 유전자는 전기영동 상에서 사라진 것으로 나타남
② 전사인자 결합부위를 식별할 수 있음
⑵ 전기영동 이동성 변화분석(EMSA, electrophoretic mobility shift assay) : 전사인자와 프로모터 간 결합을 확인
① gel shift assay, GMSA라고도 함
② 1st. probe 제작 : DNA를 방사성 동위원소로 표지
③ 2nd. 단백질과 DNA 혼성액을 전기영동시킨 뒤 방사선 감광 분석
④ 3rd. 결과 분석

Figure. 7. EMSA 결과 예시
○ 전제 : 위가 음극, 아래가 양극이므로 DNA는 위에서 아래로 이동, (-)는 probe만 이동시킨 경우
○ a의 해석 : A 단백질과 B 단백질은 probe와 결합, A와 B는 probe를 낀 채로 결합 (단둘이만 결합할 수도 있고..)
○ b의 해석 : C 단백질과 D 단백질은 probe와 결합, C와 D는 서로 결합하지 않음
○ c의 해석 : E 단백질은 probe와 결합, F 단백질은 probe와 결합하지 않으나 E 단백질과는 결합
⑤ 일반적으로 단백질은 전사인자를 의미하며 전사활성도를 측정하는 데 응용하기도 함
⑶ ChIP(chromatin immunoprecipitation)
⑷ south-western blotting

Figure. 8. south-western blotting
① 1st. 단백질을 먼저 western blotting시킴
② 2nd. 그 뒤 형광을 표지한 DNA를 southern blotting시킴
⑸ yeast one hybrid assay
⑹ 필터결합법 : 전사인자만 필터에 결합
⑺ DNA affinity chromatography
9. 단백질-단백질 상호작용 연구 [목차]
⑴ 이중잡종체계 : 단백질 X와 상호작용하는 단백질을 알아내는 방법
① 1st. 전사인자의 유전자를 두 부분, DNA 결합 부위와 전사 활성화 부위로 분리
② 2nd. 미끼(bait) : 단백질 X + DNA 결합 부위
③ 3rd. 먹이(prey) : 결합 여부를 알고자 하는 단백질 + 전사 활성화
④ 4th. 두 잡종 단백질이 서로 상호작용하여 결합시 먹이가 리포터 유전자 발현 유도
○ 리포터 유전자 예 : GPF 단백질
⑵ yeast two hybrid : 단백질-단백질 상호결합을 이용하여 단백질 결합관계를 확인
⑶ GST-tagged fusion protein을 이용한 단백질 분리 정제
⑷ phage display method : mono clonal 항체를 얻기 위함
① 목적 : 단백질과 결합하는 단일 클론 항체를 얻기 위함
② 시험관 내 단일클론항체를 얻는 방법
③ 보통의 단일 클론 항체 : B 림프구 - 골수종 세포 융합(하이브리도마), 줄기세포가 되어 번식 → 항체 많이 생산
④ 1단계. 돌연변이원을 도입하는 등 염기서열을 달리 하여 방대한 파지 라이브러리를 만듦
⑤ 2단계. 원하는 활성을 갖는 파지를 스크리닝하여 사후에 염기서열이 뭐였는지 확인
10. 유전자 치료(유전자 편집, gene editing) [목차]
⑴ CRISPR/Cas9 유전자 가위 기술 : 크리스퍼라고도 함
① 개요
○ 유전자 가위 : 바이러스 DNA를 제거하기 위한 박테리아(e.g., 대장균)의 유전자 절편화 기작
○ CRISPR(clustered regularly interspaced short palindromic repeat)
○ 원핵세포를 감염시켰던 박테리오파지의 DNA 절편으로부터 유래
○ 박테리아 게놈의 50%, 고세균 게놈의 90%에 발견
○ CRISPR 배열은 반복적인 DNA 서열과 각 반복 사이에 위치한 스패서(spacer)로 구성됨
○ 박테리아 혹은 고세균이 바이러스 등 외부 DNA의 조각인 스패서를 기록한 뒤 반복서열을 부가
○ 이후 CRISPR-Cas9 시스템에 의해 스패서와 일치하는 바이러스 DNA를 탐지하고 분해
○ Cas9(CRISPR-associated protein 9)
○ Streptococcus pyogenes 등에서 획득. nuclease domain HNH, RuVC9로 두 개가 있음
○ dCas9(nuclease-deficient Cas9, dead Cas9) : DNA를 자를 수는 없으나 sgRNA에 의한 DNA binding은 가능
② 단계 1. CRISPR 가공 : 다음은 Bacillus halodurans의 subtype I-C/Dvulg Cas5d 시스템의 CRISPR 가공과정을 설명
○ 1st. Cas5d는 CRISPR 반복 영역에서 헤어핀 구조와 3' 단일 가닥 서열을 인식하여 pre-crRNA를 단위 길이로 절단
○ 2nd. pre-crRNA processing : pre-crRNA를 더 작은 크기의 crRNA로 가공
○ 3rd. Cas5d가 crRNA, Csd1, Csd2 단백질과 복합체를 형성
○ 4th. 이 복합체에 있는 crRNA 부분이 바이러스 DNA를 탐지하고 제거
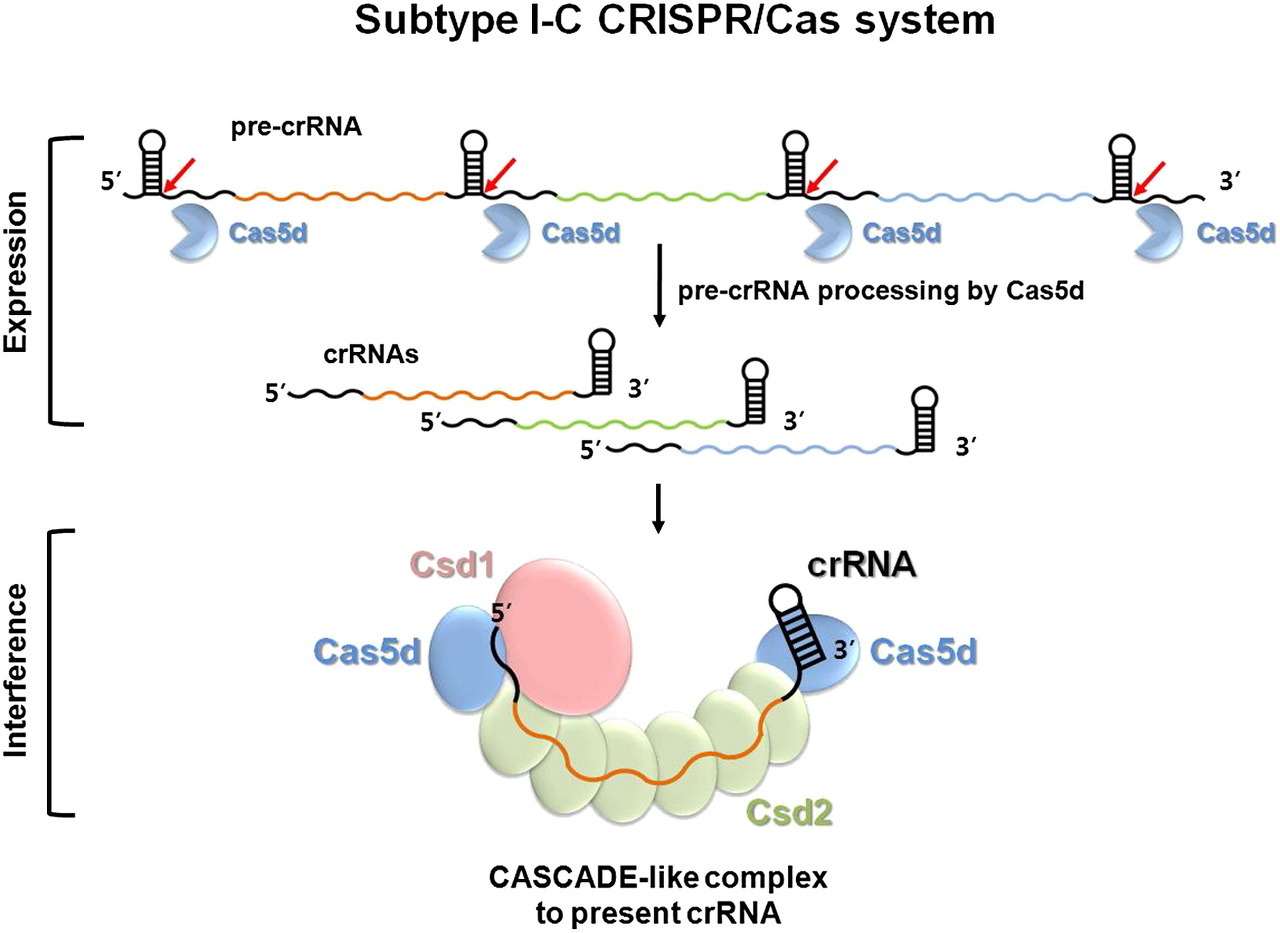
Figure. 9. CRISPR 가공
③ 단계 2. 외부 핵산에 대한 반응
○ 1st. Cas9이 이중가닥 DNA에 버블을 만들고 그 사이로 PAM site 옆에 있는 타겟 RNA와 상보적인 sgRNA (small guide RNA)가 결합
○ 그 버블을 DSBs(DNA double-strand breaks)라고 함
○ sgRNA : 박테리아(e.g., 대장균)가 바이러스에 감염 후 살아남았을 때 유전체 DNA에 남아 있는 바이러스 DNA 일부. 스패서 혹은 gRNA(guide RNA)라고도 함
○ PAM(protospacer adjacent motif) site : 5'-NGG-3'의 염기서열. self와 non-self를 구분

Figure. 10. dsDNA에서 sgRNA의 위치
○ 2nd. sgRNA-Cas9 complex가 동일한 위치의 주형 DNA, 비주형 DNA, sgRNA를 모두 절단
○ 3rd. Cas9과 sgRNA가 분리
○ 4th. 버블이 형성됐던 DNA는 수소결합에 의해 다시 결합
○ 5th. DNA 수선기작을 통해 절단된 주형, 비주형 DNA는 다시 연결
○ 5th - 1st. DNA 수선기작이 없는 바이러스는 CRISPR/Cas9 메커니즘에 의해 제거됨
○ 5th - 2nd. 비상동말단 연결(non-homologous end joining; NHEJ) : 전혀 다른 염색체가 결합할 수 있음
○ 홀리데이 모델을 따름
○ 5th - 3rd. 상동재조합 수선(homology directed repair; HDR) : 특정 염기가 치환되거나 반복서열이 부가되는 등의 효과가 나타남
○ 6th. 유전자 재조합에서 일반적으로 VNTR 유전자의 필요없는 부분을 유용한 DNA로 교체하는 연구가 이뤄지고 있음
○ 상동재조합 수선 기작을 이용하게 됨
○ DNA 뿐만 아니라 RNA, epigenome, 기타 single nucleotide 등을 editing 할 때 쓸 수 있음
○ 유전자 편집 종류 : OE(overexpression), KD(knock-down)
④ off-target 문제
○ 정의 : CRISPR/Cas9 시스템을 유전자 치료에 적용할 때 타겟 유전자 이외의 부위가 편집되는 문제
○ Streptococcus pyogenes Cas9 (SpCas9) nuclease가 효율성을 이유로 가장 많이 사용되지만 off-target 비율이 높음
○ 해결방법
○ nuclease mutation
○ PAM(protospacer adjacent motif) sequence modification
○ gRNA(guide RNA) truncation
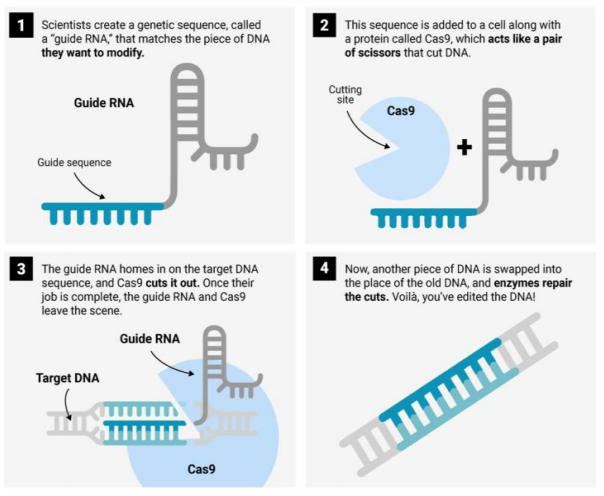
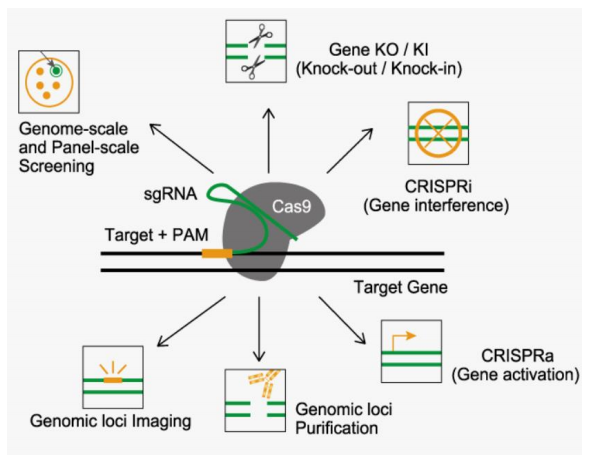
Figure. 11. CRISPR/Cas9 기술 도식도
⑤ 활용 사례
○ 신호 전달 연구 목적 : multiplexing guide RNA들로 perturbation screening을 할 수 있음
○ 이미징 연구 목적
○ 약물 연구 목적 : 특정 유전자를 억제했을 때 약물 섭취가 변하는지 등
○ 유전자 치료 : 희귀 질환을 유발하는 대립 유전자를 편집
○ 시간적 시퀀싱 (e.g., Record-seq)
⑵ 용원성 바이러스 : 유전자 치료에 주로 이용되는 바이러스는 아데노 바이러스와 레트로 바이러스임
⑶ siRNA, miRNA 치료제 : 현재 시장 상황은 좋지 않음
⑷ 핵산 약물전달 시스템
① 개요
○ COVID-19 팬더믹 상황에서 모더나, 화이자가 채택한 전략으로 상업적으로 굉장한 성공을 하였음
○ mRNA는 체내에서 불안정하기 때문에 target tissue 내지 cell에 도달할 때까지 안정할 수 있도록 전달체가 필요
② 종류 1. mRNA 약물전달 시스템
○ SLN(solid lipid nanoparticle) : 모더나, 화이자도 채택한 가장 인기 있는 mRNA 전달체
○ 80-100 nm의 SLN 1개는 약 100개의 mRNA를 담지함
○ 예 : ALC-0315 (Pfizer/BioNTech), SM-102 (Moderna), ALC-0159 (Pfizer/BioNTech), PEG-DMG (Moderna)
○ cationoic liposome
○ polymer and polymer/lipid hybrid particle
○ micelle
○ emulsion
③ 종류 2. DNA 약물전달 시스템
④ 종류 3. AAV(adeno-associated virus)
○ 치매 치료제 제형으로도 사용됨
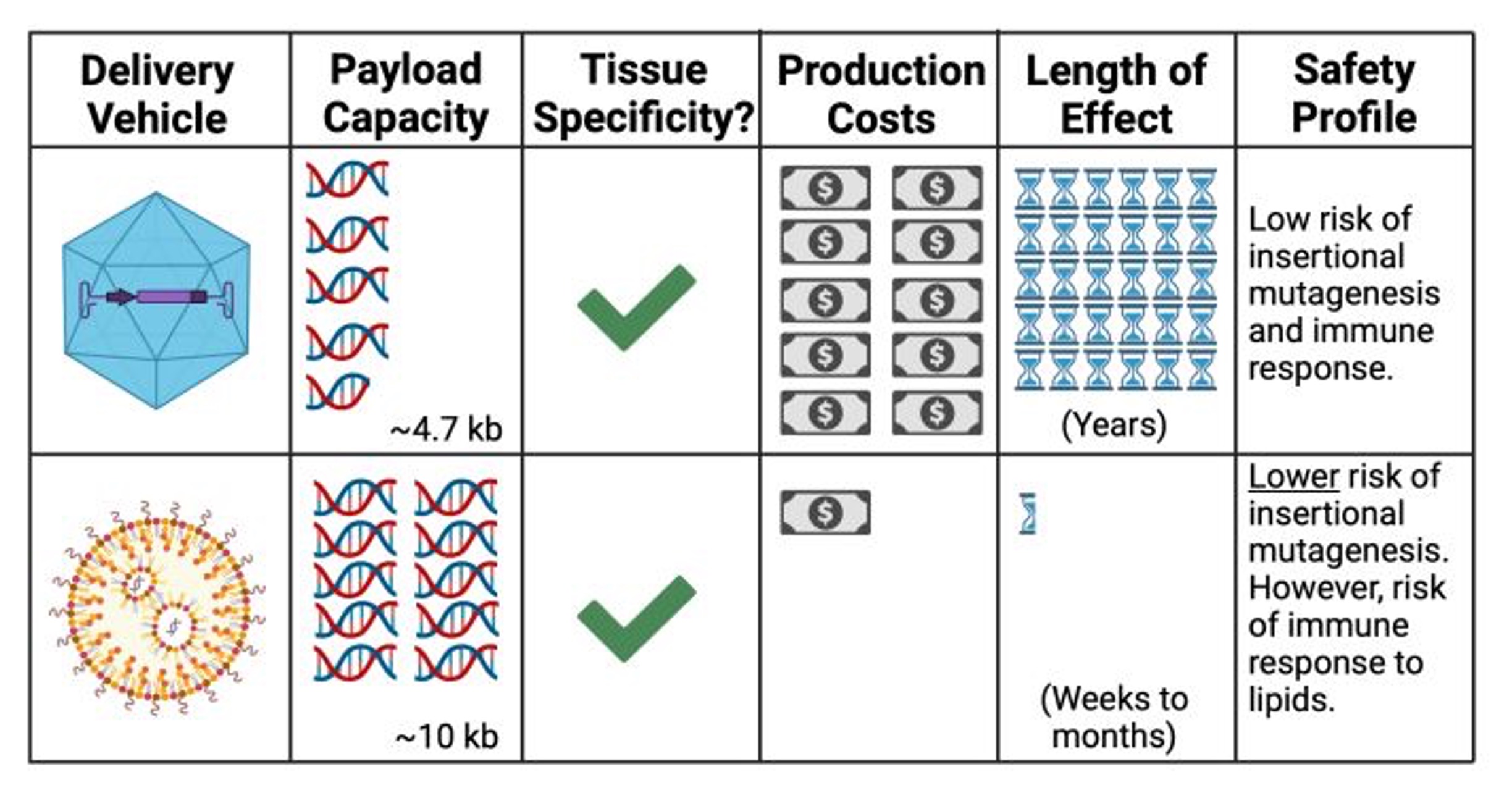
Figure. 12. AAV와 LNP의 비교
⑸ mega nuclease
⑹ TALEN(transcription activator-like effector nuclease)
⑺ ZFN(zinc finger nuclease)
입력: 2015.07.03 21:57
수정: 2021.01.17 10:36
'▶ 자연과학 > ▷ 일반생물학' 카테고리의 다른 글
| 【생물학】 12강. 동물의 체계화 수준 (0) | 2019.02.16 |
|---|---|
| 【생물학】 9-3강. 로마노프 왕가와 법의학 (0) | 2019.02.16 |
| 【생물학】 5-1강. 유전자의 수평적 전달 (0) | 2019.02.16 |
| 【생물학】 8강. 중심학설 (5) | 2019.02.16 |
| 【생물학】 7강. 유전과 유전학 (4) | 2019.02.16 |




최근댓글