10-2강. 후성유전체 시퀀싱(epigenomics sequencing)
추천글 : 【생물학】 10강. 게놈 프로젝트와 시퀀싱 기술
1. 종류 1. gene function 식별 [본문]
2. 종류 2. transcription regulation 식별 [본문]
3. 종류 3. post-translational regulation 식별 [본문]
4. 종류 4. programmable cell function [본문]
a. 후성유전학 라이브러리
1. 종류 1. gene function 식별 [목차]
⑴ Perturb-seq
① 1st. 여러 종류의 gRNA library를 Cas9-expressing cell을 처리
○ CRISPR-Cas9을 사용함으로써, 각 세포에서는 gRNA의 종류에 따라 다양한 변화(perturbation)가 발생
○ 필터링 후 250만 개 이상의 세포가 있어야 함
○ perturbation 당 평균 100개 이상의 세포가 있어야 함
○ 세포당 10,000개 정도의 UMI가 있어야 함
② 2nd. gRNA와 mRNA를 모두 탐지하는 시퀀싱 기술을 사용 (e.g., scRNA-seq, MERFISH)
③ 3rd. gRNA를 발현하는 세포별로 그룹핑 : 자연스럽게 perturbation 조건별로 세포들이 그룹핑
④ 4th. gene 기능 식별
○ 전제 : 비슷한 기능을 하는 gene끼리는 비슷한 expression 패턴을 보일 가능성이 높음
○ 알 수 있는 결과 1. perturbation에 따른 유전자 발현 변화
○ 알 수 있는 결과 2. 유전자 변이에 따른 perturbation 영향 차이
Figure. 1. 검증 실험을 포함한 Perturb-seq의 일반적인 모식도
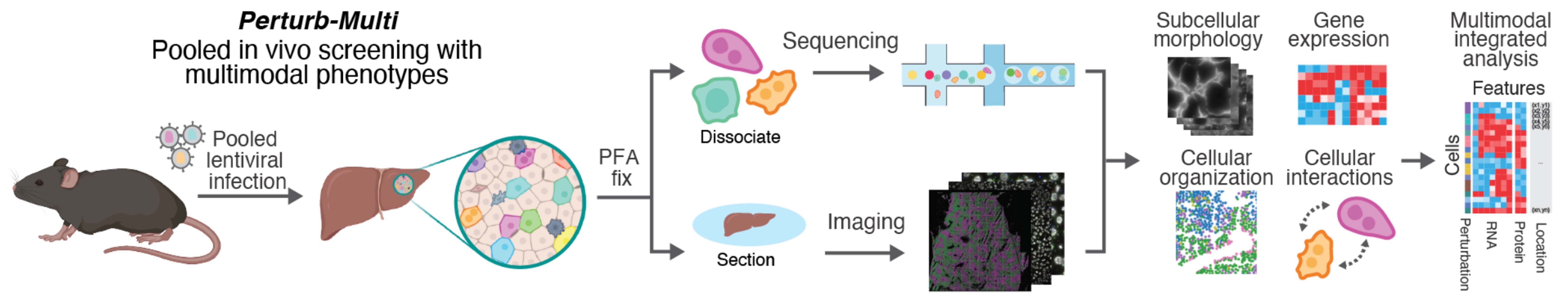
⑵ in vivo Perturb-seq
① 1st. 마우스에 Cre를 포함한 AAV(Adeno-associated virus)를 전달하여 마우스가 Cas9을 발현하도록 함
② 2nd. sgRNA를 담고 있는 lentivirus를 전달
③ 3rd. CRISPR-Cas9 시스템에 의해 in vivo에서 각 Cas9 발현 세포에서 gRNA에 따라 다양한 변화(perturbation)가 발생
④ 4th. 약 10일 간 반응을 기다린 뒤 scRNA-seq 및 MERFISH를 수행
Figure. 2. in vivo Perturb-seq 과정
2. 종류 2. transcription regulation 식별 [목차]
⑴ BS-seq(bisulfite sequencing)
① bisulfite를 처리하여 methylation 패턴을 결정
② epigenomic profilig을 가능하게 함
③ 종류 1. snmC-seq
○ 플레이트 기반 시퀀싱
○ 5-methyl-cytosine과 5-hydroxymethyl-cytosine의 합을 측정함
○ 세포당 100만 ~ 200만 리드
⑵ ChIP-seq(chromatin immunoprecipitation sequencing)
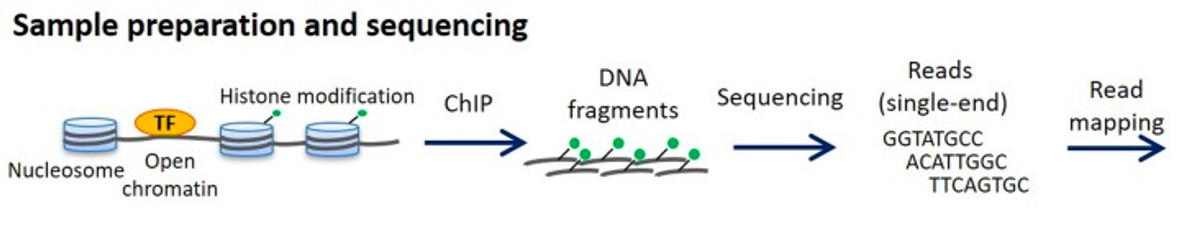
① 정의 : 전사인자 등 특정 단백질과 결합한 DNA 부위를 분석할 수 있음
② DNA-associated protein의 결합 자리를 알기 위해 DNA 시퀀싱과 ChIP(chromatin immunoprecipitation)를 결합
③ 1st. 단백질(예 : 전사인자)과 DNA를 고정하기 위해 cross-linking agent (예 : formaldehyde)를 처리
④ 2nd. 세포를 용해시켜 DNA/protein complex만 남도록 함
⑤ 3rd. sonication을 통해 DNA를 작은 조각들로 파괴 : 200-350 bp. cross-link 된 DNA는 파괴되지 않음
⑥ 4rd. magnetic bead가 부착된 항체를 첨가 : 자기장을 가하면 특정 단백질 및 그와 연결된 DNA를 분리할 수 있음
⑦ 5th. reverse cross-linking : 침전물에 열을 가하여 DNA를 단백질로부터 분리
⑧ 6th. 염기서열을 알기 위해 sequencing을 진행 : PCR amplification, fragment size selection, adaptor addition이 진행됨
⑨ 7th. 최종적으로 게놈 레퍼런스와 비교하여 전사인자 등의 binding site를 알 수 있음
Figure. 3. ChIP-seq 과정
⑩ 응용 1. ChIP-chip : ChIP-chip의 HTS 버전이 ChIP-seq
⑪ 응용 2. ChIP-PET
⑫ 응용 3. ChIA-PET : ChIP-seq은 전사인자 등 특정 단백질의 결합 위치만 알 뿐이고 ChIA-PET은 결합된 DNA 사이의 상호작용 조사
⑬ 응용 4. RIP-seq
⑭ 응용 5. CLIP-seq
⑮ 응용 6. meDIP-seq : DNA methylation 또는 hydroxymethylation
⑯ 응용 7. ChIP-exo : lambda exonuclease digestion 사용. 실험 기법이 어려움
⑰ 응용 8. CUT&RUN (cleavage under targets and release using nuclease)
○ MNase (micrococcal nuclease) 사용
⑱ 응용 9. CUT&Tag (cleavage under targets and tagmentation)
○ 개요
○ Tn5 transposase는 DNA의 열린 구간을 자르는 효소로 single nucleosome, dimer, trimer, ⋯ 등 다양한 절편을 만듦
○ Tn5 transposase는 sticky end를 만들어서 DNA 삽입을 촉매함
○ adaptor를 직접 병합할 수 있어서 바로 시퀀싱을 진행할 수 있음
○ 과정
○ 단계 1. 핵들에 magnetic bead를 붙인 뒤 자기장을 이용해 세포들을 획득
○ 단계 2. 1차 항체 첨가 : 항체의 퀄리티가 데이터 퀄리티에 큰 영향을 줌
○ 단계 3. 2차 항체 첨가
○ 단계 4. conjugated Tn5-transposase가 복합체에 결합하고 열린 DNA 영역 주변을 절단
○ 단계 5. PCR amplification, fragment size selection, 시퀀싱
○ 종류
○ MuLTI-Tag : direct barcode conjugation을 통해 multiplexing에서의 crossover를 최소화
○ multi-CUT&Tag : barcoded Tn5/pA-antibody complex를 이용. mark들의 colocalization 등을 확인할 수 있음
○ spatial-CUT&Tag : 조직 상에 공간적으로 히스톤 수식 및 염색체 상태를 보여줌
| CUT&Tag | CUT&RUN | ChIP-Seq | |
| native condition? | Yes | Yes | No |
| sample input | nuclei | cells or nuclei | sheared chromatin |
| cell number | 100,000 cells | 500,000 cells | 1-10 million cells |
| chromatin fragmentation | Tn5-based tagmentation | MNase digestion | sonication |
| ideal target | histone PTM | histone PTM, chromatin-associated protein, remodeler | histone PTM, chromatin-associated protein |
| secondary antibody | Yes | No | No |
| library preparation | No (direct-to-PCR) | Yes | Yes |
| integrated library | possible; uses tagmentation | impossible | impossible |
| sequencing depth | 5-8 million reads | 3-5 million reads | 20-50 million reads |
| workflow length | < 2 days | < 3 days | ~ 1 week |
| automation compatibility | high | high | low |
| signal-to-noise | high | high | low |
Table. 1. CUT&Tag vs. CUT&RUN vs. ChIP-Seq (ref, ref, ref, ref)
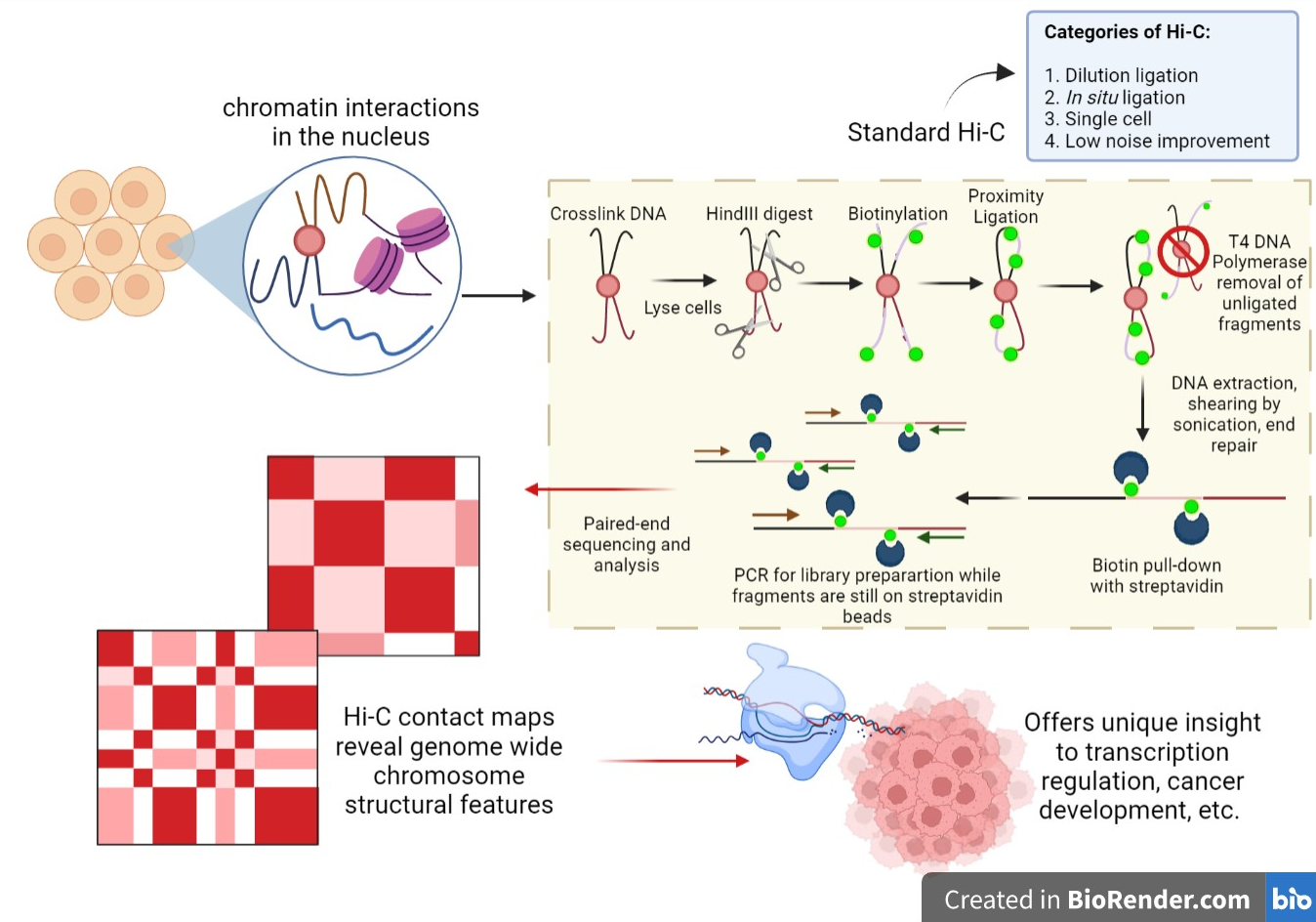
⑶ Hi-C(high throughput chromatin conformation capture sequencing)
① 정의 : 자연적으로 염색체 상에서 가깝게 인접한 염기서열을 조사
② DNA 간 거리를 조사하여 핵 내 염색체의 3차원 접힘 구조를 볼 수 있음
Figure. 4. Hi-C 과정
③ 응용 1. ChIA-PET
○ 차이 : Hi-C는 자연적으로 형성된 모든 DNA-DNA 상호작용. ChIA-PET은 특정 단백질이 매개하는 DNA-DNA 상호작용을 연구
⑷ DNA ticker tape (prime editing)
① 1st. 맨 처음에는 첫 번째 사이트만 활성화 돼 있음
② 2nd. 첫 번째 이벤트가 일어나면 두 번째 사이트가 활성화됨
③ 3rd. 이런 식으로 시계열적 분자적 이벤트를 기록할 수 있음
⑸ ENGRAM(enhancer-driven genomic recording of transcriptional activity in multiplex)
① synthetic enhancer와 연결된 perRNA를 이용
② 신호전달의 순서와 강도를 기록할 수 있음
③ 레퍼런스 : Chen et al., bioRxiv (2021)
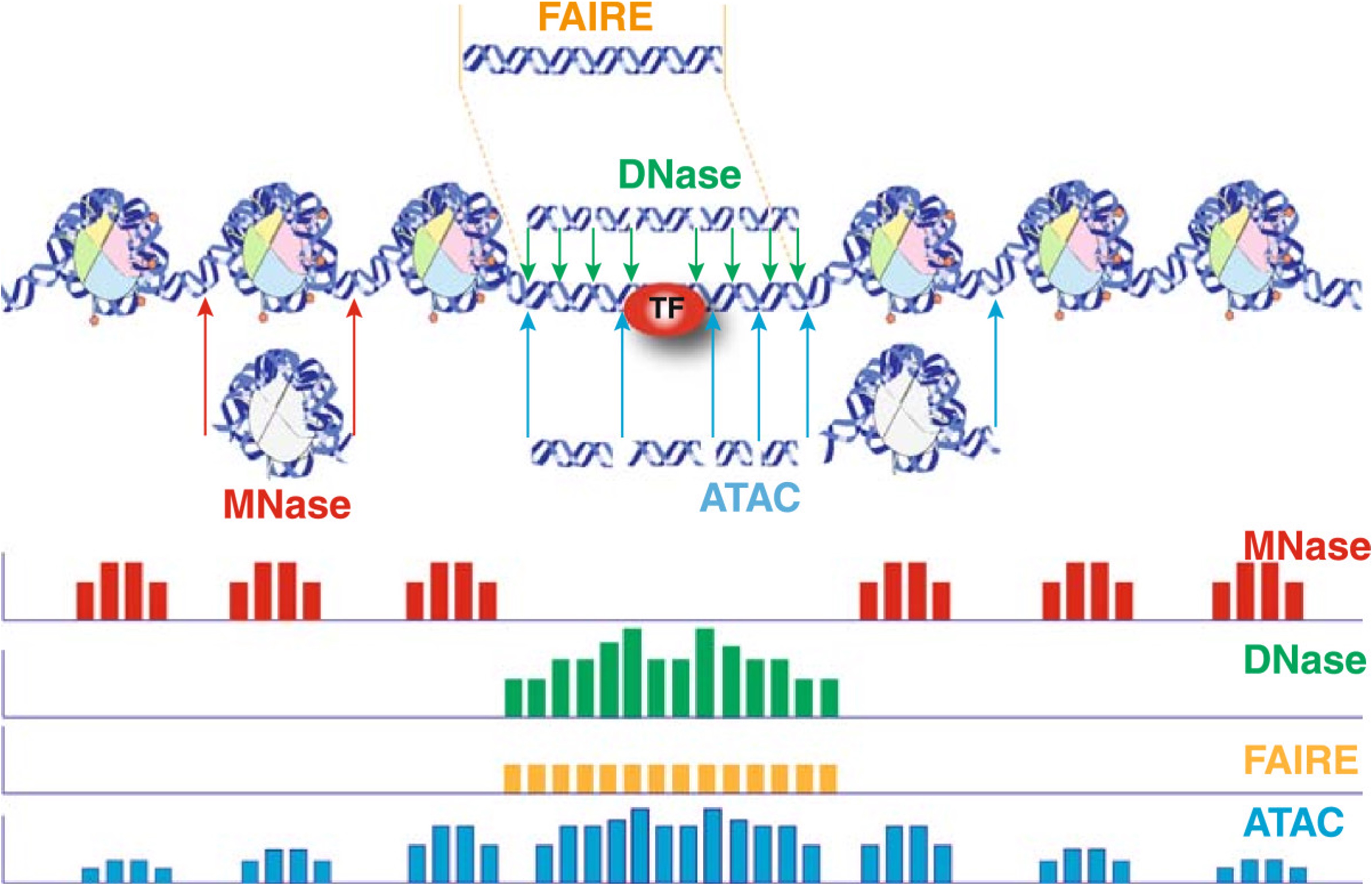
⑹ ATAC-seq (assay for transposase-accessible chromatin with sequencing)
① 개요
○ 정의 : 진정염색질 부분을 표시해 주는 시퀀싱 기법. 염색체는 1쌍이므로 0, 1, 2의 값을 가질 수 있음
○ pseudo-expression : 진정염색질은 gene expression이 있는 영역으로 추정할 수 있음
○ Tn5 transposase는 DNA의 열린 구간을 자르는 효소로 single nucleosome, dimer, trimer, ⋯ 등 다양한 절편을 만듦
○ Tn5 transposase는 sticky end를 만들어서 DNA 삽입을 촉매함
○ adaptor를 직접 병합할 수 있어서 바로 시퀀싱을 진행할 수 있음
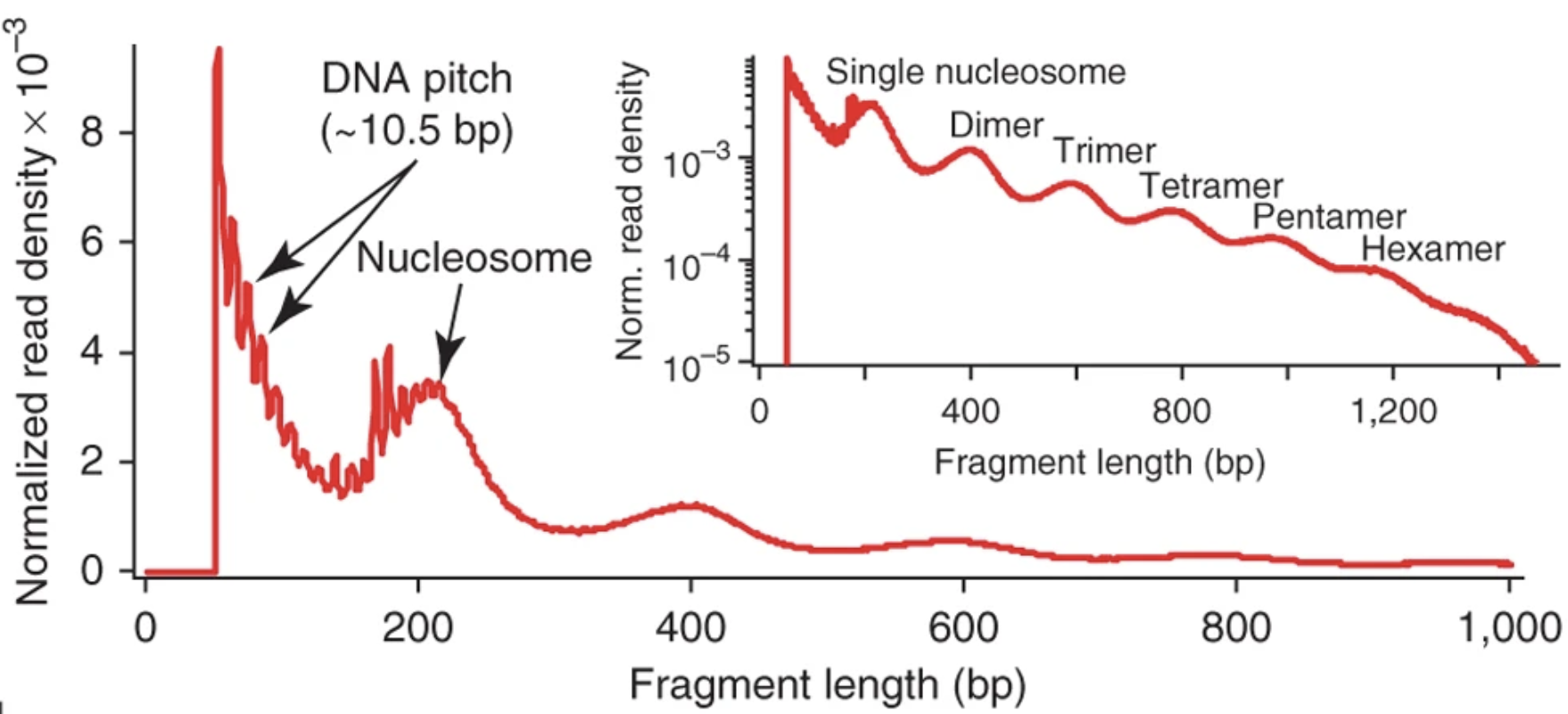
○ ATAC-seq에서 관찰되는 10.5 bp 정도의 periodicity가 DNA 1 회전당 10 bp가 필요한 것과 관련
○ Tn5의 네 번째 알파나선은 인식 부위 : DNA major groove에서 염기쌍 특이적인 접촉을 7번에서 13번 위치에서 수행 (ref)
○ Tn5는 나선 축을 구부려서 roll 및 tile 각도가 증가하고, 일반적인 B형 DNA와 비교했을 때 major groove 및 minor groove의 폭과 깊이에서 편차가 발생하게 함 (ref)
○ 이것이 일반적인 B-DNA에서의 10bp 주기성과는 약간 다른 10.5bp 주기성을 설명할 수 있음
Figure. 5. ATAC-seq fragment size distribution
② 종류 1. bulk ATAC-seq
○ 단계 1. 핵 분리(nuclei isolation)
○ 단계 2. Tn5 transposase 처리 : 이는 염색체에서 응축이 덜한 open region을 잘라 DNA sequence tag를 삽입
○ 단계 3. amplification & sequencing
○ 단계 4. 데이터 분석
③ 종류 2. scATAC-seq
○ cell type에 특이적인 CRE를 정의하여 regulatory TF를 규명하고 질환 및 형질과 관련된 cell type을 식별
○ GWAS variant를 해석할 때 scATAC-seq이 활용됨
○ scRNA-seq과 scATAC-seq의 비교
○ scRNA-seq : xij ∈ ℤ≥0
○ scATAC-seq : xij ∈ {0, 1}, j ≫ i
④ 종류 3. spatial ATAC-seq
⑤ 종류 4. FAIRE-seq : 염색질 교차결합을 위해 포름알데하이드를 처리하고, sheared DNA를 추출하기 위해 페놀-클로로포름 사용
⑥ 종류 5. DNaseI-seq : 염색질을 digest하기 위해 DNase I 처리. 염색질과 상호작용하는 전사인자의 종류를 알 수 있음
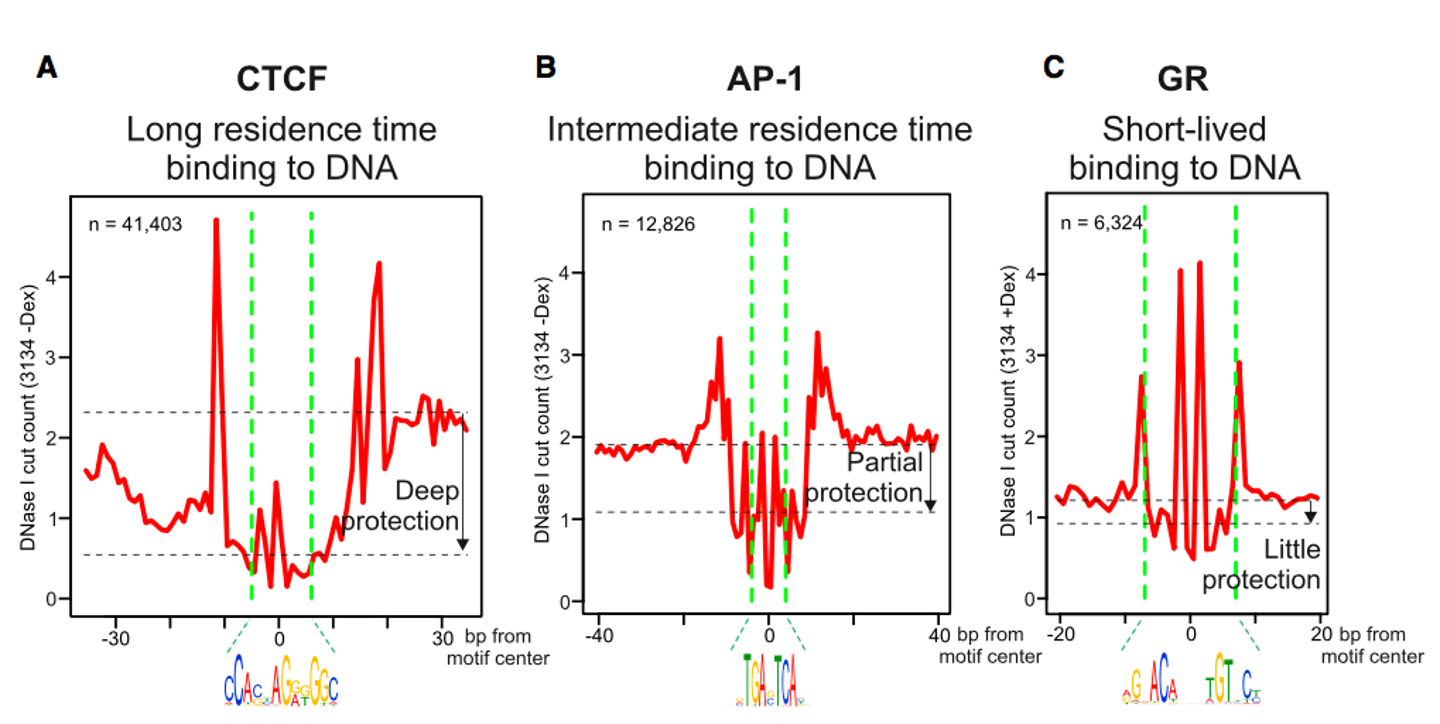
Figure. 6. DNaseI-seq 결과 예시
⑦ 종류 6. MNase-seq : endo-exonuclease 처리
Figure. 7. ATAC-seq과 다양한 시퀀싱 기법 비교
⑺ NOMe-seq (nucleosome occupancy and methylome sequencing)
① TF가 결합하는 뉴클레오솜 고갈 영역(NDR)을 정의
② 염색질과 상호작용하는 전사인자의 종류를 알 수 있음
⑻ MBD-seq
⑼ Bru-seq & BruChase-seq
① 전사된지 얼마 안 된 nascent RNA를 Bru(bromouridine)를 표지한 뒤 시퀀싱
② RNA 합성, RNA 안정성, 스플라이싱 등을 연구할 때 사용
⑽ TT-seq
⑾ MNIST-seq : RNA methylation on chromatin regulation
⑿ GRO-seq : global run-on sequencing
3. 종류 3. post-translational regulation 식별 [목차]
⑴ Ribo-seq
① 리보솜에 의해 보호된 RNA를 시퀀싱. 활발한 번역을 나타냄
② scRibo-seq : 각 single codon 별 ribosomal occupancy를 측정
○ 1st. FACS 및 lysis
○ 2nd. nuclease footprinting : MNase nuclease → inactivation → release of footprints
○ 3rd. small-RNA library 생성 : end repair → 3' ligation → 5' ligation → cDNA synthesis → indexing PCR
⑵ STAMP-RBP
① RBP(RNA binding protein)을 찾기 위해 scRNA-seq을 이용
② 1st. RBP에 APOBEC를 붙임
③ 2nd. APOBEC와 mRNA가 붙은 지점에서 C-U editing이 되도록 함 : 즉, C 염기를 U 염기로 치환
④ 3rd. RNA-seq
⑤ 4th. SAILOR를 이용하여 C-U editing이 일어난 지점을 식별
⑥ long read sequencing을 이용하여 isoform-specific binding profile을 식별할 수도 있음
4. 종류 4. programmable cell function [목차]
⑴ RADARS
① 1st. target transcript가 있을 때 dsRNA를 형성하여 ADAR로 하여금 A-C editing이 일어나게 함
② 2nd. 위 editing으로 인해 GFP, caspase 등 cellular behavior가 일어나도록 함
⑵ LADL(light-activated dynamic looping)
① photo-activatable gene expression의 예시 중 하나
입력: 2022.01.10 00:03
수정: 2023.01.28 23:12
'▶ 자연과학 > ▷ 일반생물학' 카테고리의 다른 글
| 【생물학】 4-5강. 단백질 합성 (0) | 2022.04.12 |
|---|---|
| 【생물학】 약물 스크리닝 기술 (0) | 2022.01.18 |
| 【생물학】 TEM negative staining 프로토콜 (0) | 2021.12.17 |
| 【생물학】 알부민 (0) | 2021.10.13 |
| 【생물학】 1-3강. 생체고분자 라이브러리 (0) | 2021.10.11 |



최근댓글