2023년 제60회 변리사 2차 국가자격시험
추천글 : 【분자생물학】 변리사 2차 분자생물학 기출문제 풀이
a. 중심학설
b. DNA 테크놀로지
문제 1. DNA 복제에 관한 다음 물음에 답하시오.
⑴ 1-1. DNA 두 가닥의 방향성과 DNA 중합효소의 특성으로 인하여, 연속적인 복제와 불연속적인 복제 두 가지 방식으로 일어난다. 이와 관련이 있는 두 가닥의 방향성과 DNA 중합효소의 특성을 설명하시오.
○ DNA 복제가 개시될 때, DNA 나선을 풀어야 하는데 이때 헬리케이스 등의 효소가 관여한다. 헬리케이스는 지연가닥(lagging strand)의 주형에 결합하여 5' → 3'으로 이동한다. 비슷하게 DNA 중합효소 역시 5' → 3'으로 중합반응을 일으키므로 헬리케이스와 이동방향이 같은 쪽에서 선도가닥(leading strand)의 연속적인 복제가, 이동방향이 반대인 쪽에서 지연가닥(lagging strand)의 불연속적인 복제가 발생한다. 지연가닥은 여러 종류의 fragment로 나뉘어 있는데 이를 오카자키 절편(Okazaki fragments)이라고 한다.
⑵ 1-2. 지연가닥(lagging strand)은 복제분기점 근처에서 짧은 오카자키 조각(Okazaki fragment)을 형성하기 시작하여, 이전에 형성된 이웃한 오카자키 조각과 연결하기까지 일련의 과정을 반복하여 합성된다. 세균의 복제에서 틈 이동(nick translation)이 일어나는 이유와 이에 관여하는 효소들의 기능을 설명하시오. (단, DNA 중합효소 Ⅰ과 Ⅲ을 구분하고, 프라이머(primer)와 오카자키 조각의 말단 방향을 5'과 3'으로 표시하여 답하시오.)
○ nick translation이란, 프라이머를 제거하고 그 자리를 dNTP로 교체하는 과정으로 기본적으로 프라이머가 RNA이기 때문에 DNA 복제가 완전히 진행되기 위해서는 RNA 교체가 필요하기 때문이다.
○ 대장균의 DNA 중합효소는 DNA pol Ⅰ, Ⅱ, Ⅲ가 존재하고 그 각각의 기능은 다음과 같다.
○ DNA pol Ⅰ : 틈 번역(nick translation). 프라이머 제거 및 RNA 프라이머 자리를 dNTP로 교체
○ DNA pol Ⅱ : 외부 요인에 의해 손상된 DNA 수리, DNA 합성이 중단되면 활성화
○ DNA pol Ⅲ : DNA 신장, 이합체로 작용하며 실제로는 자기가 이동하지 않고 DNA 가닥이 움직임
○ 그림 표시

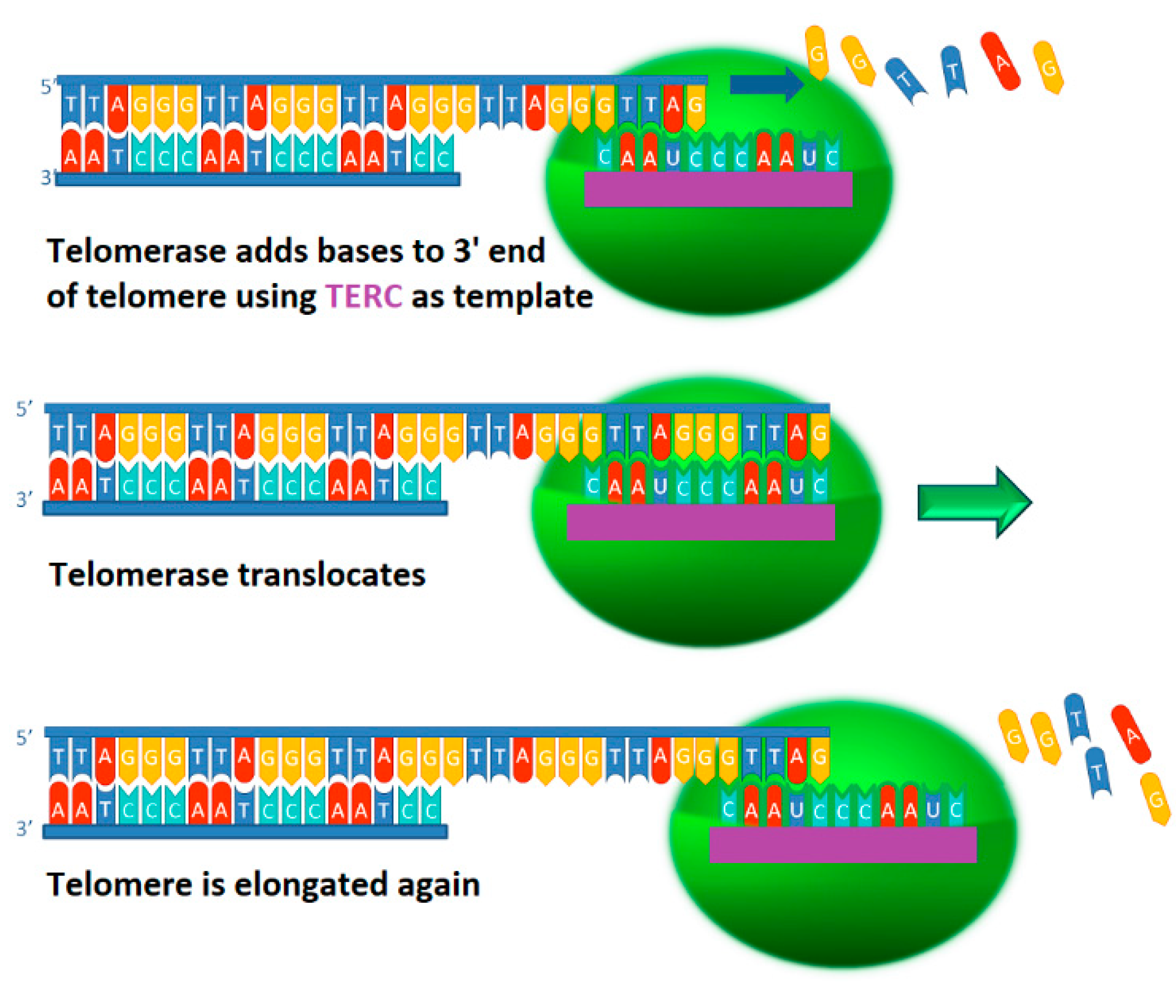
⑶ 1-3. 선형 염색체 복제에서 일반적으로 DNA 중합효소로는 지연가닥의 5' 말단 부위의 복제를 완성하지 못하여 말단 부위는 여러 번 복제를 거듭할수록 점점 길이가 짧아진다. 텔로머레이스(telomerase)가 염색체 말단 부위의 길이를 어떻게 보존하는지 설명하시오.
○ 텔로미어(telomere)란, 무의미한 반복서열로서 주로 6개의 뉴클레오타이드가 300 ~ 5000번 반복되는 식으로 구성된다. 위 그림에서 위쪽 strand의 3' 말단이 노출돼 있지만, 텔로미어 상의 tetra G가 t-loop를 형성하여 노출된 텔로미어 3' 말단을 헤어핀 구조로 변형하여 이 노출된 말단이 보호된다.
○ 텔로머레이스(telomerase)는 1) 분자 내 RNA가 있고, 2) 역전사 효소이며, 3) RNA 의존성 DNA 중합효소라는 특징이 있다. 따라서 텔로머레이스 분자 내 짧은 RNA(즉, 3'-CCCAAUCCC-5' RNA 주형)가 주형으로 기능함과 동시에 텔로머레이스 특유의 DNA 중합효소 활성이 있어서 짧아진 텔로미어를 다시 길어지게 한다. 이로 인해 염색체 말단 부위의 텔로미어 길이가 보존된다.
문제 2. 진핵생물의 단백질 암호화 유전자들은 일반적으로 유전자 프로모터(promoter)에 가까운 근거로 조절 요소들과 프로모터로부터 100 kb 이상의 먼 거리에 떨어져 있는 원거리 조절 요소(long-range regulatory element)들에 의해 전사가 조절된다. 다음 물음에 답하시오.
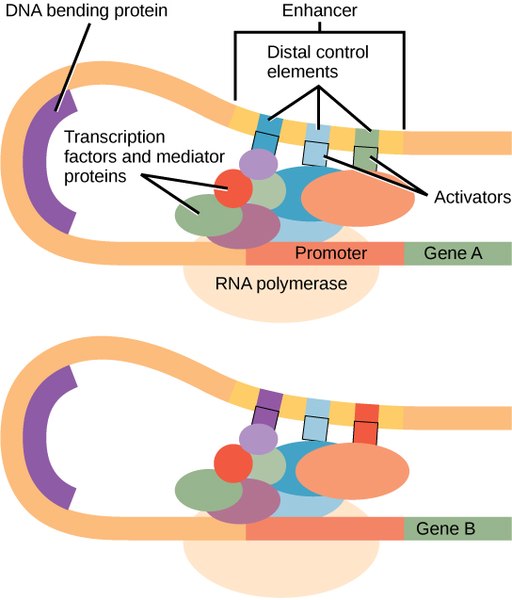
⑴ 2-1. 유전자가 염색체의 어느 부위에 위치하는가에 따라 그 유전자의 발현이 달라지는 현상을 위치 효과(position effect)라 한다. 이 위치 효과에서 인핸서(enhancer)의 특징을 설명하시오.
○ RNA 중합효소의 프로모터 결합 촉진한다.
○ 프로모터에 대하여 상류나 하류 모두에 있을 수 있다.
○ 길이는 50-1500 bp 정도이다.
⑵ 2-2. 유전자 프로모터로부터 멀리 위치한 인핸서에 결합한 활성자(activator)들이 프로모터에 결합한 RNA 중합효소 Ⅱ와 일반적 전사인자(general transcription factors)와 어떻게 상호작용하는지 설명하시오.
○ (참고) 진핵생물의 DNA dependent RNA polymerase는 3가지
○ RNA pol Ⅰ : 인에서 작용. 28S rRNA, 18S rRNA, 5.8S rRNA 합성
○ RNA pol Ⅱ : 핵질에서 작용. mRNA의 합성
○ RNA pol Ⅲ : 핵질에서 작용. tRNA, 5S rRNA, ncRNA, snRNA 합성
○ DNA 접힘 단백질(DNA bending protein), 코헤신, CTCF(11-zinc finger protein, CCCTC-binding factor) 등에 의해 인핸서에 결합한 활성자들이 RNA 중합효소 Ⅱ의 프로모터 상의 물리적 결합을 촉진함
○ 그 뒤 일반적 전사인자와 RNA 중합효소 Ⅱ의 상호작용이 일어남
○ 1st. TFIID가 TFIID 상의 TBP 서브유닛을 통해 TATA 박스에 결합
○ 2nd. TFIID는 TFIIA에 의해 안정화됨
○ 3rd. TFIIB와 TFIIH가 TATA 박스 상에 있는 TFIID-TFIIA 복합체에 결합
○ 4th. RNA pol Ⅱ-TFIIE-TFIIF 복합체가 C-terminal domain (CTD) 부분으로 앞의 복합체 상에 있는 TFIID와 결합
○ 5th. TFIIH는 인에서 RNA pol Ⅱ의 CTD를 인산화 → TFIID 방출 및 헬리케이스 활성
○ RNA pol Ⅱ는 α2ββ'ω와 달리 헬리케이스 활성 ×
○ 6th. 전사가 개시됨
문제 3. 원핵생물과 진핵생물에서 단백질 합성 과정은 서로 다르다. 다음 물음에 답하시오.
⑴ 3-1. 다음 설명 중에서 진핵세포에 해당하는 설명을 3개 고르고, 이 설명을 원핵세포에 대한 설명으로 수정하시오.
① mRNA에 캡(cap) 구조가 없다. (원핵세포)
② 80S 개시 복합체가 형성된다. (진핵세포) → 70S 개시 복합체가 형성된다. (원핵세포)
③ mRNA는 폴리시스트론성(polycistronic)이다. (원핵세포)
④ 개시 단계에서 첫 번째 아미노산은 메티오닌(methionine)이다. (진핵세포) → fMet(formyl methionine) (원핵세포)
⑤ 리보솜의 큰 소단위체는 28S rRNA, 5.8S rRNA, 5S rRNA를 포함한다. (진핵세포) → 리보솜의 큰 소단위체는 5S, 23S rRNA를 포함한다. (원핵세포)
⑥ EF-Tu는 활성화된 tRNA를 리보솜으로 전달하는데 효과적으로 작용한다. (원핵세포)
⑵ 3-2. 〈보기〉에 있는 용어를 모두 사용하여 원핵세포에서 개시복합체의 형성 단계를 3단계로 설명하시오. (단, 용어는 반복하여 사용할 수 있다.)

○ 단계 1.샤인-달가르노 서열(Shine-Dalgarno sequence)에 30S 리보솜 소단위체(16S rRNA + 단백질)가 결합한 후 개시 5'-AUG가 나올 때까지 이동합니다.
○ 단계 2. 개시 tRNA (fMet-tRNAfMet)가 mRNA의 개시 코돈을 인식한 뒤, 30S 리보솜 소단위체의 A 자리, E 자리, P 자리 중 P 자리에 결합합니다. 이때 GTP가 사용됩니다.
○ 단계 3. 개시인자(IF-1, IF-2, IF-3)들과 50S 리보솜 대단위체(오타?)가 결합하여 번역 개시복합체를 형성합니다.
⑶ 3-3. 다음은 단백질 합성 억제제이다. 각 억제제가 원핵세포와 진핵세포 중 어느 세포에서 작용하는지 구별하고 억제 작용 원리를 설명하시오.
① 스트렙토마이신(streptomycin) : 원핵세포에서 작용. 진정세균의 리보솜만을 표적으로 함
② 사이클로헥시미드(cycloheximide) : 진핵세포에서 작용. 진핵세포의 번역 억제제
③ 클로람페니콜(chloramphenicol) : 원핵세포에서 작용. 진정세균의 리보솜만을 표적으로 함
문제 4. 단백질을 분리하기 위하여 크로마토그래피 방법을 사용할 수 있다. 다음 크로마토그래피 방법에 대하여 수지(resin)의 성질, 기본 원리, 단백질 용출 방법을 각각 설명하시오.
⑴ 4-1. 이온 교환 크로마토그래피(ion-exchange chromatography)
○ 정의 : bead에 표적 물질(예 : 단백질)을 붙인 후 크로마토그래피를 하는 것
○ 음이온 크로마토그래피 혹은 양이온 교환수지(positive ion exchange resin) 크로마토그래피
○ 음전하를 띤 bead를 이용
○ 표면순양전하를 띤 물질을 분리하기 위한 목적 (예 : 등전점이 높은 단백질)
○ 음전하일수록 더 빨리 용출됨
○ 양이온 크로마토그래피 혹은 음이온 교환수지(negative ion exchange resin) 크로마토그래피
○ 양전하를 띤 bead를 이용
○ 표면순음전하를 띤 물질을 분리하기 위한 목적 (예 : 등전점이 낮은 단백질)
○ 양전하일수록 더 빨리 용출됨
⑵ 4-2. 겔 여과 크로마토그래피(gel filtration chromatography)
○ 작은 분자만 통과할 수 있는 거름망을 사용 : 큰 분자일수록 전개 속도가 빠름
○ 시료의 분자량 조건 : 1.2 × 102 ~ 1.1 × 106
○ 예 : PD-10 desalting columnm, Sephacryl S300 column chromatography, 기타 크기 배제 크로마토그래피
⑶ 4-3. 친화성 크로마토그래피(affinity chromatography)
○ 충전제와의 친화력 차이를 이용하여 표적물질 분리, 가장 분리가 잘 이루어짐
○ 예 1. His-tag 크로마토그래피
○ 구조 : 〈Ni2+〉 - 〈His 및 단백질 X〉 - 〈단백질 Y〉
○ Ni2+와 His 간 배위결합 : 형성상수 K가 굉장히 큼
○ 예 2. 글루타티온(glutathione)-GST
○ 구조 : 〈글루타티온〉 - 〈GST 및 단백질 X〉 - 〈단백질 Y〉
○ 글루타티온과 GST 간 배위결합 : 형성상수 K가 굉장히 큼
○ 예 3. 비오틴(biotin)-스트렙토아비딘(streptavidin)
○ 구조 : 〈비오틴〉 - 〈스트렙토아비딘 및 단백질 X〉 - 〈단백질 Y〉
○ 비오틴과 스트렙토아비딘 간 배위결합 : 형성상수 K가 굉장히 큼
○ 스트렙토아비딘 대신 아비딘을 사용할 수도 있음
○ DAB immuno-histochemical technique에서 활용
○ 예 4. Diels-Alder reaction
○ 예 5. EDC/NHS - 카르복실산 클릭 반응
○ 예 6. thiol - maleimide 클릭 반응
입력: 2023.11.10 00:30
'▶ 자연과학 > ▷ 변리사 풀이' 카테고리의 다른 글
| 【유기화학】 2023년 제60회 변리사 2차 국가자격시험 (1) | 2023.10.21 |
|---|---|
| 【분자생물학】 2018년 제55회 변리사 2차 국가자격시험 (7) | 2023.04.21 |
| 【분자생물학】 2021년 제58회 변리사 2차 국가자격시험 (0) | 2022.12.06 |
| 【분자생물학】 2022년 제59회 변리사 2차 국가자격시험 (0) | 2022.12.06 |
| 【유기화학】 2022년 제59회 변리사 2차 국가자격시험 (8) | 2022.09.18 |





최근댓글