2022년 제59회 변리사 2차 국가자격시험
추천글 : 【분자생물학】 변리사 2차 분자생물학 기출문제 풀이
a. 중심학설
b. DNA 테크놀로지
문제 1. 단백질 합성과정인 번역(translation)은 단백질과 RNA의 복합체인 리보솜(ribosome)에서 이루어진다. 다음 물음에 답하시오.
⑴ 1-1. 번역 과정 중 리보솜 내에 존재하는 세 종류 RNA의 상대적 위치 및 각각의 역할에 대해 설명하시오.
○ rRNA
○ 진핵생물 기준, RNA pol I 에 의해 인에서 전사됨
○ rRNA 전사를 위한 프로모터는 유전자 상류 멀찍이 존재
○ 리보솜 대단위체 및 소단위체의 주성분으로 세포질에서 단백질 번역 기구를 구성함
○ mRNA
○ 진핵생물 기준, RNA pol II에 의해 핵질에서 전사되어 핵공을 통해 세포질로 이동
○ mRNA 전사를 위한 프로모터는 유전자 상류 가까이에 존재
○ 유전자를 암호화하는 염기서열을 가지고 있음
○ 3개씩 하나의 아미노산을 지정하는데 이를 코돈(codon)이라고 함
○ tRNA
○ 진핵생물 기준, RNA pol III에 의해 핵질에서 전사되어 세포질에서 번역됨
○ tRNA를 위한 프로모터는 유전자 하류 가까이에 존재
○ mRNA를 통해 폴리펩티드를 번역할 때 아미노산을 수송함
○ mRNA의 코돈과 상보적인 결합을 할 수 있는 안티코돈(anti-codon)이 있음
⑵ 1-2. 아미노산 사이의 결합을 촉매하는 리보솜 내 펩티딜 전이효소(peptidyltransferase) 활성에 대하여 설명하고, 아래의 두 아미노산 세린(Serine; Ser)과 알라닌(Alanine; Ala)이 결합한 Ser-Ala 디펩티드(dipeptide) 구조를 그리시오.

○ mRNA, 리보솜으로 구성된 번역 개시복합체에는 A site, P site, E site가 있고, 이때 P site의 펩티드 말단의 카르복실기가 A site의 아미노산의 아미노기와 펩티드 결합을 형성함 : 아미노기의 질소가 카르복실기의 카르보닐 탄소를 친핵체 공격
○ 그 결과 P site의 아미노산 사슬이 전부 A site의 tRNA에 매달리게 됨
○ 이러한 펩티드 결합은 리보솜 대단위체의 펩티드 전이효소 활성에 의해 이루어짐
○ Ser-Ala 디펩티드 구조
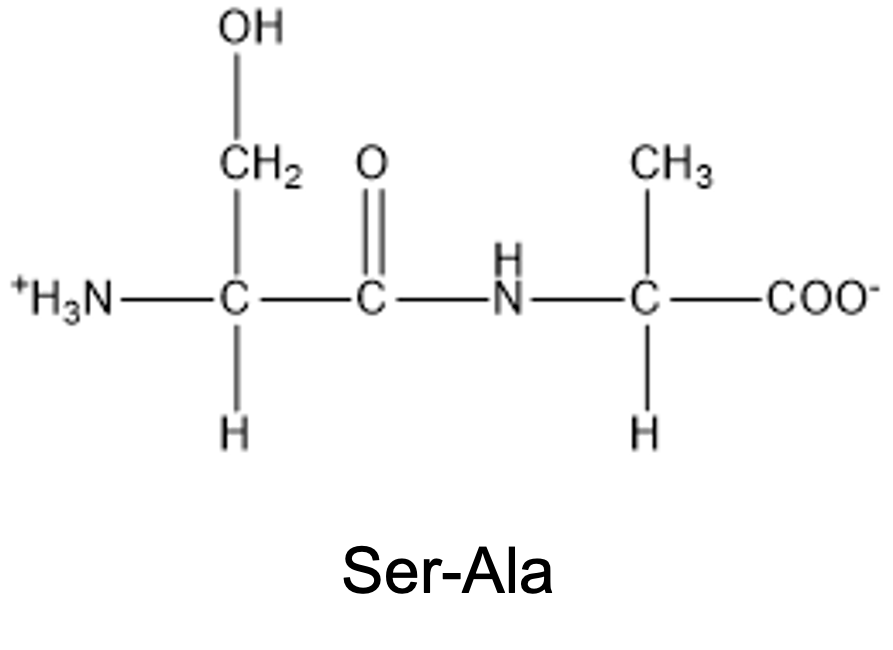
⑶ 1-3. 진핵생물과는 달리 원핵생물 유전자의 번역은 전사약화(attenuation 혹은 전사감쇠) 현상에 의해 조절되기도 한다(예, trp 오페론). 그 원인과 작용 기작을 설명하시오.
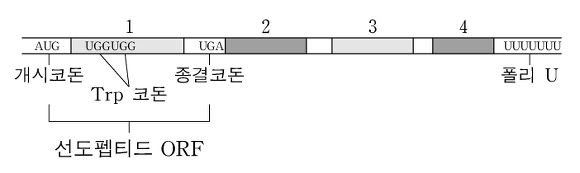
○ 원인 : 트립토판 오페론은 상시 발현하고, 오페론 중에서도 전사 활성이 높음. 실제로 이러한 특성을 이용해 DNA technology에서 자주 활용되고 있음. 트립토판 오페론 또한 젓당 오페론처럼 음성적 조절 기작이 있으나 이것으로는 충분하지 않기 때문에 자체적인 감쇄 조절기작으로 추가적인 음성적 조절 기능을 꾀함
○ 배경설명 1. 트립토판 오페론은 트립토판 효소를 합성하는 오페론이고, 이보다 먼저 선도펩티드가 만들어짐
○ 배경설명 2. 트립토판 오페론의 선도펩티드 mRNA 서열은 1, 2, 3, 4로 구분할 수 있고 2-3 헤어핀과 3-4 헤어핀이 생길 수 있음
○ 트립토판이 부족한 경우 : RNA pol에 의해 RNA가 전사되어도 트립토판 tRNA에 의한 번역속도가 느려서 리보솜이 1번에 있음 → 2-3 헤어핀이 생김 → 오페론을 따라 RNA 위를 움직이고 있는 RNA pol이 헤어핀에 의해 잡아당겨지지 않아 전사는 계속 일어남 → 선도펩티드 RNA 이후에 있는 트립토판 효소 RNA가 전사됨
○ 트립토판이 풍부한 경우 : RNA pol에 의해 RNA가 전사됐을 때 트립토판 tRNA에 의한 번역속도가 빨라서 리보솜이 벌써 1-2 서열에 있음 → 3-4 헤어핀이 생김 → 오페론을 따라 RNA 위를 움직이고 있는 RNA pol이 헤어핀에 의해 잡아당겨져서 전사가 중단됨 → 선도펩티드 RNA 이후에 있는 트립토판 효소 RNA는 전사되지 못함
○ 트립토판이 전무한 경우 : RNA pol에 의해 RNA가 전사됐을 때 트립토판 부분을 리보솜이 통과하지 못해서 리보솜이 1번에도 와 있지 않음 → 3-4 헤어핀이 생김 → 오페론을 따라 RNA 위를 움직이고 있는 RNA pol이 헤어핀에 의해 잡아당겨져서 전사가 중단됨 → 선도펩티드 RNA 이후에 있는 트립토판 효소 RNA는 전사되지 못함
문제 2. 진핵생물의 염색질(chromatin)은 DNA와 단백질의 복합체로 구성되어 있다. 다음 물음에 답하시오.
⑴ 2-1. 염색질 구성의 기본 단위인 뉴클레오솜(nucleosome)의 구조에 대하여 설명하시오.
○ DNA와 히스톤 단백질이 결합하여 약 10 nm의 섬유(bead-on-string)를 구성한 것
○ 146 bp DNA가 히스톤 8량체(즉, H2A, H2B, H3, H4가 한 쌍씩 존재)를 1.65 바퀴 회전
○ 링커 : DNA : H1 ~ H까지를 지칭. 200 뉴클레오티드
○ 이때 히스톤의 아미노산 중 20% 이상이 양전하 염기성 아미노산이므로 음전하의 DNA와 이온 결합으로 붙어 있음
⑵ 2-2. 유전자 발현 및 염색질 구조에 영향을 미치는 주요 후성유전학적 변형(eptigenetic modification)
○ 화학적 변형 : 당화, 인산화, 이황화결합, 프레닐화, 유비퀴틴화, 아세틸화, 메틸화, 나이트로화, 아민화, 포르밀화, 팔미토일화, 히드록실화, 황화, SUMOylation, lipid anchoring 등
○ 샤페론 단백질
○ 자이모겐
○ 수명 조절
⑶ 2-3. 염색질은 유전자 발현 양상에 따라 진정염색질(euchromatin)과 이질염색질(heterochromatin)로 나뉜다. 진정염색질과 이질염색질의 특성을 비교∙구분하여 설명하시오.
○ 진정염색질
○ 응축이 안 된 염색질로 발현 정도가 큼
○ 아세틸화, 탈메틸화와 같은 히스톤 N 말단 수식이 주된 특징
○ 이질염색질
○ 응축된 염색질로 발현 정도가 낮음
○ 일반적으로 염색체의 동원체, 텔로미어가 이질염색체 형태로 존재
○ 탈아세틸화, 메틸화와 같은 히스톤 N 말단 수식이 주된 특징
○ 단, 인산화 등의 히스톤 N 말단 수식은 상황 별로 이질 / 진정 여부가 상이함
○ 메틸화, 아세틸화라고 하여 이질 / 진정 여부가 일정한 것도 아님
| Type | Histone | ||||||
| H3K4 | H3K9 | H3K14 | H3K27 | H3K79 | H4K20 | H2BK5 | |
| mono- methylation |
activation | activation | activation | activation | activation | activation | |
| di- methylation |
repression | repression | activation | ||||
| tri- methylation |
activation | repression | repression | activation, repression |
repression | ||
| acetylation | activation | activation | activation | ||||
Table. 1. 히스톤 수식에 따른 활성 / 억제
예를 들어, H3K27은 H3 히스톤의 27번째 K 아미노산 (리신, Lys)을 의미함
문제 3. 1960년대 일본 과학자인 Reiji Okazaki에 의해 반불연속 DNA 복제 모델(semidiscontinuous model of replication)이 제안되었다. 다음 물음에 답하시오.
⑴ 3-1. 오카자키 절편(Okazaki fragment)을 생성할 때 지체가닥(lagging strand) 합성에 요구되는 효소들인 프리메이스(primase), DNA 중합효소 I (DNA polymerase I), DNA 중합효소 III (DNA polymerase III), DNA 연결효소(DNA ligase)의 역할에 대하여 각각 설명하시오.
○ 프리메이스(primase) : 프라이머 하벙 및 부착에 관여. 프라이머는 대략 10 nt 정도의 크기인 짧은 RNA 단편
○ DNA 중합효소 I (DNA polymerase I) : 프라이머 제거 및 RNA 프라이머 자리를 dNTP로 교체
○ DNA 중합효소 III (DNA polymerase III) : DNA 신장. 이합체로 작용하며 실제로는 자기가 이동하지 않고 DNA 가닥이 움직임
○ DNA 연결효소(DNA ligase) : RNA 프라이머가 제거되면 DNA 연결효소(ligase, DnaG)가 DNA 사슬 연결반응을 수행. ATP 사용
⑵ 3-2. 나선풀림효소(helicase)의 기능을 설명하고 복제 분기점(replication fork)에서 이 효소의 활성이 필요한 이유를 설명하시오.
○ 기능 : 헬리케이스는 DnaA 혹은 ORC가 생성한 복제기포가 있어야 DNA에 결합하며, 결합 시 두 상보적 가닥의 수소결합을 풀어줌
○ 원핵생물의 헬리케이스의 종류 : DnaB, PcrA
○ 진핵생물의 헬리케이스의 종류 : McM (minichromosome maintenance complex)
○ 특히 복제 분기점에서 헬리케이스가 활성화돼야 하는 이유 : 헬리케이스 특성 실험에 따르면, 헬리케이스는 DNA 복제 분기점에서 지연가닥의 주형에 결합하고 5' → 3'으로 이동함 (단, 모든 생물이 이런 것은 아님)
⑶ 3-3. DNA 중합효소 III 전효소(DNA polymerase III holoenzyme) 중 α-소단위체(α-subunit)와 ε-소단위체(ε-subunit) 역할을 설명하시오.
○ 전체 구조 : αεθβ2τ2 - αεθβ2γ2(δδ'χψ)2
○ α-소단위체 : polymerase
○ ε-소단위체 : 3' → 5' exonuclease
문제 4. 중합효소연쇄반응(polymerase chain reaction, PCR)은 DNA 또는 mRNA의 특정 영역을 대량으로 증폭하는 유용한 기술이다. 다음 물음에 답하시오.
⑴ 4-1. PCR 방법을 통하여 mRNA를 증폭하는 과정을 설명하시오.
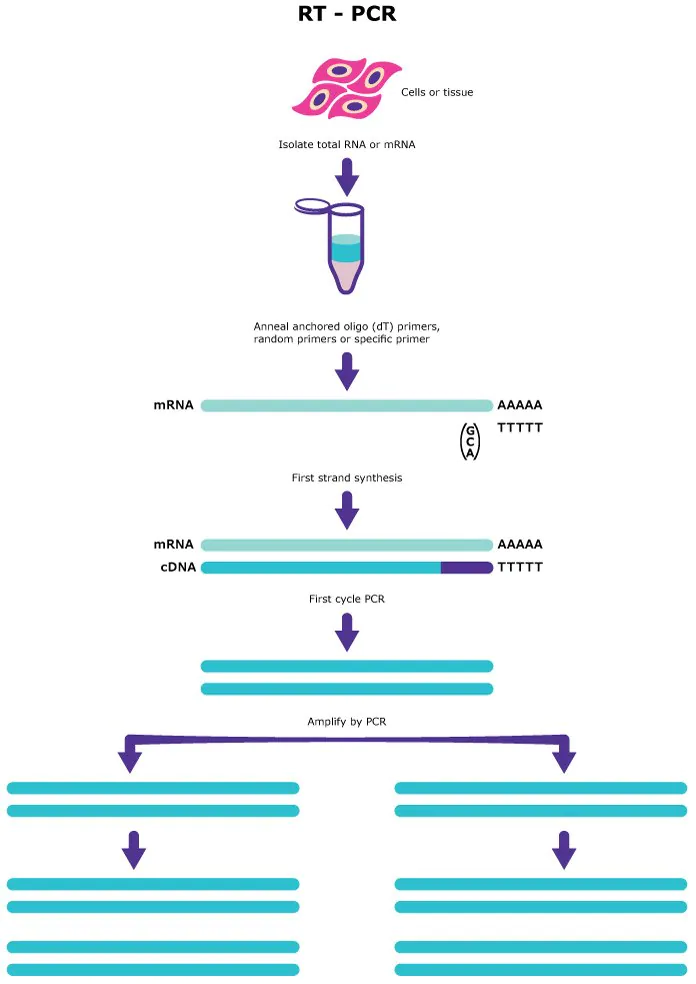
○ 1단계. mRNA 용액에 DNA 프라이머를 첨가하고 역전사 효소(reverse transcriptase)를 1회 처리
○ oligo dT 프라이머 : 진핵생물 유래 mRNA에는 꼭 있는 poly A를 주형으로 완전한 cDNA를 합성할 수 있음
○ random 프라이즈 (주로 hexomer) : 다양한 cDNA 풀을 형성할 수 있음
○ 2단계. 일반 PCR처럼 DNA 복제 원리를 이용하여 cDNA를 증폭시킴
○ reverse-transcriptase PCR (RT-PCR)이라고 함
⑵ 4-2. PCR의 연쇄반응을 수행할 때 순환 과정(cycle)은 변성(denature), 결합(annealing), 신장(elongation)의 3단계로 이루어져 있다. 각 단계에서 일어나는 과정을 설명하시오.
○ 변성 : DNA 두 가닥을 분리하기 위해 가열하는 단계. 94 ℃. 30 ~ 60초
○ 결합 : 프라이브 부착 단계. 혼합물이 식음에 따라 프라이머들이 DNA 주형에 결합. 50 ~ 55 ℃. 1분
○ 신장 : DNA 중합 단계. 중합효소는 프라이머에서 합성을 개시. 72 ℃. 1000 bp 당 1분 정도의 시간
⑶ 4-3. 다음 354 nt의 크기를 가진 유전자의 PCR 증폭을 위하여 밑줄 친 ⓐ와 ⓑ 부분을 이용하여 primer를 만들려고 한다. ⓐ primer와 ⓑ primer의 방향성(5'과 3')을 포함하여 primer를 제작하시오. (단, primer의 Tm 값은 고려하지 않는다.)
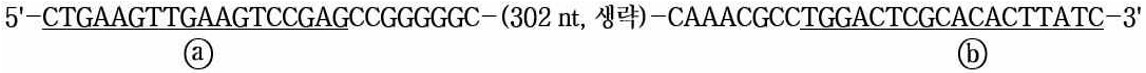
○ 1단계
○ 5'-CTGAAGTTGAAGTCCGAGCCGGGGGC-∙∙∙-CAAACGCCTGGACTCGCACACTTATC-3' (354 nt)
○ 3'-GACTTCAACTTCAGGCTCGGCCCCCG-∙∙∙-GTTTGCGGACCTGAGCGTGTGAATAG-5'
○ 2단계
○ 5'-CTGAAGTTGAAGTCCGAGCCGGGGGC-∙∙∙-CAAACGCCTGGACTCGCACACTTATC-3'
○ 3'-GACTTCAACTTCAGGCTCGGCCCCCG-∙∙∙-GTTTGCGGACCTGAGCGTGTGAATAG-5' (프라이머)
○ 5'-CTGAAGTTGAAGTCCGAGCCGGGGGC-∙∙∙-CAAACGCCTGGACTCGCACACTTATC-5' (프라이머)
○ 3'-GACTTCAACTTCAGGCTCGGCCCCCG-∙∙∙-GTTTGCGGACCTGAGCGTGTGAATAG-5'
○ 3단계
○ 5'-CTGAAGTTGAAGTCCGAGCCGGGGGC-∙∙∙-CAAACGCCTGGACTCGCACACTTATC-3'
○ 3'-GACTTCAACTTCAGGCTCGGCCCCCG-∙∙∙-GTTTGCGGACCTGAGCGTGTGAATAG-5' (프라이머)
○ 3'-GACTTCAACTTCAGGCTCGGCCCCCG-∙∙∙-GTTTGCGGACCTGAGCGTGTGAATAG-5'
○ 5'-CTGAAGTTGAAGTCCGAGCCGGGGGC-∙∙∙-CAAACGCCTGGACTCGCACACTTATC-5' (프라이머)
○ 5'-CTGAAGTTGAAGTCCGAGCCGGGGGC-∙∙∙-CAAACGCCTGGACTCGCACACTTATC-5'
○ 3'-GACTTCAACTTCAGGCTCGGCCCCCG-∙∙∙-GTTTGCGGACCTGAGCGTGTGAATAG-5' (프라이머)
○ 3'-GACTTCAACTTCAGGCTCGGCCCCCG-∙∙∙-GTTTGCGGACCTGAGCGTGTGAATAG-5'
○ 5'-CTGAAGTTGAAGTCCGAGCCGGGGGC-∙∙∙-CAAACGCCTGGACTCGCACACTTATC-3' (프라이머)
○ 제작해야 하는 프라이머 (DNA 프라이머)
○ 5'-CTGAAGTTGAAGTCCGAG-3'
○ 5'-GATAAGTGTGCGAGTCCA-3'
입력: 2022.12.06 21:10
'▶ 자연과학 > ▷ 변리사 풀이' 카테고리의 다른 글
| 【분자생물학】 2018년 제55회 변리사 2차 국가자격시험 (7) | 2023.04.21 |
|---|---|
| 【분자생물학】 2021년 제58회 변리사 2차 국가자격시험 (0) | 2022.12.06 |
| 【유기화학】 2022년 제59회 변리사 2차 국가자격시험 (8) | 2022.09.18 |
| 【유기화학】 2014년 제51회 변리사 2차 국가자격시험 (17) | 2022.09.10 |
| 【유기화학】 2021년 제58회 변리사 2차 국가자격시험 (9) | 2021.08.08 |




최근댓글