20강. 오토인코더(autoencoder)
추천글 : 【알고리즘】 알고리즘 목차
1. 개요 [본문]
2. 종류 1. 가변 오토인코더 [본문]
3. 종류 2. ZINB 오토인코더 [본문]
4. 종류 3. matrix factorization [본문]
1. 개요 [목차]
⑴ 정의 : 입력 데이터를 최대한 압축시킨 후, 압축된 데이터를 다시 본래의 형태로 복원시키는 신경망
① 인코더(encoder) : 입력층에서부터 은닉층으로의 인공 신경망. 인지 네트워크라고도 함
○ 인지 네트워크(recognition network) : 패턴을 감지하고 의미를 부여하는 특수화된 네트워크
② 디코더(decoder) : 은닉층에서부터 출력층으로의 인공 신경망. 생성 네트워크라고도 함
○ 생성 네트워크(generative network) : 입력 데이터와 매우 유사한 새로운 데이터를 생성할 수 있는 네트워크
③ 인코더와 디코더가 함께 트레이닝 됨
Figure. 1. 오토인코더의 구조
⑵ 수식화
Z = Eθ(X), X̂ = Dϕ(Z)
① Z : latent variable
② Eθ : 인코더 네트워크
③ X̂ : 재구성된 입력
④ Dϕ : 디코더 네트워크
⑶ 특징
① 비지도 학습 신경망
② 인코더는 차원 축소 역할을 수행
③ 디코더는 생성 모델의 역할을 수행
④ 입력층의 노드 개수는 출력층의 노드 개수와 동일
⑤ 은닉층의 노드 개수는 입력층의 노드 개수보다 작음
⑥ 차원 축소 알고리즘도 오토인코더의 한 예시라고 할 수 있음
⑷ 효과
① 효과 1. 데이터 압축 : 은닉층이 입력층보다 더 작은 개수의 노드를 가지면, 이 네트워크는 입력 데이터를 압축할 수 있음
② 효과 2. 데이터 추상화 : 복잡한 데이터를 다차원 벡터으로 변환하여 입력 데이터 분류 혹은 재조합을 가능케 함
○ 이러한 다차원 벡터를 은닉층, 피처(feature)라고도 함
○ 재조합 알고리즘으로는 대표적으로 PCA 알고리즘이 있음
③ 효과 3. 은닉 변수 추론 : 입력 데이터를 구성하는 prior를 latent variable로 두면, 오토인코더는 이를 추론하는 기능을 함
○ 디콘볼루션(deconvolution)이라고도 함
2. 종류 1. 가변 오토인코더(VAE, variational autoencoder) [목차]
⑴ 구성
① 인코더 : 입력 데이터를 은닉층에서의 확률분포, 즉 parametrized distribution으로 맵핑함
② 디코더 : parameterized distribution을 Bayesian inference에서의 prior로 보고 출력층을 맵핑함
○ 학습 : parametric posterior와 true posterior 간의 차이를 비용함수를 통해 최소화
③ 오토인코더와의 차이 : 오토인코더는 결정론적인 데 반해, 가변 오토인코더는 확률론적
④ latent 매칭을 자연스럽게 하는 것은 오토인코더보다 가변 오토인코드가 더 좋음
⑵ 수식화
Z ~ qθ(Z | X), X̂ = Dϕ(Z)
① Z : latent variable
② qθ : 조건부 확률분포
③ X̂ : 재구성된 입력
3. 종류 2. ZINB 오토인코더(zero-inflated negative binomial autoencoder) [목차]
추후 업데이트
4. 종류 3. matrix factorization [목차]
⑴ 정의 : 알고 있는 A 행렬을 W 행렬과 H 행렬의 곱으로 분해하는 알고리즘. A ~ W × H
① A 행렬 : 샘플-특성을 나타내는 행렬. 샘플로부터 알 수 있음
② H 행렬 : 변수-특성을 나타내는 행렬
③ K means clustering, PCA 알고리즘과 유사함
④ 오토인코더는 non-linear transformation을 포함하므로 matrix factorization보다 넓은 개념
⑤ 다음 알고리즘들은 least square method에 기반을 두고 있지만, gradient descent method 등을 활용할 수도 있음
⑵ 알고리즘 : R = UV, R ∈ ℝ5×4, U ∈ ℝ5×2, V ∈ ℝ2×4인 U, V를 탐색
import numpy as np
R = np.outer([3,1,4,2.,3],[1,1,0,1]) + np.outer([1,3,2,1,2],[1,1,4,1])
U=np.random.rand(5,2)
V=np.random.rand(2,4)
for i in range(3):
V,_,_,_=np.linalg.lstsq(U, R, rcond=None)
Ut,_,_,_=np.linalg.lstsq(V.T, R.T, rcond=None)
U=Ut.T
U@V # it is similar to R!
⑶ NMF(non-negative matrix factorization)
import numpy as np
R = np.outer([3,1,4,2.,3],[1,1,0,1]) + np.outer([1,3,2,1,2],[1,1,4,1])
U=np.random.rand(5,2)
V=np.random.rand(2,4)
for i in range(20):
V,_,_,_=np.linalg.lstsq(U, R, rcond=None)
V = np.where(V >= 0, V, 0) #set negative entries equal zero
Ut,_,_,_=np.linalg.lstsq(V.T, R.T, rcond=None)
U=Ut.T
U = np.where(U >= 0, U, 0) #set negative entries equal zero
U@V # it is similar to R!
⑷ matrix completion (Netflix 알고리즘) : 마스킹된 R에 대하여 matrix factorization을 진행하는 것
import numpy as np
R = np.outer([3,1,4,2.,3],[1,1,0,1]) + np.outer([1,3,2,1,2],[1,1,4,1])
mask=np.array([[1.,1,1,1],
[1,0,0,0],
[1,0,0,0],
[1,0,0,0],
[1,0,0,0]])
U=np.random.rand(5,2)
V=np.random.rand(2,4)
RR = U@V
RR = RR*(1-mask)+R*mask
for i in range(20):
V,_,_,_=np.linalg.lstsq(U, R, rcond=None)
V = np.where(V >= 0, V, 0)
Ut,_,_,_=np.linalg.lstsq(V.T, R.T, rcond=None)
U=Ut.T
U = np.where(U >= 0, U, 0)
RR = U@V
RR = RR*(1-mask)+R*mask
RR*(1-mask)+R*mask # it is similar to R!
⑸ SVD(singular value decomposition)
⑹ 응용 1. cell type classification
① 조직으로부터 얻은 scRNA-seq로부터 cell type proportion을 얻기 위한 목적
② cell type heterogeneity에 의한 confounding effect를 줄이는 게 중요
③ 1-1. constrained linear regression
④ 1-2. reference-based approach
○ 1-2-1. CIBERSORT(cell-type identification by estimating relative subsets of RNA transcript) : 샘플별로 cell type proportion, p value를 확인할 수 있음
⑺ 응용 2. joint NMF : 멀티오믹스까지 확장하게 함
⑻ 응용 3. metagene 추출
⑼ 응용 4. Starfysh : 다음은 공간전사체에서 archetype을 추론하고, 각 archetype을 대표하는 anchor를 결정하는 알고리즘
① 단계 1. 오토인코더 구성
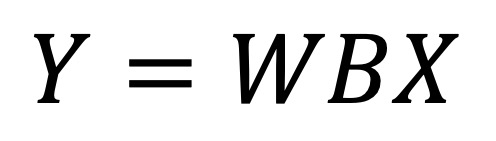
○ X ∈ ℝS×G : 입력 데이터 (스팟 × 유전자)
○ D : archetype의 수
○ B ∈ ℝD×S : 인코더. archetype을 추론하는 상황으로, 이때 각 archetype별로 전체 스팟에 대한 분포의 합은 1이어야 함
○ H = BX : latent variable
○ W ∈ ℝS×D : 디코더. 입력 데이터를 재구성하는 상황으로, 이때 각 스팟별로 전체 archetype 가중치의 합이 1이어야 함
○ Y = WBX : 재구성된 입력
② 단계 2. 최적화 알고리즘을 풀어 W, B를 계산
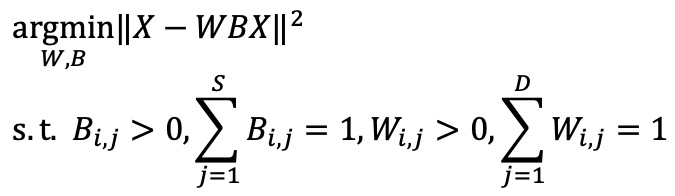
③ 단계 3. W 행렬에서 각 archetype에 대한 가중치가 가장 높은 스팟들이 anchor spot으로 선택됨
④ 단계 4. granularity 조정 : archetype 간의 거리가 가까운 경우 이를 병합하거나, 그 거리를 조정하는 계층적 구조를 사용
⑤ 단계 5. 각 anchor 별로 nearest spot을 탐색하여 archetypal community를 형성하고 마커 유전자를 탐색
⑥ 단계 6. signature gene set이 주어지는 경우, 기존의 gene set에 archetypal marker gene들을 추가하고 anchor를 다시 계산
○ 이때 stable marriage matching 알고리즘을 활용하여 각 archetype은 가장 유사한 signature와 대응됨
입력: 2023.06.27 00:55
수정: 2024.10.03 21:46
'▶ 자연과학 > ▷ 알고리즘·머신러닝' 카테고리의 다른 글
| 【알고리즘】 8강. 클러스터링 알고리즘 (0) | 2023.09.22 |
|---|---|
| 【알고리즘】 13강. 앙상블 학습 (0) | 2023.06.27 |
| 【알고리즘】 17강. RNN 알고리즘 (0) | 2023.06.27 |
| 【알고리즘】 21-1강. 프롬프트 엔지니어링 (0) | 2023.04.03 |
| 【알고리즘】 21강. 자연어 처리(NLP)와 거대 언어 모델(LLM) (0) | 2023.03.31 |




최근댓글