유학 합격 수기
추천글 : 【유학 입시】 2023-2024년도 유학 입시 및 장학금 정리
1. 유학 입시 사례 분석 [본문]
2. 유학 합격 수기 [본문]
3. 앞으로의 계획 [본문]
📃 2024년 9월 입학 기준, 미국 대학원 석∙박사과정
어제의 나에게 들려주고 싶은 이야기를 적어 놓다.
1. 유학 입시 사례 분석 [목차]
⑴ Stanford Bioengineering (■ biochem; Spatiotemporal Omics)
○ 지원 동기 : 외국인을 거의 뽑지 않는 biomedical informatics, biophysics나 fit이 좀 떨어지는 CS의 대안을 찾던 중 발견한 학과
○ 예상 합격률 10% : 워낙 경쟁력 있는 프로그램이고 protein engineering 쪽에 치우쳐 있어 less fit. 다만, 한국인을 많이 뽑는 학과
○ 실제 결과 : 불합격 (24.02.15)
⑵ Harvard Biophysics (■ imaging; Spatiotemporal Omics)
○ 지원 동기 : Xiaowei Zhuang 등의 교수님들이 있어 무조건 지원하기로 마음 먹은 학과
○ 예상 합격률 10% : 스탠포드와 비슷하나 모집단위는 더 작음. 다만, Harvard Faculty 한 명과 in-person contact을 했으므로 가산
○ 실제 결과 : 불합격 (24.03.07)
⑶ Michigan Bioinformatics (PIBS) (■ CS/BI; Spatial Bioinformatics)
○ 지원 동기 : bioinformatics를 검색해서 알게 된 학과. international을 거의 뽑지 않는 학과이지만, 암호론, 정보이론 등을 연구하시는 Minji Kim 교수님께 흥미가 있어 지원하기로 결심
○ 예상 합격률 0% : 모집 단위가 작은 편. PIBS에서 international을 5% 미만으로 뽑는다고 명시. international은 첫 1년을 자비 부담해야 한다고 함 (ref; 결과적으로는 랩이 학생을 펀딩하므로 틀린 정보)
○ 실제 결과 : 인터뷰 오퍼 (23.12.19) → 인터뷰 (24.02.01-02) → 합격 (24.02.09)
⑷ JHU Biomedical Engineering (■ CS/BI; Spatial Bioinformatics)
○ 지원 동기 : Jean Fan 등의 교수님들이 있어 무조건 지원하기로 마음 먹은 학과
○ 예상 합격률 5% : CS에 강하게 치중돼 있다는 인상. 사전 컨택이 실패한 이유에는 무언가 있지 않을까?
○ 실제 결과 : 불합격 (24.01.20)
⑸ UCLA Biochemistry, Molecular and Structural Biology (■ biochem; Spatiotemporal Omics)
○ 지원 동기 : Roy Wollman 교수님과 성공적인 컨택 후에 알게 된 학과. 순전히 교수님만 보고 지원하기로 결심
○ 예상 합격률 60% : admission committee head와 성공적인 사전 미팅. 다만, 생화학에 치우쳐 있어 less fit.
○ 실제 결과 : 인터뷰 오퍼 (23.12.14) → 인터뷰 (24.01.09-10) → 합격 (24.01.24)
⑹ UCSD Bioinformatics and Systems Biology (■ imaging; Spatiotemporal Omics)
○ 지원 동기 : bioinformatics를 검색해서 알게 된 학과이고 single-cell omics를 많이 하여 fit이 맞다고 생각함
○ 예상 합격률 30% : fit은 잘 맞음. GRE 등 quantitative한 측면을 많이 강조. 다만, 사전 컨택이 실패한 데엔 무언가 있지 않을까?
○ 실제 결과 : 인터뷰 오퍼 (23.12.22) → 인터뷰 (24.01.19-22) → 불합격 (24.02.28)
⑺ NYU Vilcek Institute of Graduate Biomedical Sciences (■ imaging; Spatiotemporal Omics)
○ 지원 동기 : Satija Rahul 교수님이 있는 NYU에 지원하려고 알아보던 중 분야가 넓어서 여러 포인트를 공략할 수 있는 프로그램을 발견
○ 예상 합격률 45% : 모집 단위도 상당히 크고 international 합격 비중도 높음. 분야도 넓어서 정량평가를 중요하게 보지 않을까? MRI 연구도 상당히 비중 있게 하는데 medical physics보다는 molecular biology 계열이라 fit도 맞아 보임
○ 실제 결과 : 불합격 (24.01.13)
⑻ Wisconsin-Madison Biomedical Data Science (■ CS/BI; Spatial Bioinformatics)
○ 지원 동기 : 지도교수 중 한 분이 강한 연고를 가지고 있어 지원을 결심. 이 프로그램은 최대 3개까지 한 fee로 해결되어 복수 지원을 생각
○ 예상 합격률 30% : fit도 괜찮아 보이고 그렇게 challenging한 프로그램은 아니라는 인상
○ 실제 결과 : 인터뷰 오퍼 (24.01.24) → 인터뷰 (24.01.31-02.08) → 불합격 (24.02.22)
⑼ Wisconsin-Madison Medical Physics (■ imaging; Spatiotemporal Omics)
○ 지원 동기 : 지도교수 중 한 분이 강한 연고를 가지고 있어 지원을 결심. 이 프로그램은 최대 3개까지 한 fee로 해결되어 복수 지원을 생각
○ 예상 합격률 7% : fit이 맞는 것은 아니지만 한 faculty와 강하게 연관돼 있어 가산. 내 MRI 연구도 좋게 보지 않을까?
○ 실제 결과 : 인터뷰 오퍼 (24.01.11) → 인터뷰 (24.02.09-22) → 합격 (24.02.17)
⑽ UIUC Computer Science (■ CS/BI; Spatial Bioinformatics)
○ 지원 동기 : CS와 핏도 잘 맞고, 커리어 패스도 유리하다고 생각. 암호론, 정보이론, 게임이론 등 이론에 무게중심이 잡혀 있어, CS 및 이론생물학에도 관심있는 필자에게 잘 맞을 수도 있겠다는 생각이 듦
○ 예상 합격률 10% : CS와도 fit은 맞는다고 생각하지만 높은 경쟁률을 고려
○ 실제 결과 : 불합격 (24.03.16)
⑾ Georgia Tech Bioinformatics in College of Computing (■ CS/BI; Sptial Bioinformatics)
○ 지원 동기 : 만나고 싶은 사람이 있는 곳
○ 예상 합격률 30% : 내가 CS에 꽤 잘 맞을지도 모른다는 의외의 깨닳음. 관련 실적도 압도적으로 많으니 해볼 만하지 않을까?
○ 실제 결과 : 불합격 (24.04.29)
⑿ Georgia Tech Bioinformatics in Biomedical Engineering (■ imaging; Spatiotemporal Omics)
○ 지원 동기 : 만나고 싶은 사람이 있는 곳
○ 예상 합격률 45% : GT CS보다도 덜 challenging 할 것 같은 인상. 심지어 공간생물정보학 관련 faculty도 좀 많은 것 같음
○ 실제 결과 : 인터뷰 오퍼 (24.01.20) → 인터뷰 (24.01.05, 03.08) →합격 (24.03.13)
⒀ WashU Physics (■ physics; Spatial Bioinformatics)
○ 지원 동기 : Mikhail Tikhonov 교수님과 성공적인 컨택 후에 알게 된 학과. 순전히 교수님만 보고 지원하기로 결심. 처음에는 웬 물리학과 하는 생각이 들었던 것도 사실. 하지만 내 커리어가 바이오 산업과는 맞지 않음을 알게 되었고 (∵ 바이오는 대부분 제약산업으로 귀결되기 때문), 이참에 머신러닝을 제대로 배울 수 있는 기회라고 판단.
For instance – personally, I think it's really cool how in computer science it is sometimes hard to "find hay in a haystack", or how high-dimensional geometry is very unintuitive, which is arguably the reason behind the incredible success of machine learning. What about you?
○ 예상 합격률 85% : WashU Physics는 일단 비인기 학과로 분류돼 있어 경쟁률이 낮은 편. 또한, faculty 연구 분야가 전부 제각각이라 faculty가 사전 컨택을 통해 미리 학생을 모집하는 듯한 인상을 받음. 물론, admission committee가 학생을 선발하지만, actively recruiting faculty의 영향을 무시할 수는 없을 것 (∵ 분야가 다 달라서). 성공적인 사전 컨택. 양호한 GRE Physics 점수 등등 기대되는 요인이 많음.
○ 실제 결과 : 불합격 (24.04.15)
⒁ UW Computer Science (■ CS/BI; Spatial Bioinformatics)
○ 지원 동기 : CS와 핏도 잘 맞고, 커리어 패스도 유리하다고 생각. digital pathology 쪽으로 강세라 공간생물학을 연구했던 필자와 핏이 맞는다고 판단
○ 예상 합격률 10% : CS와도 fit은 맞는다고 생각하지만 높은 경쟁률을 고려
○ 실제 결과 : 인터뷰 오퍼 (24.01.22) → 인터뷰 (24.01.23) → 불합격 (24.02.08)
⒂ UCSF Biological and Medical Informatics (■ imaging; Spatiotemporal Omics)
○ 지원 동기 : bioinformatics를 검색해서 알게 된 학과. international을 거의 뽑지 않는 학과이지만, 공간생물학 관련 교수님이 많아서 후회를 남기지 않으려고 지원
○ 예상 합격률 0% : 전체 박사과정 학생 55명 중 international이 고작 2명에 불과한 수준
○ 실제 결과 : 불합격 (23.12.22)
⒃ Tri-I Computational Biology and Medicine (■ CS/BI; Spatial Bioinformatics)
○ 지원 동기 : bioinformatics를 검색해서 알게 된 학과. 재단 선배들도 몇몇 계시고 international도 많은 편
○ 예상 합격률 35% : 공간생물정보학 관련 교수님들도 많고 실적도 많으니 합격률은 높을 것. 다만, 아이비리그라서 쉽지는 않을 듯.
○ 실제 결과 : 불합격 (24.03.08)
⒄ Gerstner Sloan Kettering Graduate School Cancer Engineering (■ imaging; Spatiotemporal Omics)
○ 지원 동기 : in-person 및 Zoom 사전 컨택 2회를 하게 되어 고민 끝에 지원하게 됨
○ 예상 합격률 35% : 분야가 완전히 맞는 건 아니지만 GRE Physics 점수도 송부했고, MRI에 대한 이해도 강해 뽑힐 가능성도 높음
○ 실제 결과 : 인터뷰 오퍼 (23.12.19) → 인터뷰 (24.01.16-18) → 합격 (24.02.02)
⒅ TSRI Computational Biology and Modeling (■ biochem; Spatiotemporal Omics)
○ 지원 동기 : 아직 정리되지 않은 미련. 그럼에도 필자가 여기를 선택할 가능성은 많이 낮지 않을까 생각
○ 예상 합격률 60% : 작년에도 합격 직전까지는 갔으므로 붙을 가능성이 꽤 있을 것으로 전망
○ 실제 결과 : 불합격 (24.02.02)
⒆ 2024-2025 유학 입시 분석
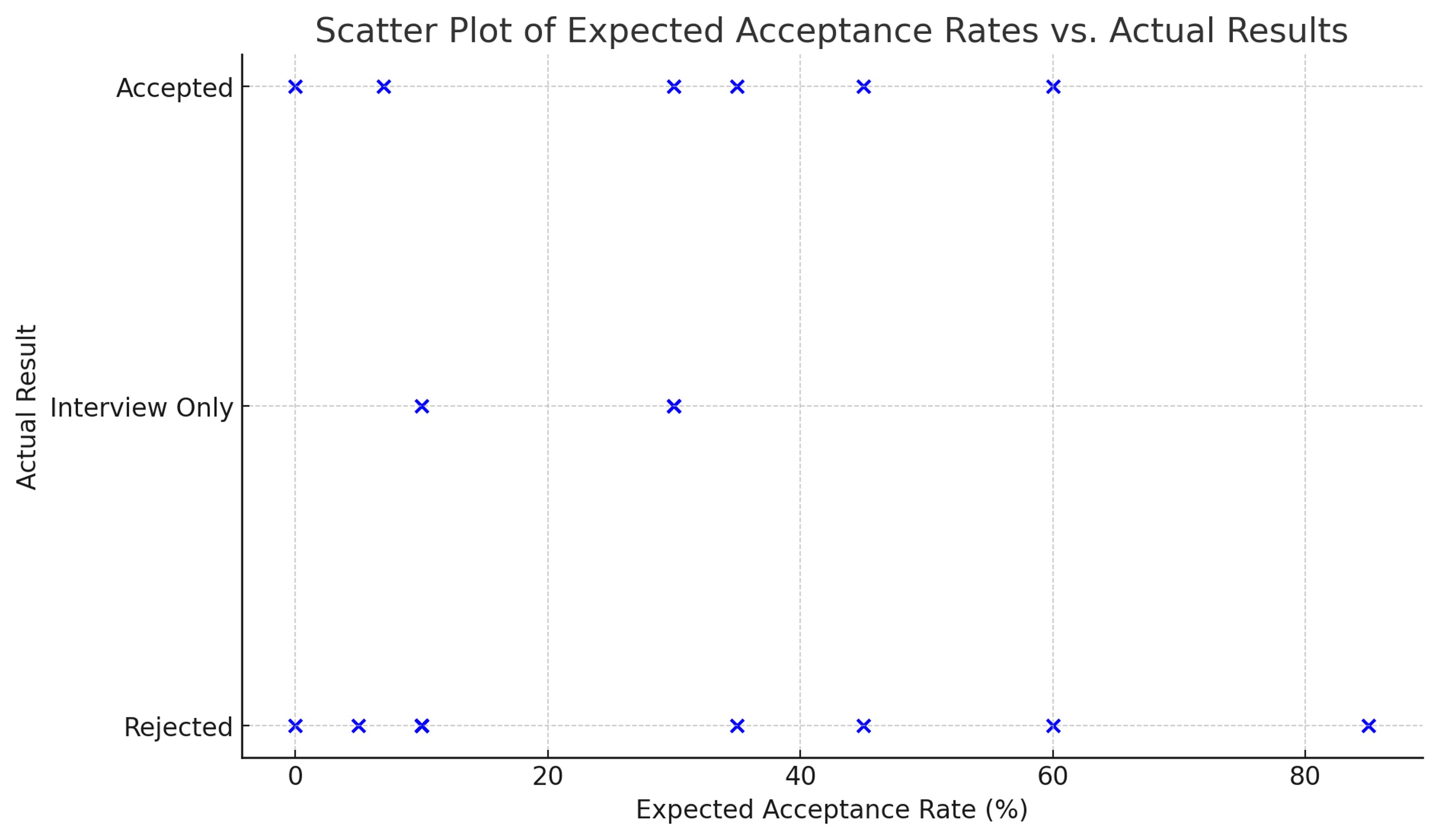
○ 예상 합격률과 실제 결과의 비교
import matplotlib.pyplot as plt
# Data mapping expected acceptance rates to actual outcomes (0: rejection, 0.5: interview only, 1: accepted)
data = {
"Stanford Bioengineering": (10, 0),
"Harvard Biophysics": (10, 0),
"Michigan Bioinformatics (PIBS)": (0, 1),
"JHU Biomedical Engineering": (5, 0),
"UCLA Biochemistry": (60, 1),
"UCSD Bioinformatics and Systems Biology": (30, 0.5),
"NYU Vilcek Institute": (45, 0),
"Wisconsin-Madison Biomedical Data Science": (30, 0.5),
"Wisconsin-Madison Medical Physics": (7, 1),
"UIUC Computer Science": (10, 0),
"Georgia Tech Bioinformatics (College of Computing)": (30, 1),
"Georgia Tech Bioinformatics (Biomedical Engineering)": (45, 1),
"WashU Physics": (85, 0),
"UW Computer Science": (10, 0.5),
"UCSF Biological and Medical Informatics": (0, 0),
"Tri-I Computational Biology and Medicine": (35, 0),
"Gerstner Sloan Kettering Cancer Engineering": (35, 1),
"TSRI Computational Biology and Modeling": (60, 0)
}
# Separating keys and values for plotting
expected_rates, actual_results = zip(*data.values())
# Recreating the scatter plot with updated y-axis label
plt.figure(figsize=(10, 6))
plt.scatter(expected_rates, actual_results, color='blue')
plt.title('Scatter Plot of Expected Acceptance Rates vs. Actual Results')
plt.xlabel('Expected Acceptance Rate (%)')
plt.ylabel('Actual Result')
plt.yticks([0, 0.5, 1], ['Rejected', 'Interview Only', 'Accepted'])
plt.grid(True)
plt.show()
○ 서류 탈락을 0, 인터뷰 오퍼만 있는 경우를 0.5, 최종 오퍼를 1로 스코어링
○ 회귀곡선 : y = (7.639e-05) x + (4.145e-01)
○ spearman rho = 0.02050878 → 거의 무시할만함
○ R2 (R-squared) = 0.001580409 (%) = 예측 가능한 영역 → 거의 무시할만함
○ 결론 : 실제 유학 입시 결과는 예측 결과와 완전히 다르게 나온다.
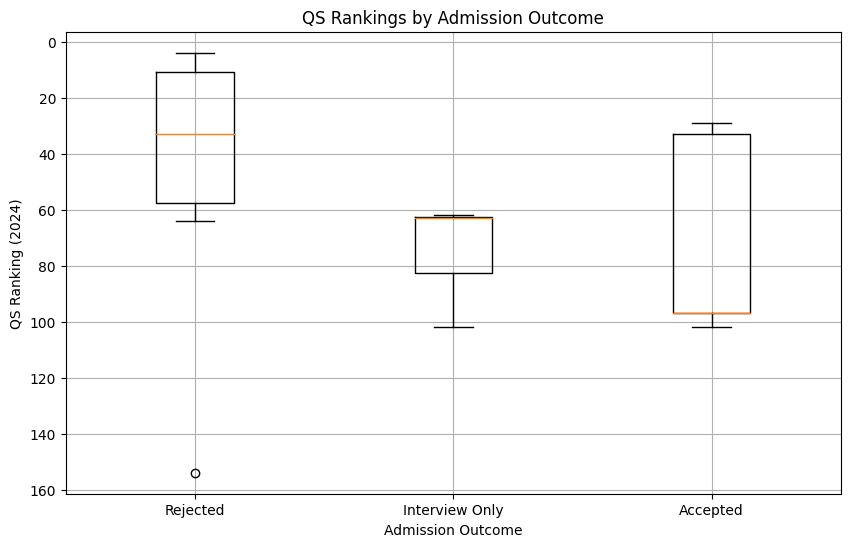
○ QS ranking 2024 별 합격률 추이
import matplotlib.pyplot as plt
import pandas as pd
# Data mapping expected acceptance rates to actual outcomes (0: rejection, 0.5: interview only, 1: accepted)
data = {
"Stanford Bioengineering": (10, 0),
"Harvard Biophysics": (10, 0),
"Michigan Bioinformatics (PIBS)": (0, 1),
"JHU Biomedical Engineering": (5, 0),
"UCLA Biochemistry": (60, 1),
"UCSD Bioinformatics and Systems Biology": (30, 0.5),
"NYU Vilcek Institute": (45, 0),
"Wisconsin-Madison Biomedical Data Science": (30, 0.5),
"Wisconsin-Madison Medical Physics": (7, 1),
"UIUC Computer Science": (10, 0),
"Georgia Tech Bioinformatics (College of Computing)": (30, 1),
"Georgia Tech Bioinformatics (Biomedical Engineering)": (45, 1),
"WashU Physics": (85, 0),
"UW Computer Science": (10, 0.5),
"UCSF Biological and Medical Informatics": (0, 0),
"Tri-I Computational Biology and Medicine": (35, 0),
"Gerstner Sloan Kettering Cancer Engineering": (35, 1),
"TSRI Computational Biology and Modeling": (60, 0)
}
# QS rankings for each institution
rankings = {
"Stanford Bioengineering": 5,
"Harvard Biophysics": 4,
"Michigan Bioinformatics (PIBS)": 33,
"JHU Biomedical Engineering": 28,
"UCLA Biochemistry": 29,
"UCSD Bioinformatics and Systems Biology": 62,
"NYU Vilcek Institute": 38,
"Wisconsin-Madison Biomedical Data Science": 102,
"Wisconsin-Madison Medical Physics": 102, # Same as above, duplicated intentionally
"UIUC Computer Science": 64,
"Georgia Tech Bioinformatics (College of Computing)": 97,
"Georgia Tech Bioinformatics (Biomedical Engineering)": 97, # Same as above, duplicated intentionally
"WashU Physics": 154,
"UW Computer Science": 63,
"UCSF Biological and Medical Informatics": None,
"Tri-I Computational Biology and Medicine": None,
"Gerstner Sloan Kettering Cancer Engineering": None,
"TSRI Computational Biology and Modeling": None
}
# Filtering out programs without a QS ranking
filtered_data = {key: (value[1], rankings[key]) for key, value in data.items() if rankings[key] is not None}
# Create a DataFrame from the filtered data
df_data = pd.DataFrame.from_dict(filtered_data, orient='index', columns=['Actual Result', 'QS Ranking'])
df_data.reset_index(inplace=True, drop=True)
# Group data by actual result for box plot
grouped_data = [df_data[df_data['Actual Result'] == outcome]['QS Ranking'].dropna() for outcome in [0, 0.5, 1]]
# Creating the box plot
plt.figure(figsize=(10, 6))
plt.boxplot(grouped_data, labels=['Rejected', 'Interview Only', 'Accepted'])
plt.title('QS Rankings by Admission Outcome')
plt.xlabel('Admission Outcome')
plt.ylabel('QS Ranking (2024)')
plt.gca().invert_yaxis() # Higher rankings are numerically lower
plt.grid(True)
plt.show()
○ 결론 : 중앙값만 보면 QS ranking이 낮을수록 합격을 많이 한 경향이 관찰됨
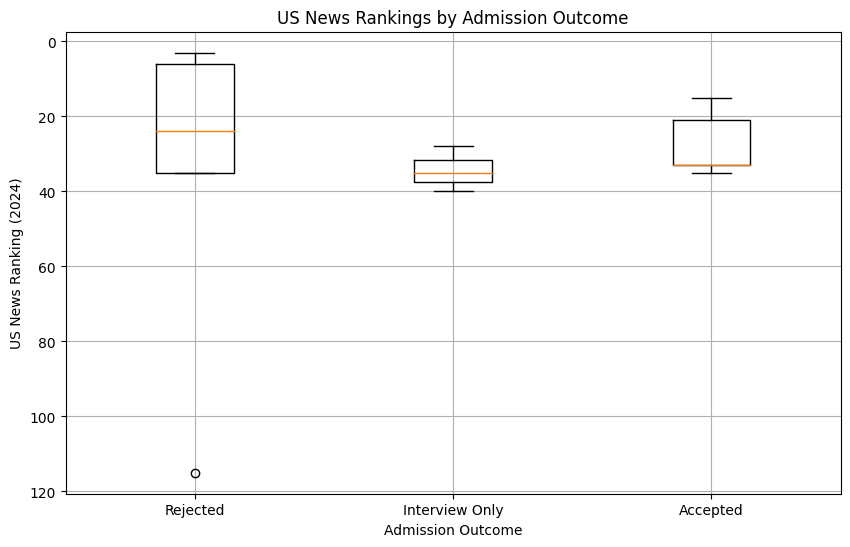
○ US News Ranking 2024 별 합격률 추이
import matplotlib.pyplot as plt
import pandas as pd
# Data mapping expected acceptance rates to actual outcomes (0: rejection, 0.5: interview only, 1: accepted)
data = {
"Stanford Bioengineering": (10, 0),
"Harvard Biophysics": (10, 0),
"Michigan Bioinformatics (PIBS)": (0, 1),
"JHU Biomedical Engineering": (5, 0),
"UCLA Biochemistry": (60, 1),
"UCSD Bioinformatics and Systems Biology": (30, 0.5),
"NYU Vilcek Institute": (45, 0),
"Wisconsin-Madison Biomedical Data Science": (30, 0.5),
"Wisconsin-Madison Medical Physics": (7, 1),
"UIUC Computer Science": (10, 0),
"Georgia Tech Bioinformatics (College of Computing)": (30, 1),
"Georgia Tech Bioinformatics (Biomedical Engineering)": (45, 1),
"WashU Physics": (85, 0),
"UW Computer Science": (10, 0.5),
"UCSF Biological and Medical Informatics": (0, 0),
"Tri-I Computational Biology and Medicine": (35, 0),
"Gerstner Sloan Kettering Cancer Engineering": (35, 1),
"TSRI Computational Biology and Modeling": (60, 0)
}
# US News rankings for each institution
rankings = {
"Stanford Bioengineering": 3,
"Harvard Biophysics": 3,
"Michigan Bioinformatics (PIBS)": 21,
"JHU Biomedical Engineering": 9,
"UCLA Biochemistry": 15,
"UCSD Bioinformatics and Systems Biology": 28,
"NYU Vilcek Institute": 35,
"Wisconsin-Madison Biomedical Data Science": 35,
"Wisconsin-Madison Medical Physics": 35, # Same as above, duplicated intentionally
"UIUC Computer Science": 35,
"Georgia Tech Bioinformatics (College of Computing)": 33,
"Georgia Tech Bioinformatics (Biomedical Engineering)": 33, # Same as above, duplicated intentionally
"WashU Physics": 24,
"UW Computer Science": 40,
"UCSF Biological and Medical Informatics": 115,
"Tri-I Computational Biology and Medicine": None,
"Gerstner Sloan Kettering Cancer Engineering": None,
"TSRI Computational Biology and Modeling": None
}
# Filtering out programs without a US News ranking
filtered_data = {key: (value[1], rankings[key]) for key, value in data.items() if rankings[key] is not None}
# Create a DataFrame from the filtered data
df_data = pd.DataFrame.from_dict(filtered_data, orient='index', columns=['Actual Result', 'US News Ranking'])
df_data.reset_index(inplace=True, drop=True)
# Group data by actual result for box plot
grouped_data = [df_data[df_data['Actual Result'] == outcome]['US News Ranking'].dropna() for outcome in [0, 0.5, 1]]
# Creating the box plot
plt.figure(figsize=(10, 6))
plt.boxplot(grouped_data, labels=['Rejected', 'Interview Only', 'Accepted'])
plt.title('US News Rankings by Admission Outcome')
plt.xlabel('Admission Outcome')
plt.ylabel('US News Ranking (2024)')
plt.gca().invert_yaxis() # Higher rankings are numerically lower
plt.grid(True)
plt.show()
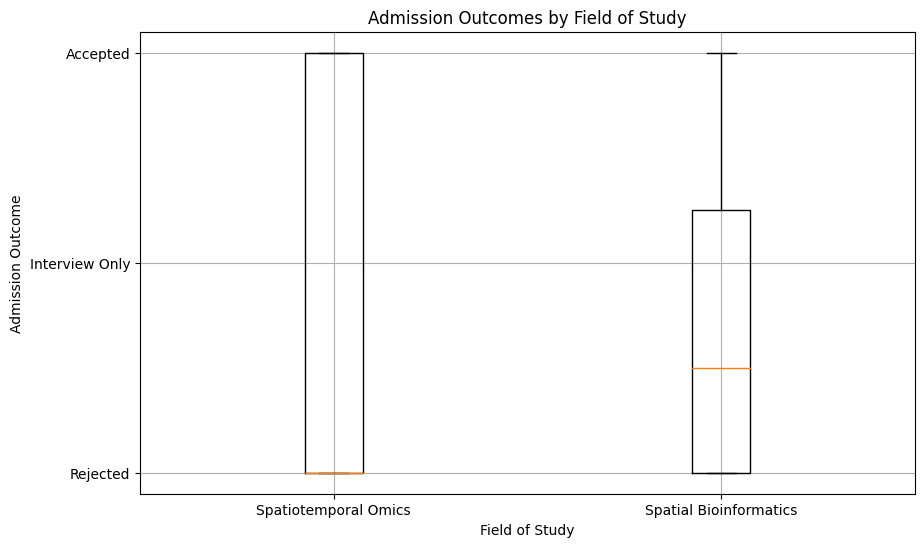
○ 연구 방향별 (spatiotemporal omics vs spatial bioinformatics) 합격률 추이
import matplotlib.pyplot as plt
import pandas as pd
# Data mapping expected acceptance rates to actual outcomes (0: rejection, 0.5: interview only, 1: accepted)
data = {
"Stanford Bioengineering": (10, 0, "Spatiotemporal Omics"),
"Harvard Biophysics": (10, 0, "Spatiotemporal Omics"),
"Michigan Bioinformatics (PIBS)": (0, 1, "Spatial Bioinformatics"),
"JHU Biomedical Engineering": (5, 0, "Spatial Bioinformatics"),
"UCLA Biochemistry": (60, 1, "Spatiotemporal Omics"),
"UCSD Bioinformatics and Systems Biology": (30, 0.5, "Spatiotemporal Omics"),
"NYU Vilcek Institute": (45, 0, "Spatiotemporal Omics"),
"Wisconsin-Madison Biomedical Data Science": (30, 0.5, "Spatial Bioinformatics"),
"Wisconsin-Madison Medical Physics": (7, 1, "Spatiotemporal Omics"),
"UIUC Computer Science": (10, 0, "Spatial Bioinformatics"),
"Georgia Tech Bioinformatics (College of Computing)": (30, 1, "Spatial Bioinformatics"),
"Georgia Tech Bioinformatics (Biomedical Engineering)": (45, 1, "Spatiotemporal Omic"),
"WashU Physics": (85, 0, "Spatial Bioinformatics"),
"UW Computer Science": (10, 0.5, "Spatial Bioinformatics"),
"UCSF Biological and Medical Informatics": (0, 0, "Spatiotemporal Omics"),
"Tri-I Computational Biology and Medicine": (35, 0, "Spatial Bioinformatics"),
"Gerstner Sloan Kettering Cancer Engineering": (35, 1, "Spatiotemporal Omics"),
"TSRI Computational Biology and Modeling": (60, 0, "Spatiotemporal Omics")
}
# Create a DataFrame from the data
df_data = pd.DataFrame.from_dict(data, orient='index', columns=['Expected Rate', 'Actual Result', 'Field'])
df_data.reset_index(inplace=True, drop=True)
# Group data by field for box plot
grouped_omics = df_data[df_data['Field'] == "Spatiotemporal Omics"]['Actual Result'].dropna()
grouped_bioinfo = df_data[df_data['Field'] == "Spatial Bioinformatics"]['Actual Result'].dropna()
# Creating the box plot
plt.figure(figsize=(10, 6))
plt.boxplot([grouped_omics, grouped_bioinfo], labels=['Spatiotemporal Omics', 'Spatial Bioinformatics'])
plt.title('Admission Outcomes by Field of Study')
plt.xlabel('Field of Study')
plt.ylabel('Admission Outcome')
plt.yticks([0, 0.5, 1], ['Rejected', 'Interview Only', 'Accepted'])
plt.grid(True)
plt.show()
○ 결론 : 중앙값을 보면 필자의 경우 Spatial Bioinformatics에서 더 합격률이 높았는데, 이는 실험 경험이 상대적으로 적기 때문
○ 연구 분야별 (e.g., biochem) 합격률 추이

import matplotlib.pyplot as plt
import pandas as pd
# Data mapping expected acceptance rates to actual outcomes (0: rejection, 0.5: interview only, 1: accepted) with new categories
data = {
"Stanford Bioengineering": (10, 0, "biochem"),
"Harvard Biophysics": (10, 0, "imaging"),
"Michigan Bioinformatics (PIBS)": (0, 1, "CS/BI"),
"JHU Biomedical Engineering": (5, 0, "CS/BI"),
"UCLA Biochemistry": (60, 1, "biochem"),
"UCSD Bioinformatics and Systems Biology": (30, 0.5, "imaging"),
"NYU Vilcek Institute": (45, 0, "imaging"),
"Wisconsin-Madison Biomedical Data Science": (30, 0.5, "CS/BI"),
"Wisconsin-Madison Medical Physics": (7, 1, "imaging"),
"UIUC Computer Science": (10, 0, "CS/BI"),
"Georgia Tech Bioinformatics (College of Computing)": (30, 1, "CS/BI"),
"Georgia Tech Bioinformatics (Biomedical Engineering)": (45, 1, "imaging"),
"WashU Physics": (85, 0, "physics"),
"UW Computer Science": (10, 0.5, "CS/BI"),
"UCSF Biological and Medical Informatics": (0, 0, "imaging"),
"Tri-I Computational Biology and Medicine": (35, 0, "CS/BI"),
"Gerstner Sloan Kettering Cancer Engineering": (35, 1, "imaging"),
"TSRI Computational Biology and Modeling": (60, 0, "biochem")
}
# Create a DataFrame from the data
df_data = pd.DataFrame.from_dict(data, orient='index', columns=['Expected Rate', 'Actual Result', 'Category'])
df_data.reset_index(inplace=True, drop=True)
# Group data by category for box plot
grouped_biochem = df_data[df_data['Category'] == "biochem"]['Actual Result'].dropna()
grouped_imaging = df_data[df_data['Category'] == "imaging"]['Actual Result'].dropna()
grouped_cs_bi = df_data[df_data['Category'] == "CS/BI"]['Actual Result'].dropna()
grouped_physics = df_data[df_data['Category'] == "physics"]['Actual Result'].dropna()
# Creating the box plot
plt.figure(figsize=(10, 6))
plt.boxplot([grouped_biochem, grouped_imaging, grouped_cs_bi, grouped_physics],
labels=['Biochem', 'Imaging', 'CS/BI', 'Physics'])
plt.title('Admission Outcomes by Research Category')
plt.xlabel('Research Category')
plt.ylabel('Admission Outcome')
plt.yticks([0, 0.5, 1], ['Rejected', 'Interview Only', 'Accepted'])
plt.grid(True)
plt.show()
○ 결론 : 필자는 Imaging과 CS/BI 쪽에 경쟁력을 가지고 있다.
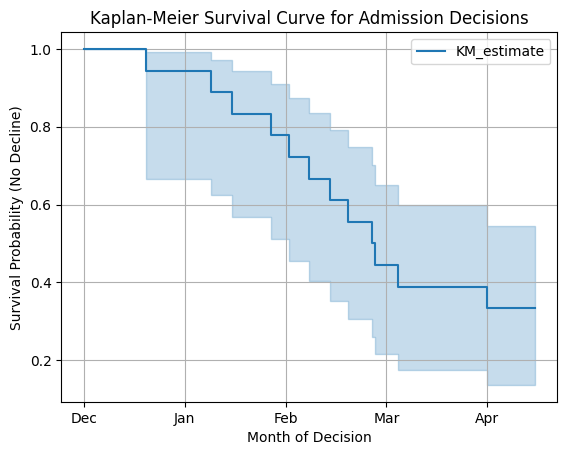
○ 시간에 따른 decline 추이를 Kaplan-Meier survival curve로 표현
from lifelines import KaplanMeierFitter
import matplotlib.pyplot as plt
import pandas as pd
import numpy as np
# Actual admission data
admission_data = {
'Program': [
'Stanford Bioengineering', 'Harvard Biophysics', 'Michigan Bioinformatics (PIBS)',
'JHU Biomedical Engineering', 'UCLA Biochemistry', 'UCSD Bioinformatics and Systems Biology',
'NYU Vilcek Institute', 'Wisconsin-Madison Biomedical Data Science', 'Wisconsin-Madison Medical Physics',
'UIUC Computer Science', 'Georgia Tech Bioinformatics (College of Computing)',
'Georgia Tech Bioinformatics (Biomedical Engineering)', 'WashU Physics', 'UW Computer Science',
'UCSF Biological and Medical Informatics', 'Tri-I Computational Biology and Medicine',
'Gerstner Sloan Kettering Cancer Engineering', 'TSRI Computational Biology and Modeling'
],
'Decision_Date': [
'2024.02.15', '2024.03.07', '2024.02.09', '2024.01.20', '2024.01.24', '2024.02.28',
'2024.01.13', '2024.02.22', '2024.02.17', '2024.03.16', '2024.03.13', '2024.03.13',
'2024.04.15', '2024.02.08', '2023.12.22', '2024.03.08', '2024.02.02', '2024.02.02'
],
'Status': [
'Declined', 'Declined', 'Accepted', 'Declined', 'Accepted', 'Declined',
'Declined', 'Declined', 'Accepted', 'Declined', 'Accepted', 'Accepted',
'Declined', 'Declined', 'Declined', 'Declined', 'Accepted', 'Declined'
]
}
for i in range(18):
if admission_data['Status'][i] == 'Accepted':
admission_data['Decision_Date'][i] = '2024.05.01' # An arbitrary large value
# Convert to DataFrame
df_admissions = pd.DataFrame(admission_data)
# Map the 'Status' to a numerical value, where 1 indicates an event (decline) occurred
df_admissions['Event_Occurred'] = df_admissions['Status'].apply(lambda x: 1 if x == 'Declined' else 0)
# Assume all applications were submitted on December 1, 2023 (this is the 'start' of our study)
application_start_date = pd.to_datetime('2023-12-01')
df_admissions['Decision_Date'] = pd.to_datetime(df_admissions['Decision_Date'])
df_admissions['Days'] = (df_admissions['Decision_Date'] - application_start_date).dt.days
# Fit the Kaplan-Meier survival estimator on the data
kmf = KaplanMeierFitter()
kmf.fit(df_admissions['Days'], event_observed=df_admissions['Event_Occurred'])
# Define a time range from the start date to the end of April 2024 for the plot
end_date = pd.to_datetime('2024-04-15')
time_range = (end_date - application_start_date).days
timeline = range(0, time_range + 1)
# Plot the survival function over the defined timeline
kmf.plot_survival_function()
# Convert numerical days back to dates for plotting on the x-axis
xticks_days = np.linspace(0, time_range, num=5)
xticks_dates = [application_start_date + pd.Timedelta(days=day) for day in xticks_days]
plt.xticks(xticks_days, [date.strftime('%b') for date in xticks_dates])
# Display the plot with labels
plt.title('Kaplan-Meier Survival Curve for Admission Decisions')
plt.xlabel('Month of Decision')
plt.ylabel('Survival Probability (No Decline)')
plt.grid(True)
plt.show()
○ 4월 15일이 4월 1일인 것처럼 보이는 등 x축 표시와 관련된 사소한 오류가 있음을 유의
○ Harvard, Stanford 등의 탑스쿨들은 상당히 늦게 decline 결과가 나오는 것으로 유명하다는 것을 유의
○ 함수 개형은 4PL regression curve와 많이 닮아 있음
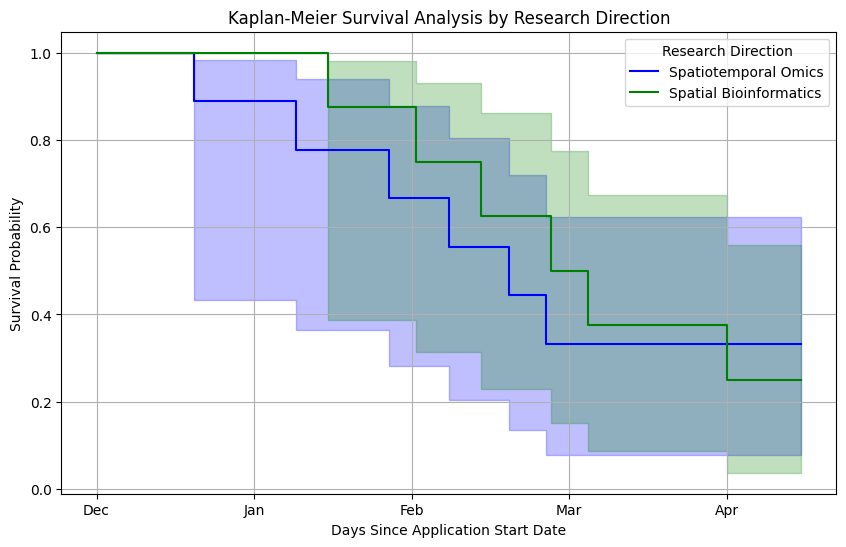
○ 연구 방향별 (spatiotemporal omics vs spatial bioinformatics) 시간에 따른 decline 추이
from lifelines import KaplanMeierFitter
import matplotlib.pyplot as plt
import pandas as pd
import numpy as np
# Actual admission data
admission_data = {
'Program': [
'Stanford Bioengineering', 'Harvard Biophysics', 'Michigan Bioinformatics (PIBS)',
'JHU Biomedical Engineering', 'UCLA Biochemistry', 'UCSD Bioinformatics and Systems Biology',
'NYU Vilcek Institute', 'Wisconsin-Madison Biomedical Data Science', 'Wisconsin-Madison Medical Physics',
'UIUC Computer Science', 'Georgia Tech Bioinformatics (College of Computing)',
'Georgia Tech Bioinformatics (Biomedical Engineering)', 'WashU Physics', 'UW Computer Science',
'UCSF Biological and Medical Informatics', 'Tri-I Computational Biology and Medicine',
'Gerstner Sloan Kettering Cancer Engineering', 'TSRI Computational Biology and Modeling'
],
'Decision_Date': [
'2024.02.15', '2024.03.07', '2024.02.09', '2024.01.20', '2024.01.24', '2024.02.28',
'2024.01.13', '2024.02.22', '2024.02.17', '2024.03.16', '2024.03.13', '2024.03.13',
'2024.04.15', '2024.02.08', '2023.12.22', '2024.03.08', '2024.02.02', '2024.02.02'
],
'Status': [
'Declined', 'Declined', 'Accepted', 'Declined', 'Accepted', 'Declined',
'Declined', 'Declined', 'Accepted', 'Declined', 'Accepted', 'Accepted',
'Declined', 'Declined', 'Declined', 'Declined', 'Accepted', 'Declined'
],
'Field': [
"Spatiotemporal Omics", "Spatiotemporal Omics", "Spatial Bioinformatics",
"Spatial Bioinformatics", "Spatiotemporal Omics", "Spatiotemporal Omics",
"Spatiotemporal Omics", "Spatial Bioinformatics", "Spatiotemporal Omics",
"Spatial Bioinformatics", "Spatial Bioinformatics", "Spatiotemporal Omic",
"Spatial Bioinformatics", "Spatial Bioinformatics", "Spatiotemporal Omics",
"Spatial Bioinformatics", "Spatiotemporal Omics", "Spatiotemporal Omics"
]
}
for i in range(18):
if admission_data['Status'][i] == 'Accepted':
admission_data['Decision_Date'][i] = '2024.05.01' # An arbitrary large value
# Create a DataFrame from the data
df_admissions = pd.DataFrame(admission_data)
# Map the 'Status' to a binary value, where 1 indicates an event (decline) occurred
df_admissions['Event_Occurred'] = df_admissions['Status'].apply(lambda x: 1 if x == 'Declined' else 0)
# Assume all applications were submitted on December 1, 2023 (this is the 'start' of our study)
application_start_date = pd.to_datetime('2023-12-01')
df_admissions['Decision_Date'] = pd.to_datetime(df_admissions['Decision_Date'])
df_admissions['Days'] = (df_admissions['Decision_Date'] - application_start_date).dt.days
# Fit the Kaplan-Meier survival estimator for each 'Field'
kmf = KaplanMeierFitter()
# Plot setup
fig, ax = plt.subplots(figsize=(10, 6))
# Colors for the plot
colors = ['blue', 'green']
# Perform the Kaplan-Meier fit and plot for each field
for i, field in enumerate(df_admissions['Field'].unique()):
if i > 1:
continue
# Filter the data for the field
field_data = df_admissions[df_admissions['Field'] == field]
# Fit the Kaplan-Meier model
kmf.fit(field_data['Days'], event_observed=field_data['Event_Occurred'], label=field)
# Define a time range from the start date to the end of April 2024 for the plot
end_date = pd.to_datetime('2024-04-15')
time_range = (end_date - application_start_date).days
timeline = range(0, time_range + 1)
# Plot the survival curve
kmf.plot_survival_function(ax=ax, color=colors[i])
# Convert numerical days back to dates for plotting on the x-axis
xticks_days = np.linspace(0, time_range, num=5)
xticks_dates = [application_start_date + pd.Timedelta(days=day) for day in xticks_days]
plt.xticks(xticks_days, [date.strftime('%b') for date in xticks_dates])
plt.title('Kaplan-Meier Survival Analysis by Research Direction')
plt.xlabel('Days Since Application Start Date')
plt.ylabel('Survival Probability')
plt.legend(title='Research Direction')
plt.grid(True)
plt.show
○ 결론 : 실험과 관련된 spatiotemporal omics 쪽이 좀 더 decline이 빠른 경향
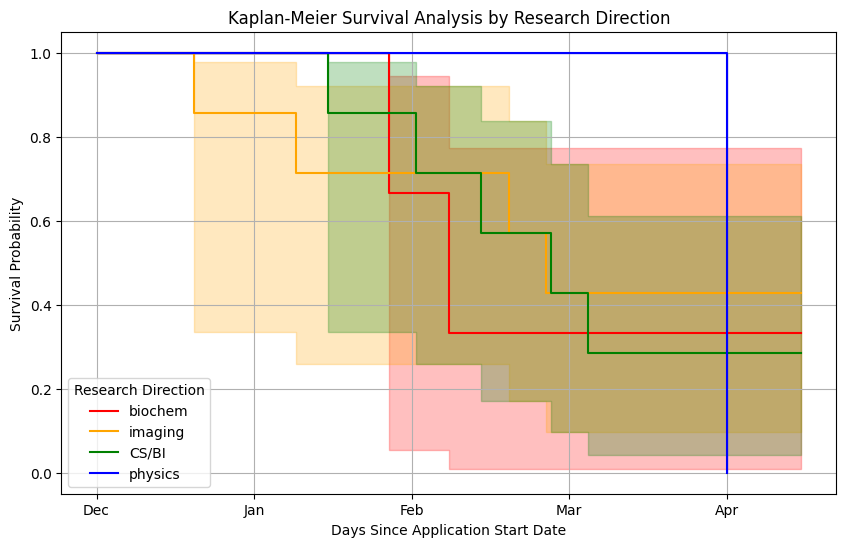
○ 연구 분야별 (e.g., biochem) 시간에 따른 decline 추이
from lifelines import KaplanMeierFitter
import matplotlib.pyplot as plt
import pandas as pd
import numpy as np
# Actual admission data
admission_data = {
'Program': [
'Stanford Bioengineering', 'Harvard Biophysics', 'Michigan Bioinformatics (PIBS)',
'JHU Biomedical Engineering', 'UCLA Biochemistry', 'UCSD Bioinformatics and Systems Biology',
'NYU Vilcek Institute', 'Wisconsin-Madison Biomedical Data Science', 'Wisconsin-Madison Medical Physics',
'UIUC Computer Science', 'Georgia Tech Bioinformatics (College of Computing)',
'Georgia Tech Bioinformatics (Biomedical Engineering)', 'WashU Physics', 'UW Computer Science',
'UCSF Biological and Medical Informatics', 'Tri-I Computational Biology and Medicine',
'Gerstner Sloan Kettering Cancer Engineering', 'TSRI Computational Biology and Modeling'
],
'Decision_Date': [
'2024.02.15', '2024.03.07', '2024.02.09', '2024.01.20', '2024.01.24', '2024.02.28',
'2024.01.13', '2024.02.22', '2024.02.17', '2024.03.16', '2024.03.13', '2024.03.13',
'2024.04.15', '2024.02.08', '2023.12.22', '2024.03.08', '2024.02.02', '2024.02.02'
],
'Status': [
'Declined', 'Declined', 'Accepted', 'Declined', 'Accepted', 'Declined',
'Declined', 'Declined', 'Accepted', 'Declined', 'Accepted', 'Accepted',
'Declined', 'Declined', 'Declined', 'Declined', 'Accepted', 'Declined'
],
'Category': [
"biochem", "imaging", "CS/BI",
"CS/BI", "biochem", "imaging",
"imaging", "CS/BI", "imaging",
"CS/BI", "CS/BI", "imaging",
"physics", "CS/BI", "imaging",
"CS/BI", "imaging", "biochem"
]
}
for i in range(18):
if admission_data['Status'][i] == 'Accepted':
admission_data['Decision_Date'][i] = '2024.05.01' # An arbitrary large value
# Create a DataFrame from the data
df_admissions = pd.DataFrame(admission_data)
# Map the 'Status' to a binary value, where 1 indicates an event (decline) occurred
df_admissions['Event_Occurred'] = df_admissions['Status'].apply(lambda x: 1 if x == 'Declined' else 0)
# Assume all applications were submitted on December 1, 2023 (this is the 'start' of our study)
application_start_date = pd.to_datetime('2023-12-01')
df_admissions['Decision_Date'] = pd.to_datetime(df_admissions['Decision_Date'])
df_admissions['Days'] = (df_admissions['Decision_Date'] - application_start_date).dt.days
# Fit the Kaplan-Meier survival estimator for each 'Category'
kmf = KaplanMeierFitter()
# Plot setup
fig, ax = plt.subplots(figsize=(10, 6))
# Colors for the plot
colors = ['red', 'orange', 'green', 'blue']
# Perform the Kaplan-Meier fit and plot for each category
for i, category in enumerate(df_admissions['Category'].unique()):
if i > 3:
continue
# Filter the data for the category
category_data = df_admissions[df_admissions['Category'] == category]
# Fit the Kaplan-Meier model
kmf.fit(category_data['Days'], event_observed=category_data['Event_Occurred'], label=category)
# Define a time range from the start date to the end of April 2024 for the plot
end_date = pd.to_datetime('2024-04-15')
time_range = (end_date - application_start_date).days
timeline = range(0, time_range + 1)
# Plot the survival curve
kmf.plot_survival_function(ax=ax, color=colors[i])
# Convert numerical days back to dates for plotting on the x-axis
xticks_days = np.linspace(0, time_range, num=5)
xticks_dates = [application_start_date + pd.Timedelta(days=day) for day in xticks_days]
plt.xticks(xticks_days, [date.strftime('%b') for date in xticks_dates])
plt.title('Kaplan-Meier Survival Analysis by Research Direction')
plt.xlabel('Days Since Application Start Date')
plt.ylabel('Survival Probability')
plt.legend(title='Research Direction')
plt.grid(True)
plt.show
○ 결론 : imaging 분야가 좀 더 decline이 빠른 경향
2. 유학 합격 수기 [목차]
○ 결국 big question과 관련된 내용을 SOP에 담아야 한다.
○ 마치 대담한 사업가가 사람들의 마음을 흥분시키는 것처럼, SOP에 담긴 미래 과학 비전이 교수들의 마음을 움직이는 형태가 바람직하다.
○ 이에 반해, 벌써 40년도 더 전의 기술인 웨스턴 블롯팅을 아무리 잘한다고 강조한들 역효과만 날 뿐이다.
○ 따라서 말 잘듣는 전형적인 아시안임을 강조하는 것보다 다소 적극적으로 내가 반드시 이 구상을 완성하겠다는 의지를 피력함이 중요하다.
○ 다만, too bold 하면 떨어트릴 수 있으므로 적절한 스탠스를 유지하는 게 중요하다. (ref)
○ 가장 안전한 것은 역대 올해의 기술 등을 참고하여 많은 교수님들이 공감하는 미래 트렌드를 선택하면 좋다.
○ 필자의 경우, 시공간 오믹스를 구현하여 red, white, green bio 모두에 시공간 오믹스가 표준기술로 쓰이는 미래를 구상하고 있다.
○ 각 프로그램별 인상
○ UCLA BMSB : 생화학 + 생물정보학과 같이 dry-lab 중에서도 soft knowledge를 많이 요구하는 학문이 잘 발달한 느낌이다. 아마 UCB, UCLA, UCSD, UCSF 등 UC들이 위치한 캘리포니아에 많은 글로벌 제약회사가 있는 것과 관련하여 chemoinformatics, biochem 계열 학문이 강세를 보이는 듯 하다. 하지만, stipend가 검색해도 잘 안 나오는 건, 요즘 UC 계열이 돈이 적다는 것과 2023-2024 입시에서 UC 내 학생 시위가 있었던 것과 관련 있는 듯.
○ UMich Bioinformatics : 보통 졸업 기간이 3 ~ 5년 정도인데 석사 학위 소지자는 3 ~ 4년 정도라는 듯 하다. 어떤 PI의 랩은 웬만해서 3년만에 졸업시켜 주기도 한다는 듯. 필자가 여길 진학한다면 3.5년을 목표로 할 것 같다. 정보이론 등 dry-lab 중에서도 hard knowledge를 많이 요구하는 학문이 잘 발달한 느낌이다. 다만, wet-lab도 자체 개설하여 데이터 생산 독립성을 높임으로써 BI 학과의 생존 가능성을 높이려는 시도가 돋보인다. tuition fee full funding + ~40k USD/yr. PhD 과정 내에는 international이 거의 없다는 걸 알고 지원했지만 합격을 한 점이 의아하다.
○ GT Bioinformatics : 졸업자 진로를 보면 GT는 UMich보다도 FAANG 취업이 더 잘 되는 것처럼 보인다 (CS뿐만 아니라 Bioinformatics 프로그램도 해당하는 설명). stipend를 적게 줄 수도 있다는 말을 들은 적은 있었는데, 필자의 경우 tuition fee full funding + ~40k USD/yr 정도 된다는 모양. CS와 BME에서 bioinformatics를 둘 다 지원했는데 어차피 학위는 bioinformatics로만 뜨기 때문에 중복 지원이라고 했다. BME의 경우 지리적으로 인접한 Emory University와 공동으로 프로그램이 운영되는 것 같다. international은 많은 편
○ Sloan Kettering Institute Cancer Engineering : 슬론 연구소, 그와 연결된 록펠러 대학, 웨일 코넬을 비지팅 하였을 때, bioengineering 학문의 최전선을 볼 수 있었다. 예를 들어, 전사인자(TF)들에 서로 다른 형광을 붙인 뒤 빛의 간섭과 FRET을 이용하여 TF 간 거리를 시공간적으로 보는 연구가 있었다. 이렇듯 search space가 하나의 우주를 이룰 법한 분야가 최근의 트렌드인 것 같고, 그렇게 해야 BME 계열에 있어 미국의 PI가 되는 것으로 보인다. computational track도 물론 많지만, wet-lab 80% + dry-lab 20% 정도 되는 느낌. 뉴욕시 물가를 고려해서, Columbia University처럼 tuition fee full funding + ~50k USD/yr 정도의 stipend를 갖는다.
○ UW-Madison Medical Physics : PET, MRI, US 등 이미징/핵의학 중심의 프로그램. direct admit을 해야만 입학을 할 수 있고, 이미 합이 맞는 PI를 찾았다면 PI를 지원자가 찾아다녀야 하는 인터뷰 절차는 굉장히 쉽게 풀릴 수 있다. 비록 필자는 이곳에 진학하지 않았지만, 컨택된 PI들이 지원자에 맞는 다른 PI들을 열심히 찾아주는 것 같아 인터뷰 절차는 합격이 보장된 것처럼 보인다.
○ '계기'라는 것은 '실패'라는 모습으로 잔인하게 찾아온다. 뜻하지 않게 만난 실패 속에 난 무엇을 얻고 무엇을 알게 되었는가?
○ 지금부터 한 발짝. 이 세상이 재미있어지기 시작했다. 앞으로 하고 싶은 게 잔뜩 있다는 것을 알게 됐다.
○ 존재론적 각성. 무인도에 갇혀도 종이와 펜만 있다면 행복할 것 같다는 깨닳음. 그러므로 사람으로부터 마음의 평화를 찾지 않겠다는 선언
○ 나는 상상을 초월할 정도로 치열한 삶을 살고 싶다. 이 세상을 움직이는 거인들의 시선에 닿기 위해 노력하고 싶다. 주 120시간씩 한 번도 내 머리는 쉬지 않겠지. 그 과정은 나에게 얼마나 가혹하고 고독할까. 그럴수록 나는 더욱 끓어오를 것이다. 트럼프의 한 구절이 떠오르는 요즘이다.
I do it for fun, I do it for game. - Donald J. Trump
○ 내가 미시간을 고른 이유
○ AACR 2024를 참가하면서 느낀 점. Aviv Regev, Christina Curtis, Dana Pe'er, Sanja Vickovic, Joakim Lundeberg와 같은 교수님들을 만날 수 있었다. 이들의 강연에서 공간생물학은 향후 모든 제약업계를 휩쓸 파급력이 있다는 것을 알았고, 내가 생각하는 공간생물학의 다음 단계를 가장 잘 연구할 수 있는 미시간을 골랐다.
3. 앞으로의 계획 [목차]
○ 방향에 대하여
○ 유학입시 당시 UCLA를 생각했으나 UMich을 진학한 것처럼 초기에 설정한 방향에 따라 인생은 흘러가지 않는다. 하긴 그것은 인간관계도 마찬가지. 마치 첫사랑이 이뤄지지 않는 것처럼. 하지만 그건 이미 취리히 입시를 통해 깨닳은 것.
○ 다만, '방향'이라는 것은 모멘텀(momentum)을 주고, 실제로 흘러갈 인생의 방향은 초기에 설정한 방향과 얼추 비슷하게는 간다.
○ 내가 다른 사람들을 이롭게 하는 이유
○ 한때 꿈이 많았으나 현실의 어려움에 부딪혀 충분히 자신의 잠재력을 발휘하지 못했던 사람이 있었다. 그렇지만 그 사람은 성공을 위한 갈증이 있었고, 그래서 나는 그 사람을 유학의 길로 이끌었다. 결국 그 사람은 석사로 입학했고, 풀펀딩을 받았으며, 1년만에 박사로 트랜스퍼를 했다. 대학원 재학 중에 아마존에서 인턴 소프트웨어 엔지니어로 일할 예정이라는 소식도 전했다.
○ 인간의 성장은 위대하다. 때로는 인과율을 바꿀 정도로.
○ 꿈을 잃어버린 사람에게 내일이 기대되는 삶을 선물해 주었다는 것. 그냥 그것이 보기 좋아서, 희망을 잃어버린 자들에게 희망을 얘기하려고 한다.
○ 인더스트리
○ 인더스트리에 대하여
○ US job market에서 벌어들일 수 있는 평생 기대 소득이 다른 진로에 비해 몇배 이상 많음
○ 어떤 길을 가더라도 빅테크 인더스트리를 경험하는 게 전략적이라고 판단
○ 최근 공간생물학이 각광받기 시작한 건 결국 이 분야가 high-throughput으로 scalable해지고 자동화됐기 때문이다. 이제 학문이란 인더스트리와 불가분의 관계가 되었다. Roche에서 시공간 오믹스가 도입되기 시작되고, Novartis가 RLT 약제에 집중하는 까닭도 신약 개발 과정의 자동화를 위한 시도로 읽힌다.
○ 방향 1. 엘론 머스크가 창업한 xAI 라는 회사에 가고 싶다는 생각이 생겼다.
○ 이유 : 지속 가능한 AI 산업에 대한 고민. AI의 실존적 위협은 분명 현실 앞으로 다가왔다.
○ 평가 : 론칭한지 불과 반년 정도밖에 안되는 신생 스타트업으로 현재 직원 8 ~ 10명 수준이다. 4년 정도 후에 수십 ~ 수백 명 정도의 회사가 될 것이다. 'Understand the Universe'라는 구호를 회사 모토로 하는데, 나와 비슷한 사람들을 만날 것 같은 기대감이 든다.
○ 예측 : AI 빅테크는 구글 딥마인드, OpenAI, X.ai 등 3개 회사의 삼파전이 될 것으로 예측한다. 그러나 구글 딥마인드와 OpenAI는 AI와 인류의 공존에 대해 적절한 답을 제시하고 있지 못하고 있다. (ref)
“이게 오픈AI 원래 설립 의도” 창립 멤버 머스크가 작심비판하며 올린 사진
이게 오픈AI 원래 설립 의도 창립 멤버 머스크가 작심비판하며 올린 사진 AI권력 쓸어모으는 오픈AI에 불만 표출
www.chosun.com
○ 방향 2. 화이자, 모더나 등 글로벌 제약회사 : bioinformatician의 수요가 많음
○ 학계(academia)
○ 학계에 대하여
○ 아카데미의 대가이면서 막대한 사회적 영향력을 가진 사람이 되는 방향도 충분히 값진 일일 것 같다.
○ Visium을 개발한 SciLifeLab 출신 연구자들이 스탠포드, 컬럼비아대 등의 교수가 되는 것을 보면, 시대를 바꿀 수 있는 굵직한 기술을 개발해야 탑스쿨의 교수가 될 수 있음을 느낀다.
○ 방향 1. 대학교형 포닥 : 하버드, 스탠포드 등 대가를 찾아가는 방향
○ 방향 2. 연구소형 포닥 : Allen Institute, Broad Institute, Sanger Institute, Francis Crick Institute 등
○ 방향 3. 기업형 포닥 : IT 계열은 아마존이 있음; 바이오는 Novartis Innovation Postdoctoral Fellowship Program과 같은 제약회사가 있음
○ 채용/포닥/교수 공고 예시
○ Pfizer
○ Stellenangebote de ETH Zürich
○ KTH Royal Institute of Technology
○ 사업(business)
○ 나는 결국 수만 개의 high-paying jobs를 만드는 사람이 돼야 할 지도 모른다. 반도체 산업에만 의존하는 것은 위험하다. 더욱이 대한민국에서 그것을 할 수 있는 사람은 많지 않다.
○ AI로 인한 산업의 지각변동은 생각한 것보다 너무 빠르다. 그러면서 AI가 쉬이 적용될 수 없는, 많은 일자리가 창출될 초격차 고맥락 규모의 산업을 일으키고 싶다.
○ 정치(politics)
○ 이건 정치인이 되고 싶다는 생각보다는 정치가 바꾸었으면 하는 세상의 모순들을 적는 공간
○ 저출산 : 분기 출산률 0.6이라는, 극단적인 전쟁 상황에서나 볼 법한 숫자가 나왔다. 이것은 내가 본 문제 중 가장 어려운 수학 문제. 이를 해결하기 위해 다음과 같은 솔루션을 구상·계획하고 있다.
○ AI·로봇을 보급하여 노동 수요를 크게 줄인다.
○ 핵심 산업을 유치하여 양질의 일자리(high-paying jobs)를 늘린다.
○ 모든 고등지식을 공개하여 평생 교육 비용을 줄이고 개인의 사회 진출을 촉진한다. 특히, 수도권·고학력 여성이 결혼·출산을 꺼리게 되는데 이는 사회진출 지연 및 경력단절과 관련 있어 보인다.
○ 연애 결혼을 억제하고 중매 결혼을 대중화 한다. 사회 진출 시기가 늦어져 결혼적령기가 늦어진다면 연애를 하지 않으면 된다.
○ 그 밖의 이민정책, 육아정책, 여성정책 등은 학자가 아닌 행정가의 영역일 것이다.
○ 혁신산업 : OpenAI의 ChatGPT의 등장은 앞으로 웬만한 산업은 다 자동화할 수 있음을 암시한다. 앞으로 AI에 의해 대체될 수 없는 산업은 고맥락 산업이면서 규모의 산업이어야 한다. 이 규모의 산업은 수만 개의 high-paying jobs를 창출할 수 있으며, 고임금 노동자와 저임금 노동자가 공존하는 이상적인 산업이다. 다음과 같이 진단한다. 참고로, 완전 자동화가 완성되면 IT 산업은 독립적인 산업이 되기는 힘들다.
○ 바이오 : 주로 신약 개발. 식품, 화장품보다 앞도적으로 시장이 큰 게 신약이다. 초코파이 회사 오리온도 이제는 신약을 만든다! (ref)
○ 양자 : 양자컴퓨팅, 양자통신, 핵융합 등등
○ 우주 : SpaceX 등등
○ 환경 : 유일하게 산업화되지 않은 잠재 혁신산업. 내가 하고자 하는 건 바이오, IT, 환경을 하나로 묶는 것 (ref)
○ 그 밖에도,
○ OECD 자살률 1위
○ 정치적 양극화 : 미국도 양극화는 심하기 때문에 우리나라만의 문제는 아니다. 스위스를 참고할 법 한데, 스위스는 히말라야 산맥에 둘러싸여 외세의 침입이 없었기 때문에 정치적 안정을 이룬 것 아닌가?
입력: 2023.11.26 11:01
수정: 2024.02.18 21:31
'▶ 인문과학 > ▷ 유학' 카테고리의 다른 글
| 【해외 지원 후 장학금】 아산의생명장학금 면접 대비 (12) | 2024.12.27 |
|---|---|
| 【유학 입시】 유학 이후의 목표 (0) | 2024.04.30 |
| 【유학 입시】 유학 인터뷰 예상 질문 및 답변 ver.2 (3) | 2023.12.30 |
| 【유학 입시】 2024년 9월 입학 기준, 미국 대학원 석∙박사과정 (16) | 2023.09.27 |
| 【유학 입시】 유학 입시 실패, 그리고 새로운 도전 (10) | 2023.09.26 |



최근댓글